中国科学院北京基因组研究所生命与健康大数据中心
(BIG Data Center, BIGD) 于2016年2月29日正式成立,是研究所三大科研体系之一。中心面向我国人口健康和社会可持续发展的重大战略需求,围绕国家精准医学和重要战略生物资源的组学数据,建立海量生物组学大数据汇交、存储与管理的应用与共享平台,发展组学大数据系统整合、挖掘与分析的新技术、新方法,力争建成支撑我国生命科学发展、国际知名的生命与健康大数据中心。
来!看一下主页画风(http://bigd.big.ac.cn/):
确实,跟NCBI有相似之处!不过,国人自己的数据库更好用!界面更清晰!服务更贴心!中国人还是得支持一下自己的事业!近两年已经有数十篇SCI高水平论文使用了GSA数据库,其中不乏有Nature、Cell杂志期刊,论文题目列表附后。
那么,先来了解一下咱们的组学原始数据存储归档系统Genome SequenceArchive(GSA)吧!
GSA的系统建设遵循了国际核酸序列共享联盟(InternationalNucleotide Sequence Database Collaboration,INSDC)的相关标准,并作为INSDC的补充,旨在减轻国际相关数据库数据存贮及数据传输的压力;立足中国,服务全球。
由于中国国际网络出口带宽的瓶颈问题,数据传输效率低下。以中国科学院北京基因组研究所的150Mbs出口带宽为例,向NCBI数据库递交1TB的数据需要花费2周以上的时间。GSA的建立可以让国人享受更快的传输速度,更贴心的服务!
如果受够了数据传输的龟速,来找我们吧!
如果受够了语言的困扰,来找我们吧!
如果受够了服务的滞缓,来找我们吧!
太懒只看重点版:
GSA数据库结构数据分为四类,即项目信息(BioProject)、样本信息(BioSample)、实验信息(Experiment)和测序信息(Run)。
截止到2017年9月中旬,共有来自全球的97个研究机构的178个课题组 递交数据,涉及380多个科研项目,涵盖超过14000个生物学样本及其组学数据。GSA相继获得PNAS、AJHG、Cell Research等多个国际期刊认可。
GSA数据库支持多种数据类型和格式、传输速度快。
数据量太大,有问题怎么办?
直接通过 Email: [email protected] 或者加 QQ 群: 548170081 联系,数据通过邮寄硬盘也可以哦!
直接学习数据如何传GSA流程:
第一步,准备工作
准备好你的reads文件,计算好他们的md5。
比如:
5aa05dd66815b027b10233c8661e07bb T112_good_1.fq.gz
1c9bd20eae0848fdcaab37cbb3f88cde T112_good_2.fq.gz
前面的这一串数字就是md5值。
这个md5呢是对数据完整性的一种校验,百度下载一个md5计算器,就可以进行计算。当然公司测序之后会连带md5一起发给你,这就方便多了。
第二步,建立bioproject
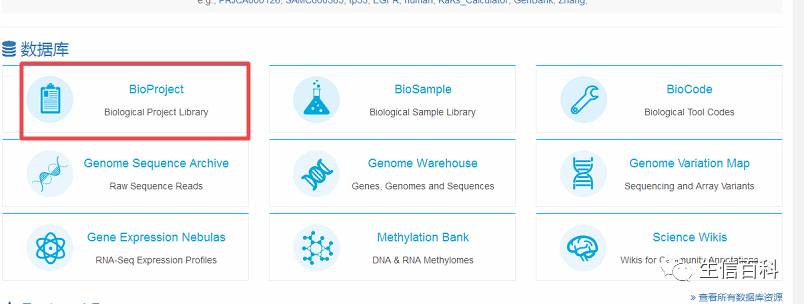
点击红框位置,进入之后页面如下:
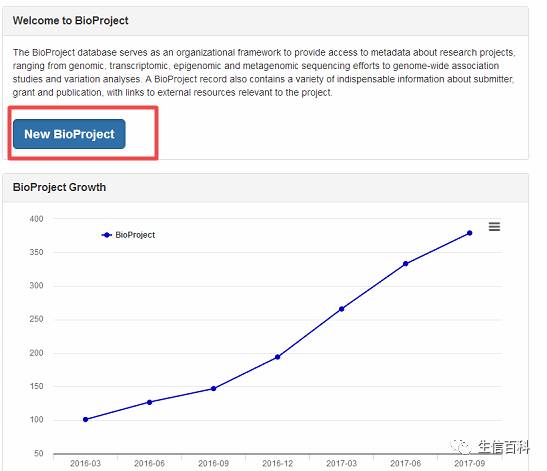
点击New Bioproject,进入页面如下:
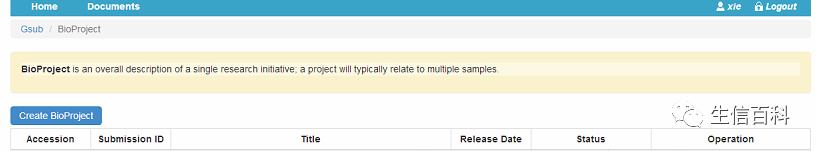
继续点击 Creat Bioproject,进入页面如下:

填好信息,星号位置必填。点击红色按钮,进行下一步,进入页面如下:
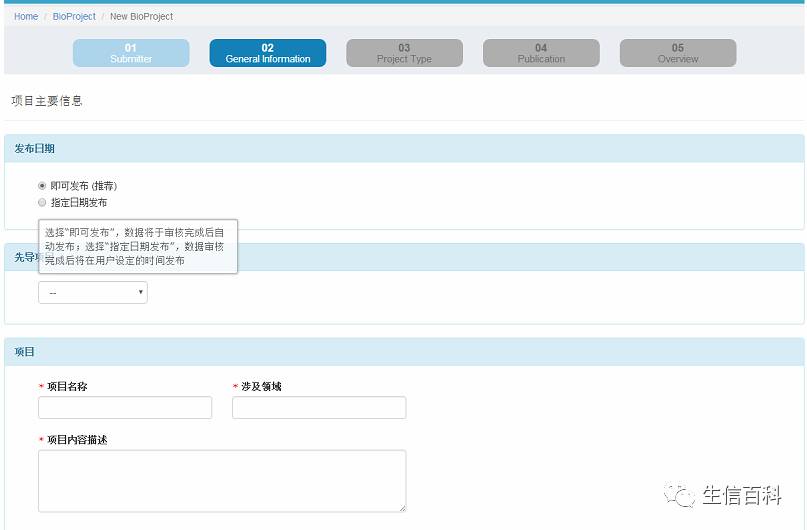
自己建立项目名称,选择是否立即释放,点击红色按钮,进行下一步,进入页面如下:
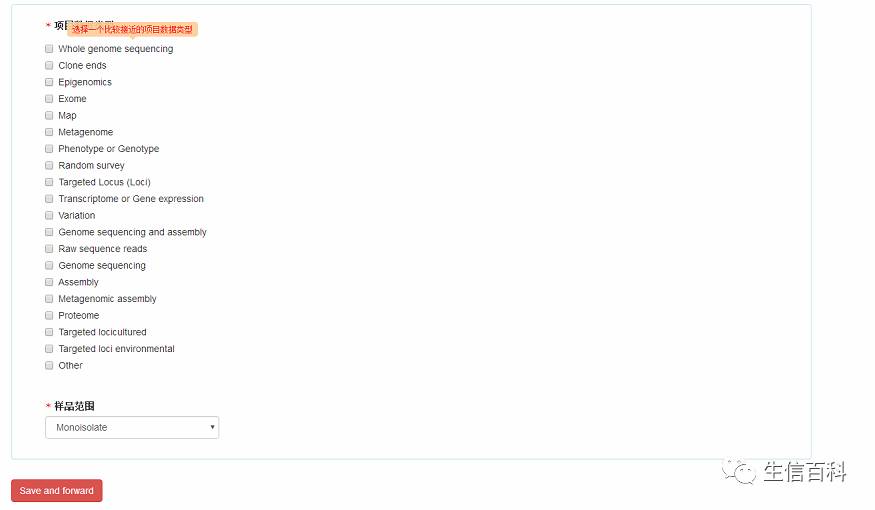
选择什么类型数据,基因组、转录组、还是甲基化数据?纯品还是混样?
点击红色按钮,进行下一步,进入页面如下:
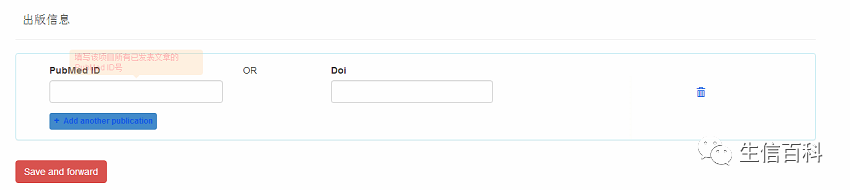
此步骤可填可不填,点击红色按钮,进行下一步,进入页面如下:
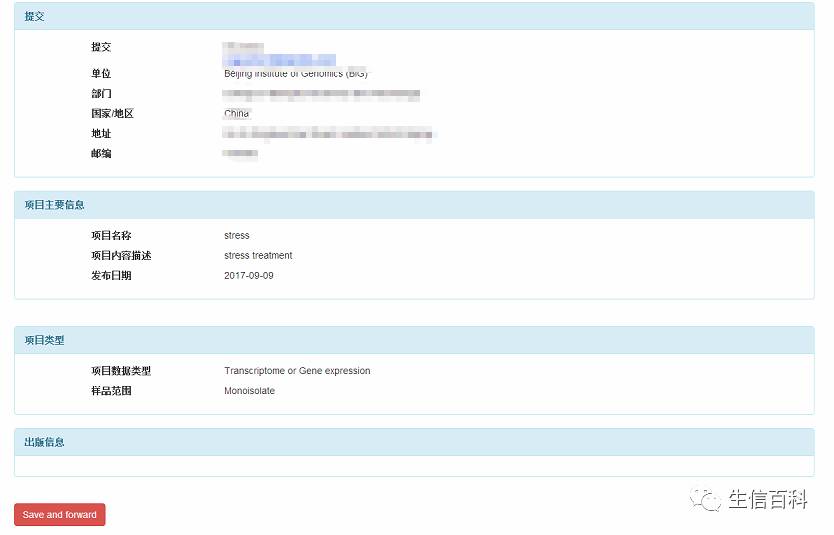
最后一步是Overview阶段,提交确认,这样,Bioproject就建立了。
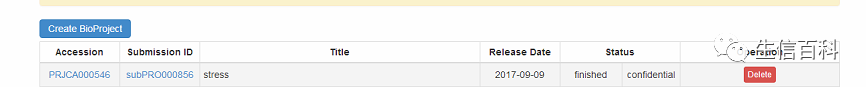
返回可以看到,Bioproject已经成功建立。
第三步,构建 Biosample
先讲一下Biosample什么概念,比如我们在四个条件下测的转录组,包括:高盐胁迫、干旱胁迫、低温胁迫、重金属胁迫。这样的四个不同的实验称为四个Biosample。
这样的话,我们需要建立四个Biosample,假如每一个处理下又存在两个重复,这样的话,一个处理需要建立两个Biosample.
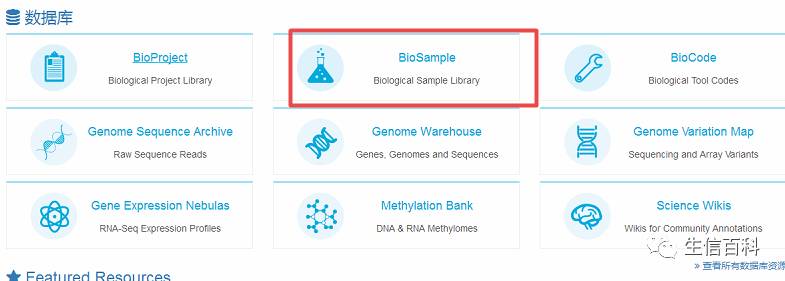
点击红框的Biosample,下一步,进入页面如下:
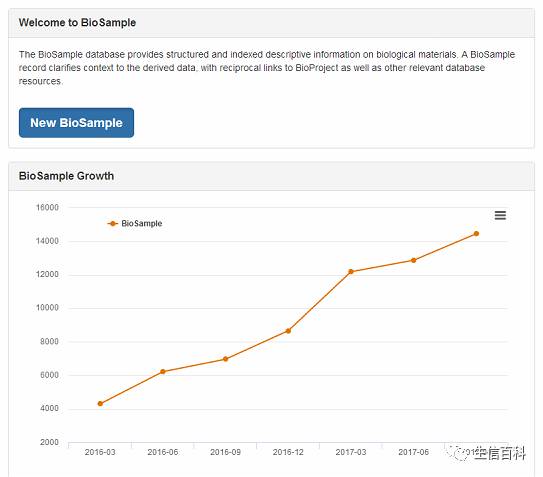
点击New Biosample,下一步,进入页面如下:
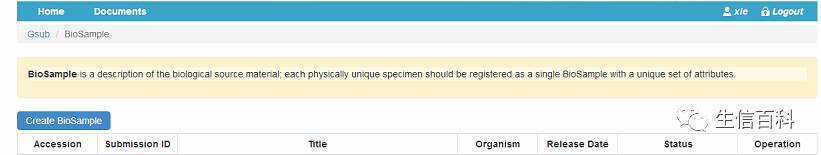
点击Creat Biosample,下一步,进入页面如下:
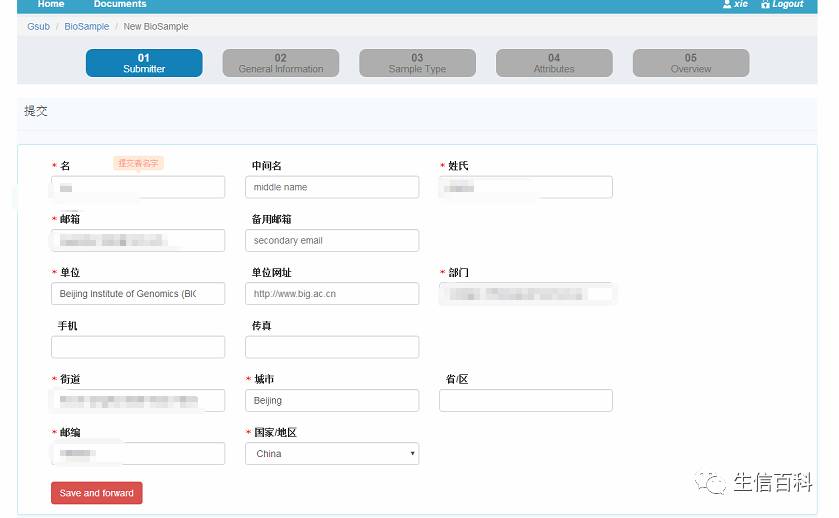
同样的,点击红色按钮,下一步,进入页面如下:
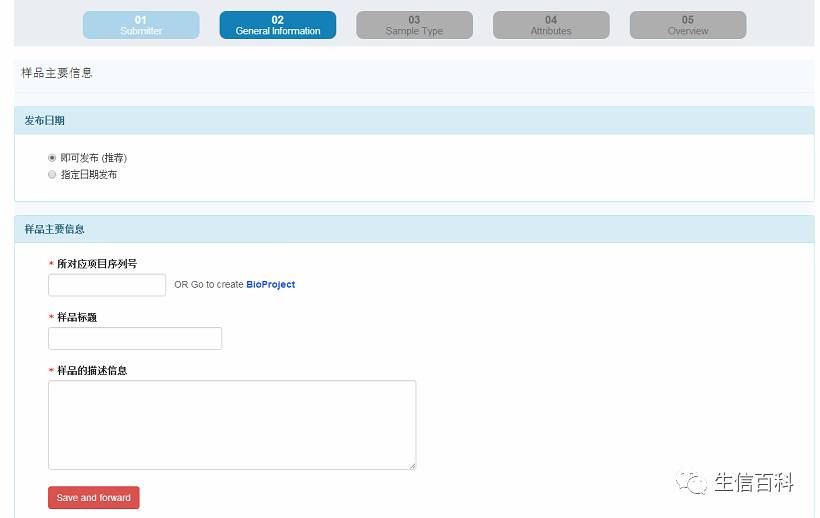
此步骤注意了,要关联bioproject的信息,将上一步构建好的bioproject号码填写进去。点击的时候,会自动进行关联,直接选择就好了。
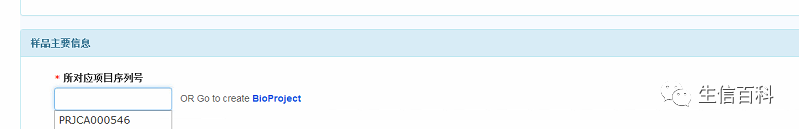
点击红色按钮,下一步,进入页面如下:
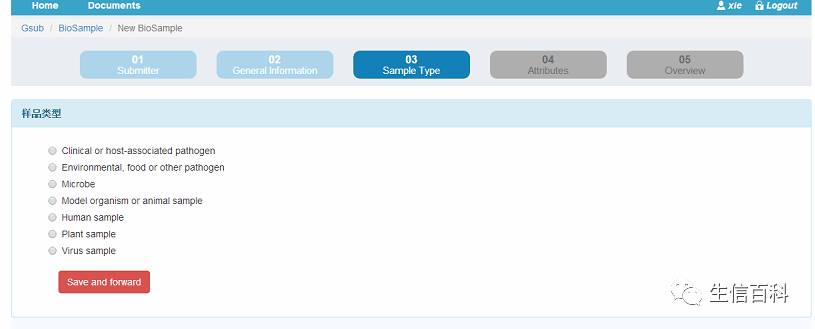
选择,进行下一步,进入页面如下:
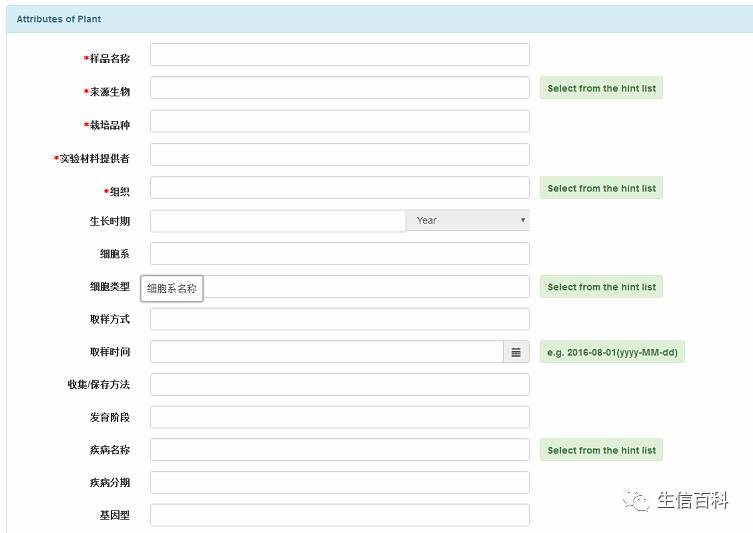
这一步信息填的稍多一些,耐心填完,星号为必填。

最终还是,overview。
没问题,提交!
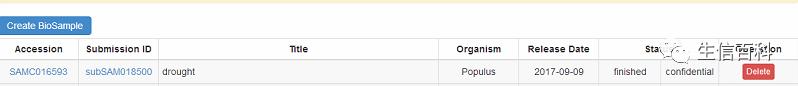
返回,会看到,biosample里面已经建立了序列号。
同样的,为了存储四个条件下的数据,我们建立四个biosample。这样的话,在biosample菜单下会存在四个序列号。如果每个处理有两个重复的话,会有八个序列号。
第四步,Reads提交
点击红色框,进入下一步
点击红框上传,进入页面如下:
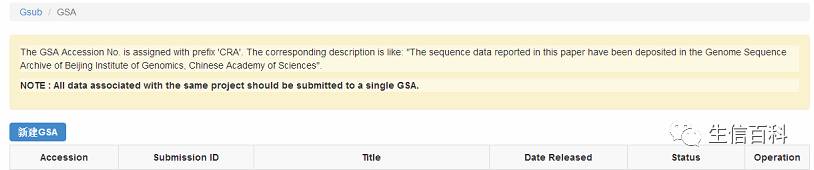
新建GSA,下一步,进入页面如下:
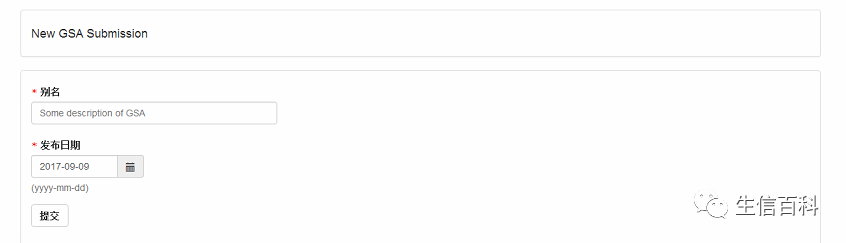
填写信息,自己认识的一个别名,提交,下一步,进入页面如下:
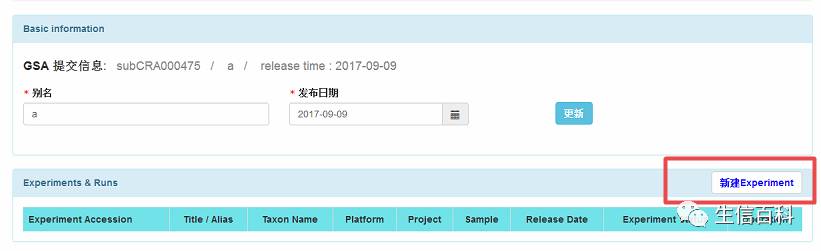
这一步注意了,需要构建Experiment。点击,进行下一步,进入页面如下:
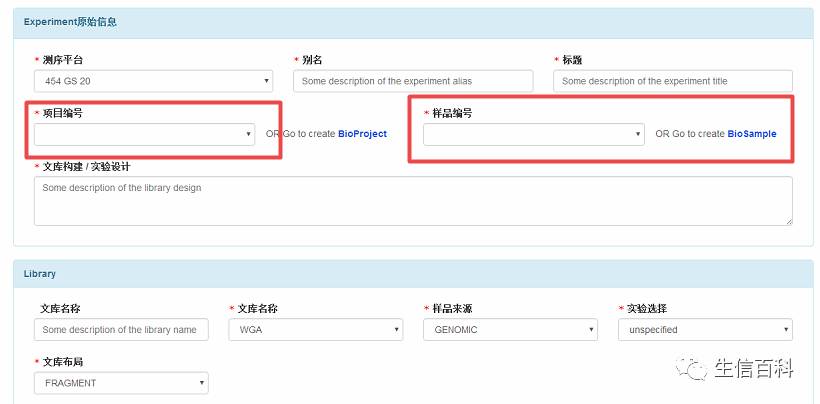
假如这四个处理都是在一个bioproject下进行的,那么其余三次选择左边红色方框的内容时候,都需要选择同一个bioproject。然而,四个实验属于同一个bioproject不属于同一个biosample,所以之前我们要在同一个bioproject下构建四个biosample,传数据时会对每个sample进行关联。
注意:不同重复需要在同一个Bioproject下建立多个biosample。
提交之后,Experiment就构建好了,确认后返回,页面如下:
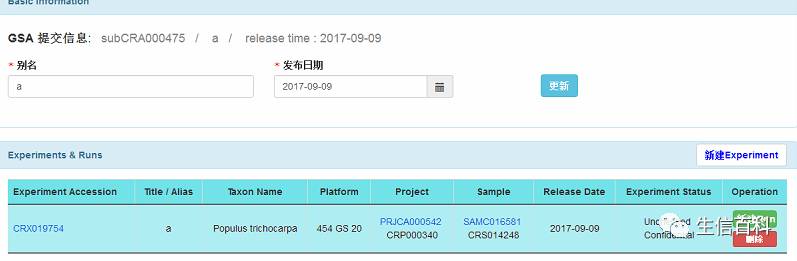
下一步就是构建run,按照操作步骤往下走就ok了。
构建run结束了,也就是传数据阶段。
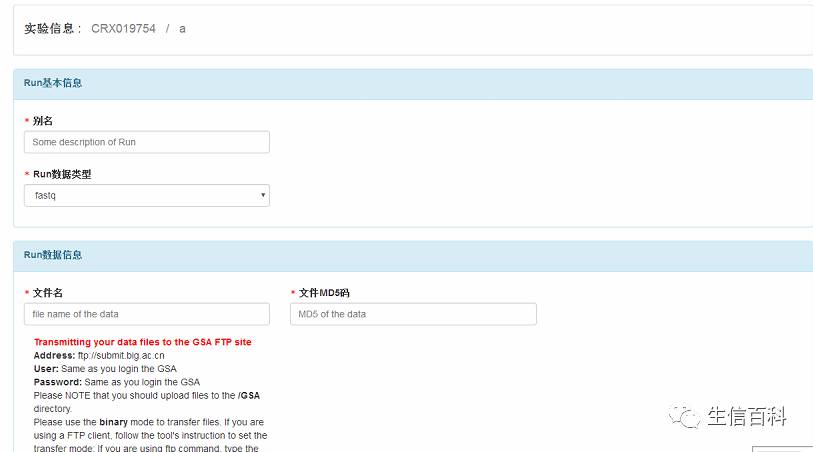
填好,刚才我们例子中提供的文件全名和md5信息。记住,一定要全名,包括后缀gz。
根据下面提供的ftp地址,上传就好了(可以使用FileZilla软件),一定要传到GSA文件夹里面,传完之后,就是等待机器审核了。
很快,系统会邮件通知你的序列号。
真粉必看长文版:
数据结构与模型
为了确保与INSDC数据库系统的兼容性,GSA遵循了INSDC数据库系统的数据标准和数据结构,并将数据分为四类,即项目信息(BioProject)、样本信息(BioSample)、实验信息(Experiment)和测序信息(Run);数据结构如下图1所示。
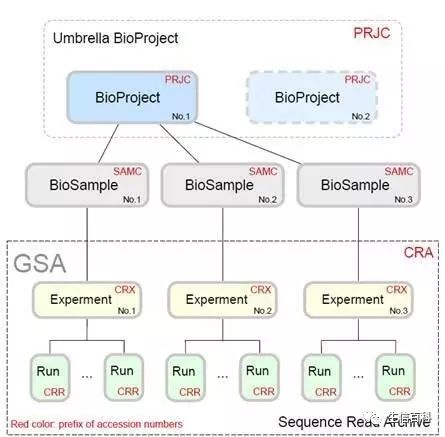
图1 GSA 数据模型
项目信息的数据获取号(Accession Number)以“PRJCA”为前缀,其中字母“C”表示中国。项目信息提供了一个针对本研究任务的概要性描述,并包括研究目的、涉及的物种、数据类型、数据递交者、基金资助机构、发表的文章等信息。样本信息的数据获取号以“SAMC”为前缀,包含一些有关生物样本的描述信息如样本类型、样本属性等。实验信息以“CRX”为前缀,为特定样本实验处理方式,包括实验目的、文库构建方式、测序类型等信息。测序信息的数据获取号以“CRR”为前缀,内容主要包括测序文件和对应的校验信息。在四类数据中,项目信息和样本信息是独立运行的模块,而实验信息和测序信息形成了测序序列的归档库。基于上述标准和结构,GSA不仅方便数据递交,而且便于管理数据权限,实现数据共享与交换。
除此之外,GSA考虑大型项目管理的需求,引入Umbrella Project概念,提供大型合作型项目的伞装结构管理。目前,已有两个中国科学院战略先导项目和一个中国科学院重点研究项目正在使用GSA系统管理和共享项目数据。
数据归档与统计
GSA接收来自全球的数据递交,接收不同测序平台产出的组学数据,并支持通用的数据文件格式如FASTQ、BAM、VCF。同时,GSA对接收到的数据进行质量评估,确保数据的完整性和可用性。在数据安全方面,类似于INSDC数据库系统,GSA允许数据递交者设置其数据的访问权限(公开访问或受控限制);公开即意味着数据可被任何人访问或下载使用,受控即其他人的访问在一段时间内将被受到限制。在GSA系统后台,可被公开访问和受控访问的数据存储于不同的磁盘空间内,以确保数据的安全性。从2015年8月份GSA系统上线至今,系统中的数据呈现显著增长的趋势(图2),截止到2017年9月,GSA已经接收了来自97个研究机构391余位科研人员的用户注册信息,并收录379个项目,14444个样本,19465个实验和21351个测序信息,涵盖了超过120个物种的信息。

图2 GSA数据统计
数据递交与信息检索
GSA系统提供用户注册和登录功能,因此在创建一个数据递交前,首先需要通过GSA系统注册用户账户,在用户账号被验证通过并激活后,方可登录系统并创建数据递交页面。通常情况下,在GSA中完成一个数据递交需要执行五个操作,分别为注册项目、样本、实验、测序四类元数据信息和提交序列文件(图3)。在元数据信息收集页面,GSA系统提供友好访问的页面向导帮助用户实现信息录入;而针对测序文件上传,GSA提供基于IPV4和IPV6两条网络链路的FTP服务器,确保数据传输的高效性。GSA系统实现了数据全局检索功能,并对检索的结果进行分类统计;同时,用户可以预览检索出的每一个数据的详细信息。
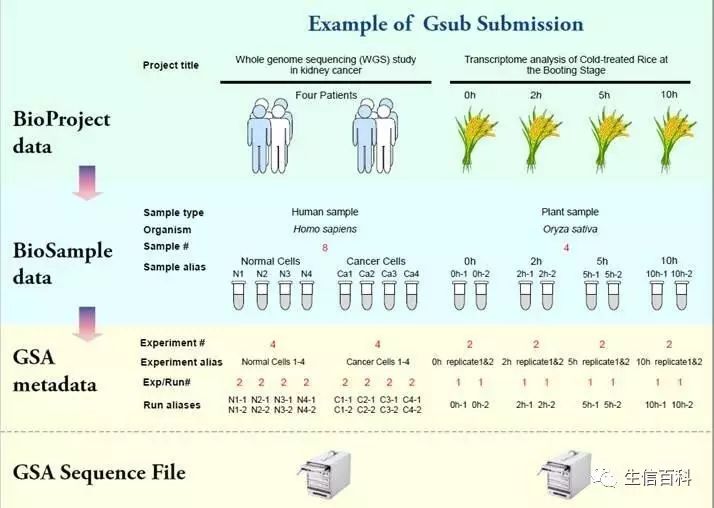
图3 GSA 数据提交
数据库目前使用情况
目前数据库已经被数十个国际期刊认可,包括众多高水平研究期刊,并有多篇文章引用本数据库序列号。

这对我们国人来讲,具有重要的意义!
这意味着中国的生物科研又向前迈进了一大步,也意味着中国的话语权又得到了进一步提升!
GSA 数据被国际期刊接收情况
以下所有文章均使用GSA数据库。
杂志 | 文章名 | 年份 | Accession Number |
Cell | 3D Chromatin Structures of Mature Gametes and Structural Reprogramming during Mammalian Embryogenesis. | 2017 | PRJCA000241 |
Nature | m6A modulates haematopoietic stem and progenitor cell specification | 2017 | PRJCA000469 |
Proc Natl Acad Sci U S A | Extremely high genetic diversity in a single tumor points to prevalence of non-Darwinian cell evolution | 2015 | PRJCA000091 |
Integrated analysis of phenome, genome, and transcriptome of hybrid rice uncovered multiple heterosis-related loci for yield increase | 2016 | PRJCA000131 |
American journal of human genetics | Ancestral Origins and Genetic History of Tibetan Highlanders | 2016 | PRJCA000246 |
Cell research | Loss of 5-hydroxymethylcytosine is linked to gene body hypermethylation in kidney cancer | 2016 | PRJCA000102 |
5-methylcytosine promotes mRNA export — NSUN2 as the methyltransferase and ALYREF as an m5C reader | 2017 | PRJCA000315 |
cell stem cell | Zika Virus Disrupts Neural Progenitor Development and Leads to Microcephaly in Mice | 2016 | PRJCA000267 |
Current Biology | Convergent Evolution of Rumen Microbiomes in High-Altitude Mammals | 2016 | PRJCA000114–PRJCA000116 |
Front Cell Infect Microbiol
|











