小编之前介绍了采用高通量测序手段来获取染色体三维结构信息相关知识如下:
3D基因组之Hi-C简介(一)
3D基因组之Hi-C简介(二)
Hi-C技术知识汇总
大家对采用生物信息学手段预测三维结构有了较好的理解,今天小编着重介绍一下如何对预测结果进行实验验证。用到的技术主要是原位杂交技术。
原位杂交技术(In situ hybridization,简称 ISH)是 1969 年由 Gall 和Pardue 提出的,也称作杂交组织化学或细胞学的杂交是一种能够从形态学上证明特异性的DNA或RNA序列存在于个别细胞、组织部分,单细胞或染色体中的技术,是分子生物学和组织化学、细胞学结合的产物,这项技术是从Southern和Northern技术衍生而来,包括染色体原位杂交和RNA原位杂交,其原理是根据核酸分子碱基互补配对的原则(A-T,A-U,G-C),将有放射性或非放射性标记的外源核酸(探针)与染色体上经过变性后的单链 DNA 互补配对,结合成专一的核酸杂交分子,再经一定的检测手段,将待测核酸在染色体上的位置显示出来,从而确定待测核酸是否与探针序列具有同源性, 达到鉴定靶核酸序列性质的目的。原位杂交技术是重复DNA序列和多拷贝基因家族物理做图、低拷贝及单拷贝DNA序列定位的常规方法,也是检测种间杂种染色体易位重组的有效手段,被广泛用于分析染色体的结构和异源多倍体物种的进化而植物核基因组的一个显著特征是其内含有大量的DNA重复序列,因此它们在核基因组结构和功能研究中居于举足轻重的地位。一些DNA重复序列已被日趋广泛地作为分子标记用于构建遗传图谱、鉴别品种、研究进化和分离目标基因等。所以近年来,原位杂交技术在植物科学研究中尤其是染色体工程中得到了广泛应用(我是本身研究方向就是研究小麦这个复杂的异源多倍体,而且主要研究染色体行为这方面所以我主要介绍染色体原位杂交这项技术)
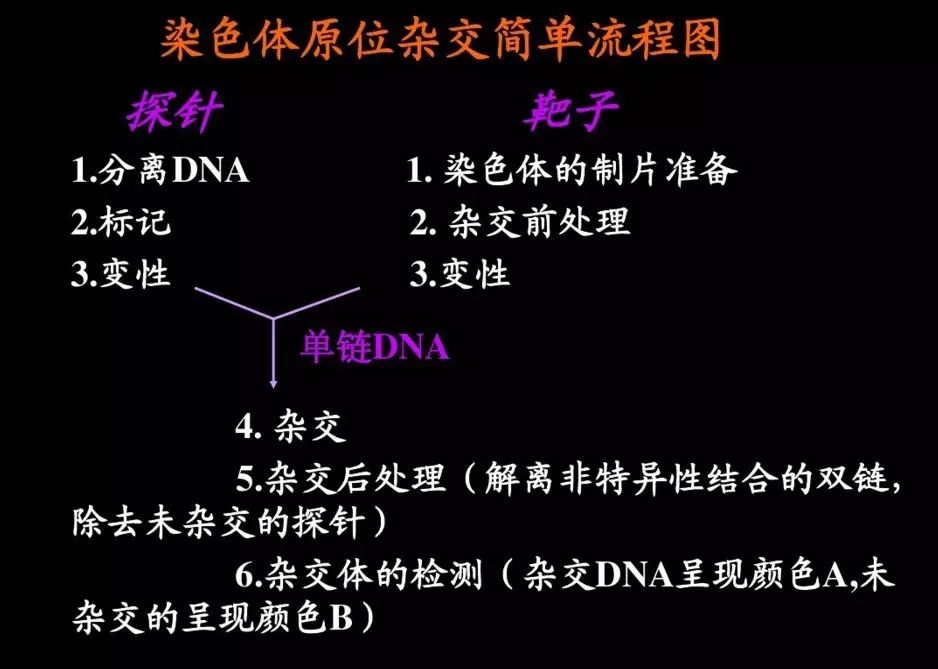
原位杂交技术主要方法有,荧光原位杂交,基因组原位杂交,原位PCR,引物原位杂交技术,原位杂交结合显带标记技术,原位杂交结合棉衣组织化学的双标记技术,电镜原位杂交技术,整体荧光原位杂交技术,DNA纤维荧光原位杂交fiber-FISH。我这里主要介绍荧光原位杂交和基因组原位杂交。
荧光原位杂交技术(fluorescence in situ hybridization,FISH)是原位杂交技术大家族的一员,用其所用探针被荧光物质标记(间接或者直接)而得名。它的基本原理是:如果被检测的染色体或DNA纤维切片上的靶DNA与所用的核酸探针是同源互补的,二者经变性-退火-复性,即可形成靶DNA与核酸探针的杂交体。将核酸探针的某一种
核苷酸
标记上报告分子如生物素、地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的免疫化学反应,经荧光检测体系在镜下对待测DNA进行定性、定量或相对定位分析基本实验流程。FISH样本的制备→探针的制备→探针标记→杂交→(染色体显带)→荧光显微镜检测→结果分析。其优点1、荧光试剂和探针经济、安全;2、探针稳定,一次标记后可在两年内使用;3、实验周期短、能迅速得到结果、特异性好、定位准确;4、FISH可定位长度在1kb的DNA序列,其灵敏度与放射性探针相当;5、多色FISH通过在同一个核中显示不同的颜色可同时检测多种序列;6、既可以在玻片上显示中期染色体数量或结构的变化,也可以在悬液中显示间期染色体DNA的结构。缺点:不能达到100%杂交,特别是在应用较短的
cDNA
探针
时效率明显下降。
在FISH技术基本确立之后,FISH不仅用于单基因或核酸检测,FISH 技术的进一步发展扩展到多色FISH多基因位点同时检测,从基因检测发展到基因组、染色体、活细胞中转录产物mRNAs原位检测以及组织水平的核酸检测,并且在今后的研究中还有可能应用到整个生物体的检测通过
比较基因组杂交
(Comparativegenomichybridization)用于检测染色体区域的缺失和重复。大片段探针一旦和样品非特异性结合就会形成一个信号,混淆染色体上基因的检测,就需要剪切成小片段<200核苷酸。现在检测手段的提高以及检测软件的不断发展使得FISH技术的检测要求越来越低,灵敏度越来越高。精确的计算机图片处理算法的不断改进形成了亚显微水平的探针高分辨技术。随着检测目标越来越小,FFISH技术已用于隐蔽的亚端粒核型
基因重排
和精确的染色体作图及单拷贝mRNA的检测。
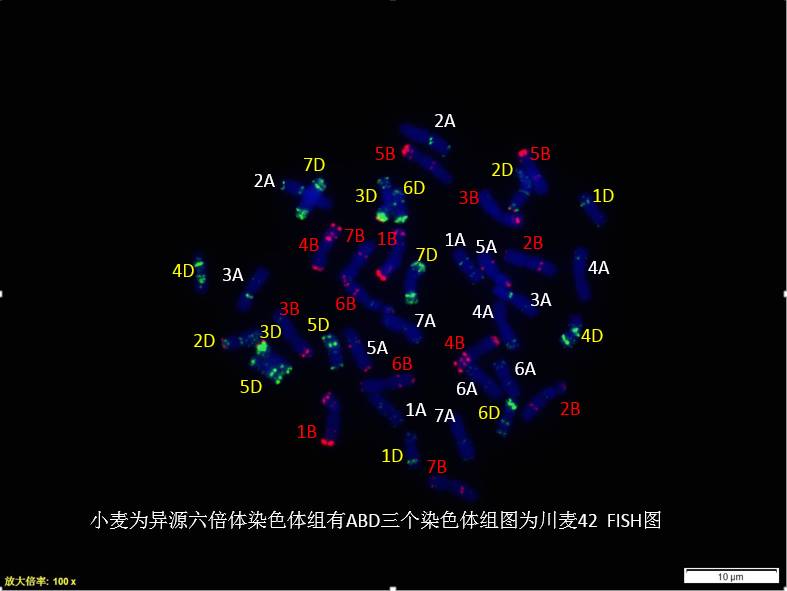
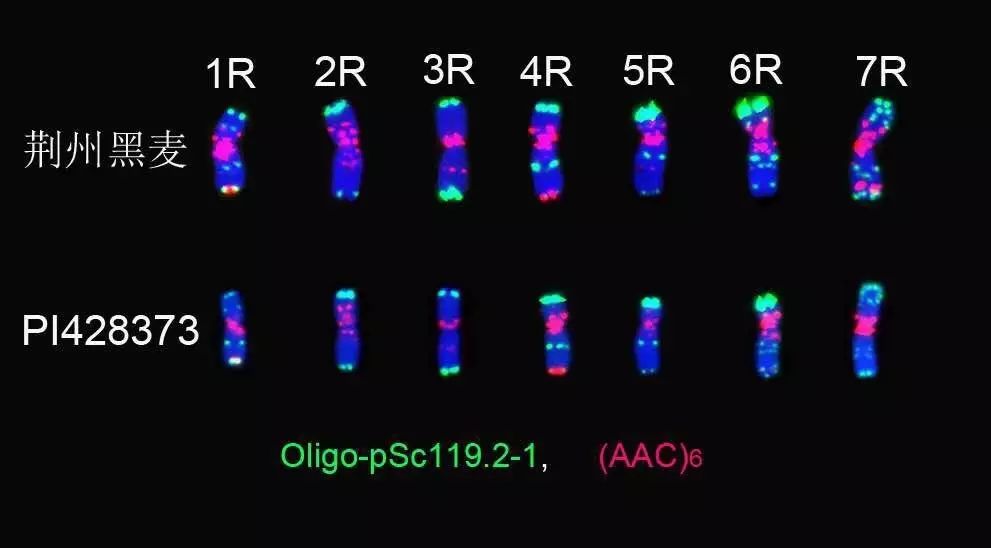
基因组原位杂交(Genomic in situ hybridiza-tion,GISH)是20世纪80年代末,由染色体原位抑制杂交(Chromosome in situ suppression CISS)衍生而来的一种重要的原位杂交技术,是一个精确、安全的研究工具,在分子细胞遗传学领域发挥着重要作用。它将供体总基因组DNA切割成合适片段,标记后作为探针,用受体总基因组DNA以适当的浓度进行封阻,在杂种细胞染色体上进行原位杂交。在封阻DNA和标记DNA探针之间,封阻DNA优先与一般序列杂交,剩下的特异性序列主要被标记探针所杂交,最后检测出不同来源的染色体组、染色体或染色体片段。GISH最初应用于动物、医学等方面的研究。利用黑麦总基因组DNA直接鉴别面包小麦品种中的黑麦染色体片段,这是该技术首次在植物中应用。之后得到迅速推广,在麦类、玉米、棉花、水稻等主要作物有大量报道,在油菜、大豆、柑橘、烟草、百合、番茄等植物中亦见应用。目前,已成为植物分子细胞遗传学的重要研究手段。其原理和荧光原位杂交相同。
基因组原位杂交技术作为植物分子细胞遗传学研究的重要手段,以其安全、精确、直观、信息丰富的特点,被广泛地应用于杂种染色体分析如亲本染色体的识别,外源染色体片段的识别和定位,易位系,代换系和附加系的鉴别,物种的起源、进化和亲缘关系探索,染色体行为考察等方面
2
个物种之间的关系越近,其基因组间的相似性就越强,在GISH上就出现越多的“杂交”位点。所以,根据杂交信号的强弱可以推测2个基因组间相似程度的高低,这是GISH技术研究基因组间关系的基础。其特点之一,就是用全部的基因组DNA参与杂交,从而能够全面地反映探针和靶序列之间在基因组水平上的相似性,避免了将序列片段直接定位于靶目标,来探测整个基因组关系时所暴露出的局限性,具有较高的定性价值。当然,GISH技术本身也有一定的局限性,仅靠GISH一项技术,不能区分导入的具体是哪条染色体或片段。同时,GISH还有一个在植物中的适用性问题。有报道说亲本间亲缘关系较近时或基因组同源性较高时,应用GISH方法难以区别开来。但有实验证明,GISH有时能分辨被认为具有很近的亲缘关系的种,而有时在属水平上分辨却相当困难。因此,建立分子与分类学的亲缘性之间的相关性是GISH的一个研
究方向。
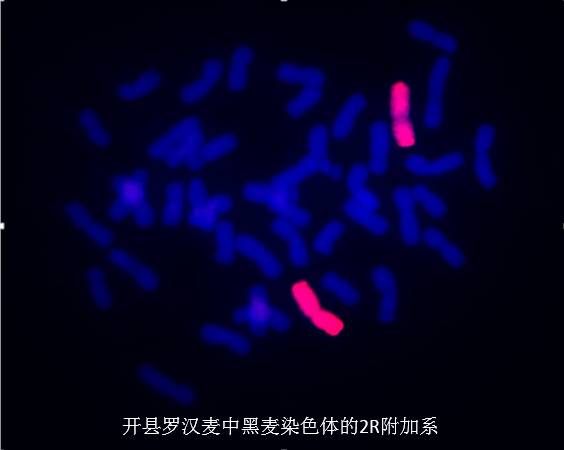
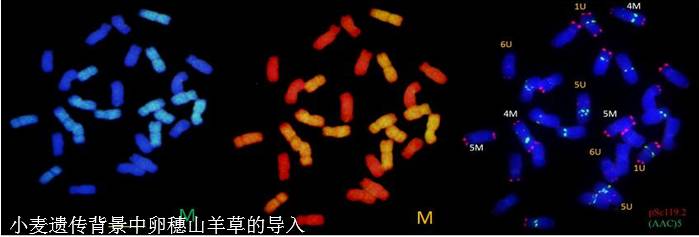
以上图片均为我个人实验所得下面附上我FISH试验详细步骤:
1
根尖准备
将每个材料的种子放在有两层滤纸的培养皿中,加入足量的水,放于4℃冰箱,24小时后,将其拿出,吸干多余的水分,置于恒温培养箱中,待根长到2厘米左右时,剪取根尖放入离心管中,先用笑气处理2小时,再用冰醋酸处理5分钟,然后保存在70%酒精中,-20℃保存(Kato 1999)。
2
制片
将保存的根尖从酒精中取出,用双蒸水清洗干净,剪去根尖的分生区,放入质量比为4:2的纤维素酶和果胶酶中,37℃ 水浴酶解45分钟,然后用双蒸水清洗两次,洗去酶液,以防后面加酒精产生沉淀,再用无水乙醇清洗2次,吸干酒精,加入20ul冰乙酸 (一个根尖用量)捣碎,制成悬浮液。吸取10ul悬浮液滴到干净的载玻片上,等晾干后于X21显微镜镜检,选取分裂象比较好的载玻片放于-20℃保存
3
荧光原位杂交
(a).
将-20℃保存的载玻片取出,放于37℃恒温箱中烘干,相差显微镜检查已干燥的载玻片,确定中期细胞及染色体的形态是否保持完好。
(b).
将载玻片放入4% 多聚甲醛中处理10min(通风橱中进行)。
(c).
用2×SSC缓冲液处理两次,每次5min。
(d).
依次在70%,95%,100%梯度酒精中处理一次,每次5min,晾干。
(e).
在晾干的载玻片上加60ul 70% FA(去离子甲酰胺,Deionized formamide),盖上24cm×24cm盖玻片,在80℃条件下变性2min。














