在上一期的推文中,小编为大家介绍了
公共数据库挖掘必备-QTL分析
,帮助了一些被
GWAS
数据困扰,无法对显著的
SNP
位点功能进行解释的同学们,让他们对自己的研究有了一些新的思路,小编是非常开心的。由于在公共数据库的资源中包含了大量的芯片数据,所以今天小编为大家介绍两款差异分析的软件,基本上可以满足大家对于芯片差异分析的所有要求。这两个软件就是
Transcriptome Analysis Console
和
GeneSpring
。
由于大家对于
Gene Expression Omnibus
数据库了解很多了
(GEO
,
https://www.ncbi.nlm.nih.gov/gds/)
,我就在此处不多讲了。此外,还有一个很大的表达数据库,是
ArrayExpress
(https://www.ebi.ac.uk/arrayexpress/)
,大家如果需要寻找感兴趣的疾病数据,不防来这个数据库里搜索一下,记得要一起下载好
芯片的注释文件
。小编尝试过,数据还是非常丰富的。
Transcriptome Analysis Console
首先,为大家介绍的差异分析软件是
Transcriptome Analysis Console
。这款软件是由
ThermoFish
公司开发,和
GeneSpring
相比,最大的优点就是完全免费,理论上来说也更加适用于
Affymetrix
的芯片。这个软件可以处理的数据包括:
Gene Arrays, 3‘Expression Arrays, Exon Arrays, miRNA Arrays
等。
此处,我们以
Affymetrix miRNA 4.0
版本的芯片为例。其分析流程如下图所示:
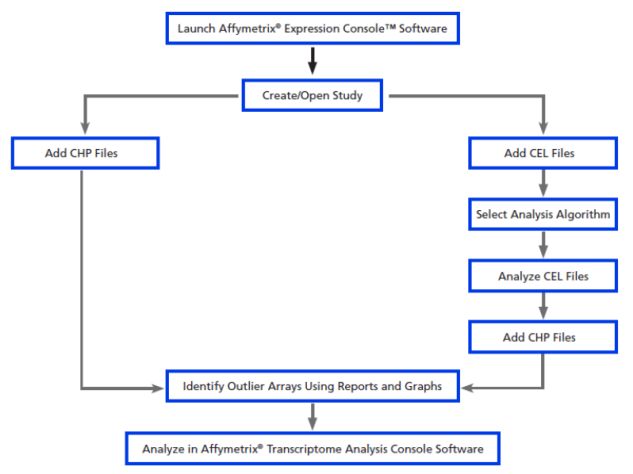
1. 首先大家需要注册一个登录账户,并且下载好你将要分析的芯片的注释;
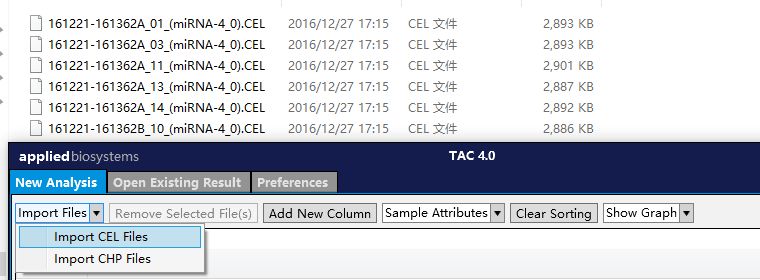
2. 将.CEL的芯片数据导入,选择Import CEL Files;
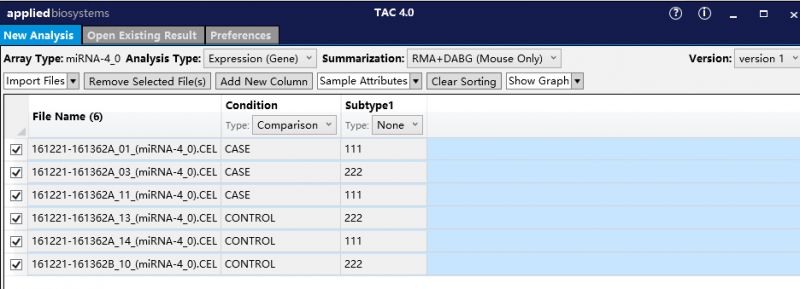
3. 在Condition中可以标记样本的不用处理,此处选择Comparison,对于有多种不同亚分组的样本,也可以对样本进行进一步分类;在Summarization中需要选择你所研究的不同物种,我们是小鼠的样本,所以选择Mouse Only;
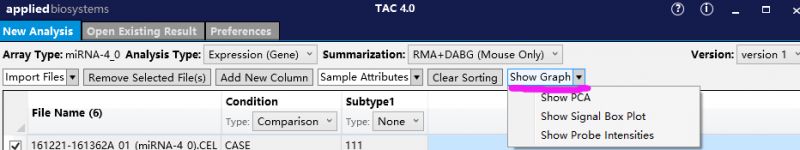
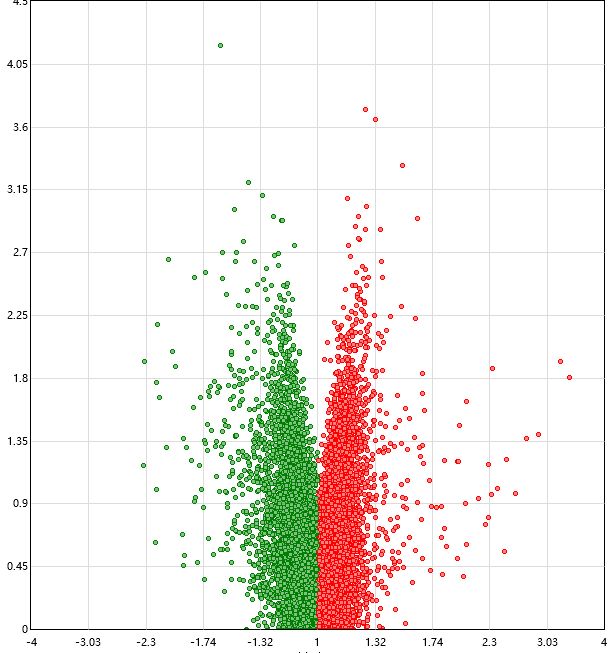
4. 在这一步就可以对你的数据进行一个基本的分析了,如PCA, Signal Box Plot, Probe intensities,这些数据可以帮助你对于芯片测序的基本结果有一个大体上的了解;
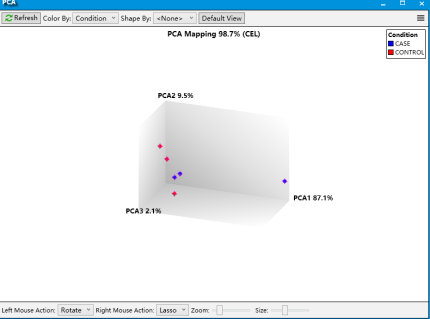
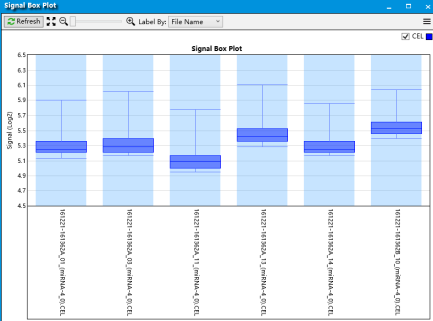
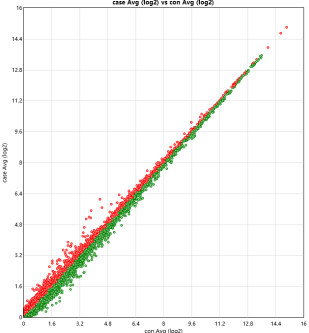
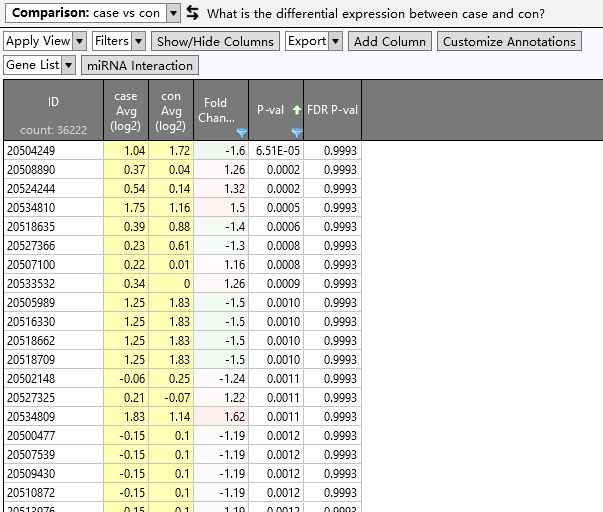
5. 点击Run Analysis,就可以得到差异基因,并且生成所需要的图,你所需要做的就是将图和结果导出来就可以了。
对于
GeneSpring
的强大,小便就不重复了,大量文献中芯片差异分析都是用的这个软件,该软件是由安捷伦公司开发的。此处小编给大家做一个使用的介绍。
1. 在导入.CEL文件时,要确定软件是否可以识别你的芯片,如果不行就尝试以下两种办法,1是下载的column中查找,如果还没有就要在公共数据库中下载你的芯片的注释信息,普遍来说,注释信息作者都会和数据一起上传。此处为大家讲解如何自己建立一个注释信息的平台。
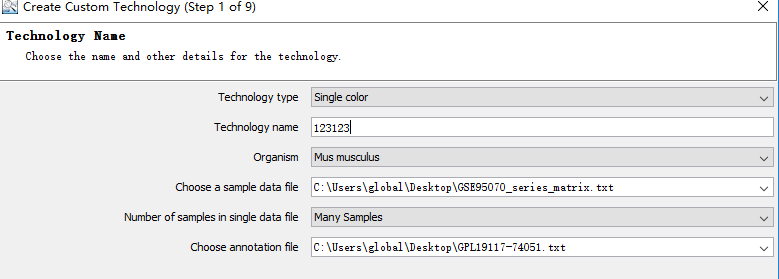
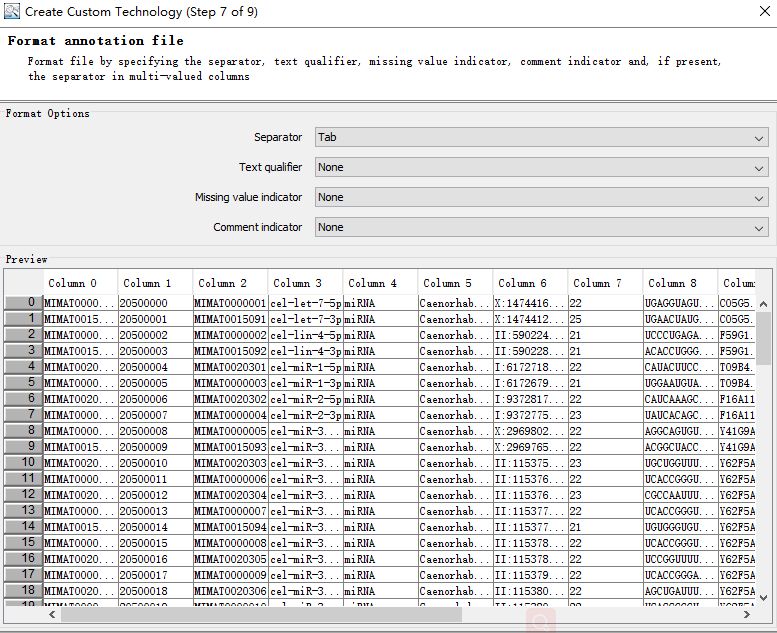
2. 选择Annotation,Create Technology,Custom from file选项。之后选择物种,并且导入你所要研究的芯片表达信号矩阵和注释矩阵。此处需要注意的是,表达矩阵和注释矩阵需要修改为可以识别的格式。

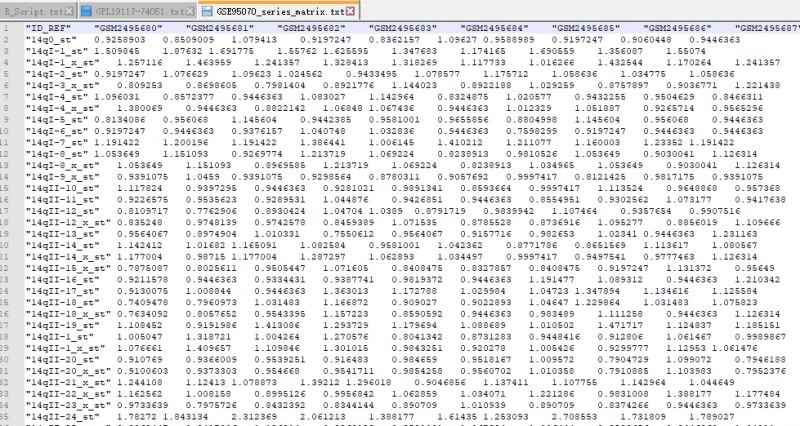
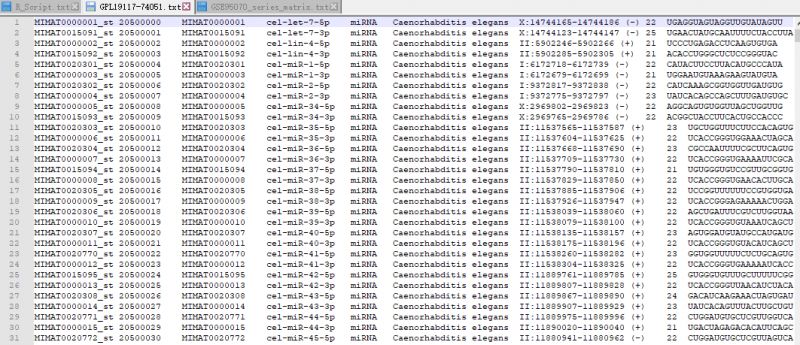
3. Text qualifer选择双引号,数据会将文件自动分开,其余选项默认就可以。之后的标题格式调整也不需要做任何修改。
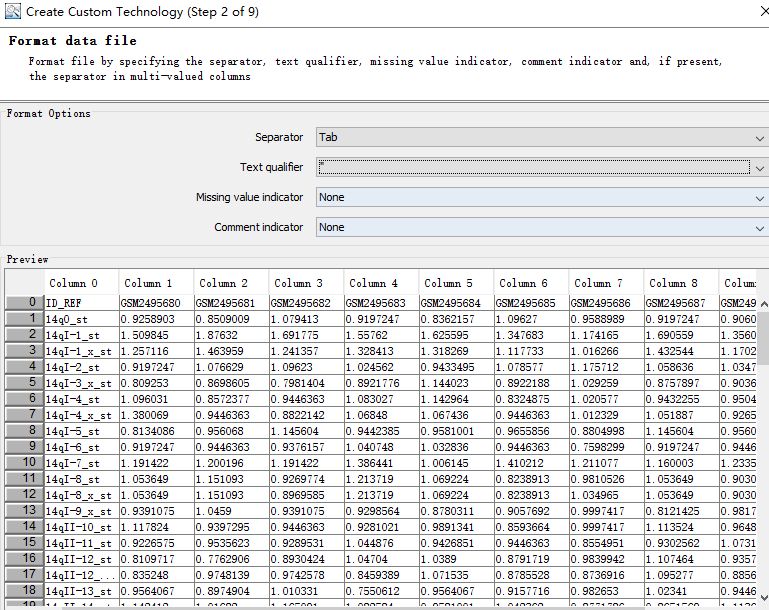
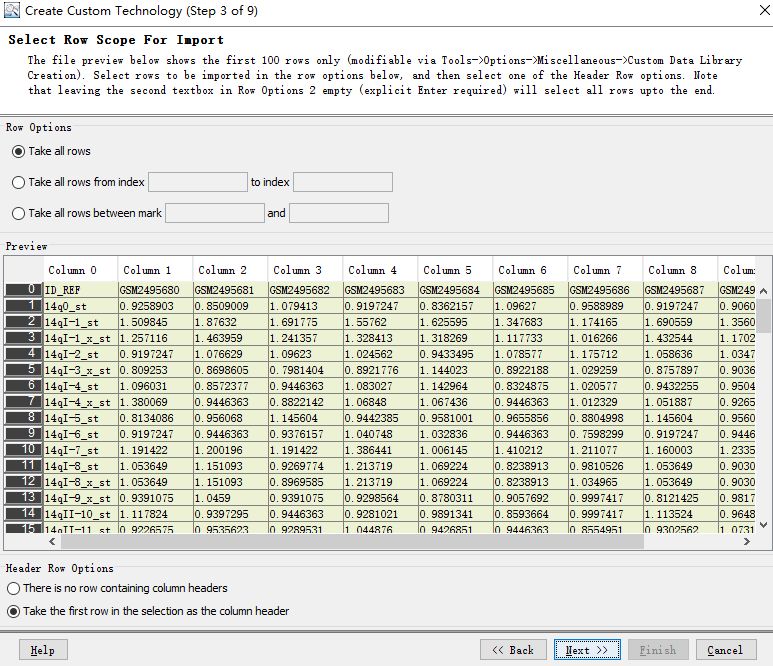
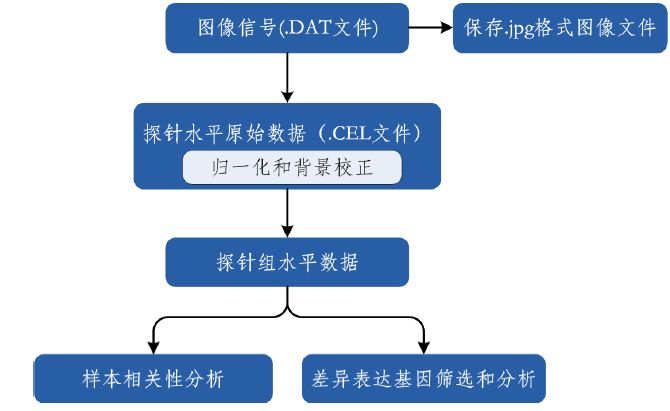
4. 定义信号行,操作如下图所示。之后是对注释文件的读取,默认参数,一路Next就好。
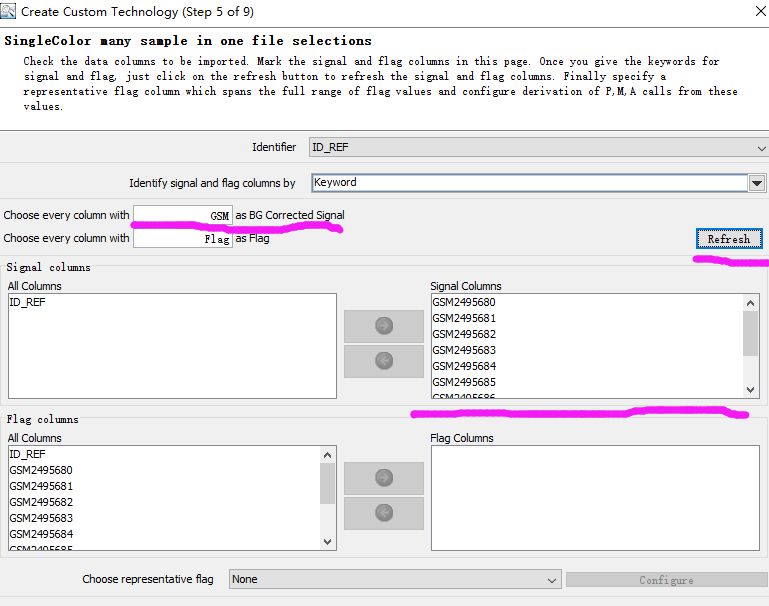
5. 将定义的信息和矩阵的信息对应起来,不同信息类型仔细分类,记得检查它们是一一对应的。
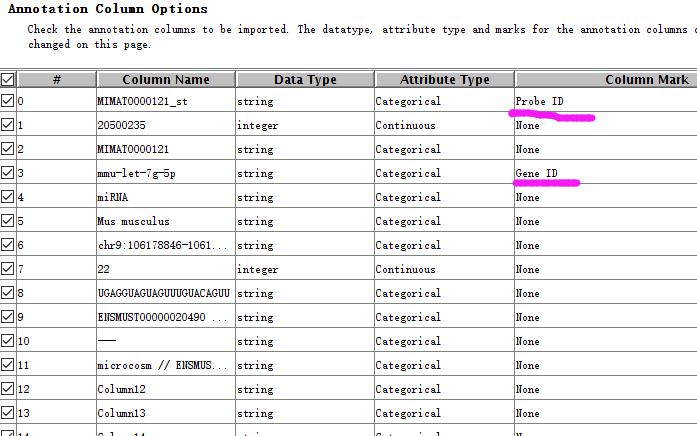
6. 此时注释文件平台就搭建好了,现在要做的就是导入你的数据了。点击Project, New Experiment
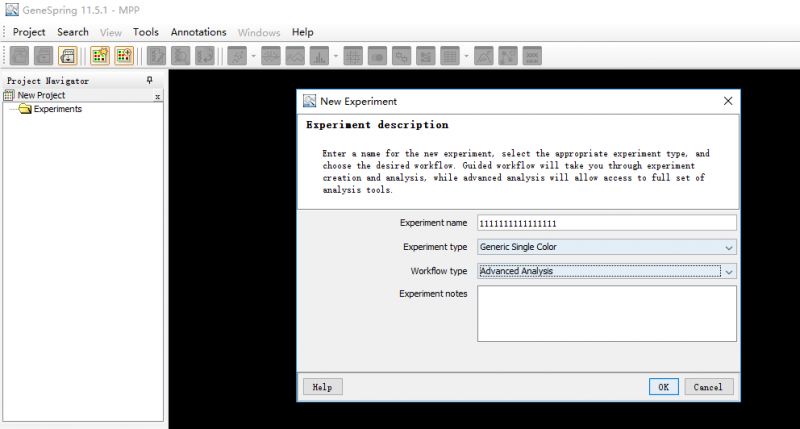
7. 如果你使用的数据是GEO下载的数据,就不需要标准化了。如果是新的数据,还有必要做一次标准化的处理。
8. 之后就是在Workflow中的Experiment Grouping对样本进行分类,定义CASE/CONTROL,求差异,画图。
相对于
GeneSpring
来说,
Transcriptome Analysis Console
有两个优势。第一就是这个软件是免费使用的,下载好注册后就可以使用了;第二就是在软件中就可以下载好芯片注释文件,对于
GeneSpring
来说,很多的注释文件都需要人工的导入其中,比较繁琐。但是就功能来说,两个软件都是非常强大的。更多的功能还需要你去仔细研究。
本文为生信草堂原创,欢迎个人转发分享,其它媒体或网站如需转载,请在正文前注明转自生信草堂并联系bioinformatics88
全集|生信方法+数据库挖掘+精品论文解读(更新)
公共数据库挖掘必备-QTL分析
公共数据库也能发NG—lncRNA研究新思路




