欢迎关注”生信修炼手册”!
对于任意的表达量数据,定量加差异分析都是一套经典的组合拳。当我们想要展示特定基因的组间差异结果时,下面这种图表就派上了用场
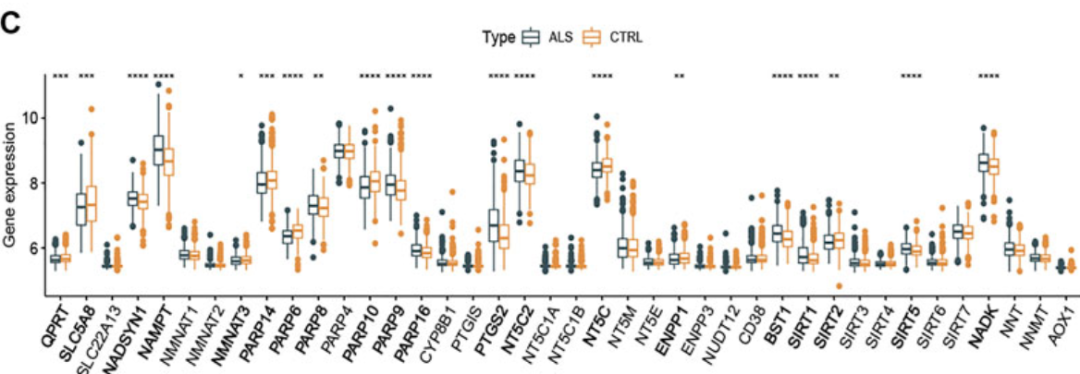
横坐标为基因,纵坐标是基因表达量,每一组的表达量采用了箱体图的形式来展现,当然也可以换成小提琴图等其他描述总体分布的可视化方式。对于每一个基因,通过并列的两组箱体来定性的展示两组间的分布差异,而图中星号则表示差异分析的p值,定量展示差异的显著性,通过这种图表,可以直观的展示差异分析结果。
对于这样的图表,推荐使用ggpubr这个R包,这个包是ggplot2的一个扩展,所以其画图的语法遵循ggplot2的定义,简单理解就是每一个属性都对应数据框的每一列,所以我们首要任务是构建一个绘图用的数据框,这个数据框的每一列对应图中的一个元素
1. 第一列,对应x轴,即基因名称
2. 第二列,对应y轴,即基因表达量
3. 第三列,对应样本
3. 第四列,对应图例中的不同颜色,即样本分组
数据准备的代码如下
# 1.基因表达量数据# 纯文本文件,每一行为基因,每一列为样本> data <- read.table("data.txt", header = T, sep = "\t", row.names = 1)> data[1:5, 1:5] GSM3076582 GSM3076584 GSM3076586 GSM3076588 GSM3076590PNP 7.123107 7.115196 7.103920 7.377837 6.960771PTGIS 2.483152 4.215764 4.174901 3.658576 3.296362PTGS2 6.156447 6.539128 6.294466 4.918229 6.269206NT5C1B 1.912044 3.691517 3.771510 4.546582 3.784036SIRT3 4.210383 3.960023 3.623266 4.658751 4.347605# 2.样本的注释信息# 纯文本文件,每一行为样本,每一列为一种注释信息> group <- read.table("sample.group.txt", header = T, sep = "\t", row.names = 1, stringsAsFactors = T)> head(group) group sex seriesGSM3076582 ALS m GSE112676GSM3076584 ALS m GSE112676GSM3076586 ALS f GSE112676GSM3076588 CON m GSE112676GSM3076590 CON m GSE112676GSM3076592 CON f GSE112676> library(tidyr)# 合并样本分组信息和表达量信息> plot_data <- cbind(sample = rownames(group), group = group$group, as.data.frame(t(data))++ )> head(plot_data[, 1:10]) sample group PNP PTGIS PTGS2 NT5C1B SIRT3 ASPDHGSM3076582 GSM3076582 ALS 7.123107 2.483152 6.156447 1.912044 4.210383 4.504081GSM3076584 GSM3076584 ALS 7.115196 4.215764 6.539128 3.691517 3.960023 3.963059GSM3076586 GSM3076586 ALS 7.103920 4.174901 6.294466 3.771510 3.623266 4.367432GSM3076588 GSM3076588 CON 7.377837 3.658576 4.918229 4.546582 4.658751 4.853395GSM3076590 GSM3076590 CON 6.960771 3.296362 6.269206 3.784036 4.347605 4.249152GSM3076592 GSM3076592 CON 7.222664 3.829693 5.185523 4.473507 4.348066 4.399335 RNLS NADK2GSM3076582 5.937300 4.224377GSM3076584 4.993297 4.035023GSM3076586 5.408174 3.590138GSM3076588 5.431993 3.921544GSM3076590 5.168883 3.773122GSM3076592 5.860502 4.037075# 数据转换,wide => long> res <- pivot_longer(+ data = plot_data,+ cols = 3:ncol(plot_data),+ names_to = "gene",+ values_to = "value",+ values_drop_na = TRUE+ )> head(res)# A tibble: 6 x 4 sample group gene value <chr> <fct> <chr> <dbl>1 GSM3076582 ALS PNP 7.122 GSM3076582 ALS PTGIS 2.483 GSM3076582 ALS PTGS2 6.164 GSM3076582 ALS NT5C1B 1.915 GSM3076582 ALS SIRT3 4.216 GSM3076582 ALS ASPDH 4.50
tidyr是一个数据转换的包,可以帮助我们快速的从常规的宽格式的表达量数据转换到绘图使用的长数据,准备好绘图用的数据之后,先用ggpubr的ggboxplot函数绘制最基本的箱体图,代码如下
> library(tidyr)> ggboxplot(res, x = "gene", y = "value", color = "group")
效果图如下
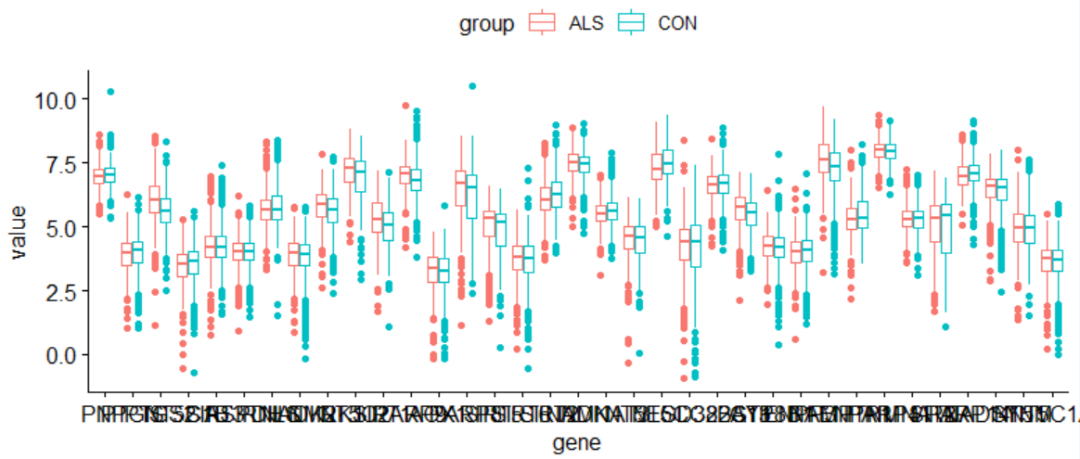
在基础图形的基础上,需要做如下调整
1. 调整x轴标签的排列方式,改为旋转排列
2. 调整分组的颜色设置
3. 调整x轴,y轴,图例的文字





