今天我又学习了一天的KEGG,几乎把KEGG的网页的每个链接点了一遍,还有几个功能没有点呢。各位小伙伴,学习数据库呢还是要耐下心来把上面的功能都试一遍,多熟悉熟悉就知道怎么用了。今天主要分享我觉得比较有点技术含量的知识点,虽然我还不知道KEGG数据下来怎么用,
基因批量注释到代谢通路并获得彩色的通路图有啥用~原帖比较坑,竟然代码有错误,还好我会改~
KEGG数据下载
1 首先打开KEGG官方网站 http://www.genome.jp/kegg/catalog/org_list.html,网页中展示出了各个物种的分类、拉丁名称、英文名称等信息。
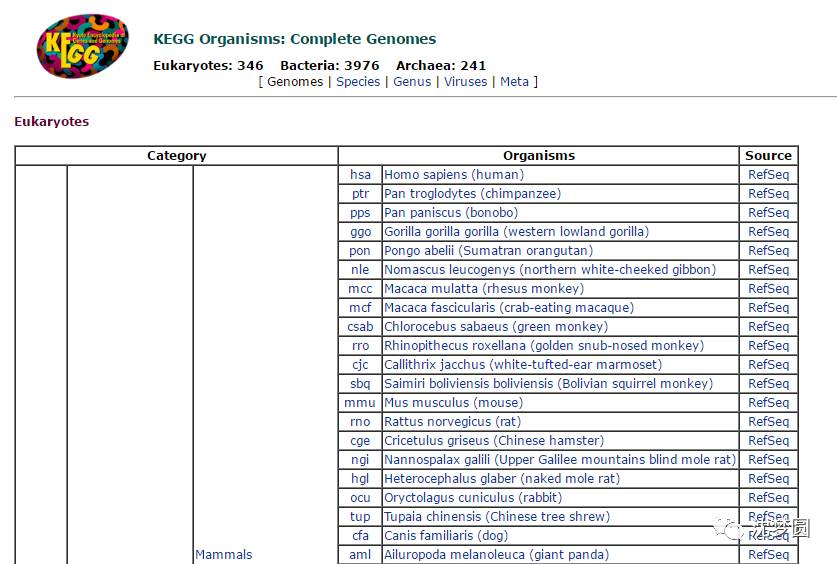
2 直接网页中搜索(Ctrl + F)需要下载的物种英文名称或拉丁名。如果不确定物种名称,网站中提供了详细的分类系统,也可根据前面的物种分类信息进行查找。
本文以拟南芥为例,搜索“Arabidopsis thaliana”即可找到。找到后点击物种名称前的3个字母缩写链接(下图红色框中的位置)。

3 进入后的网页中包含了物种的一些基因组信息,点击上方的“Brite hierarchy”,进入后再点击“KEGG Orthology (KO)”;
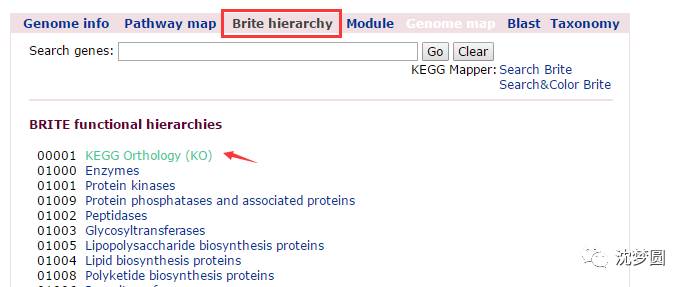
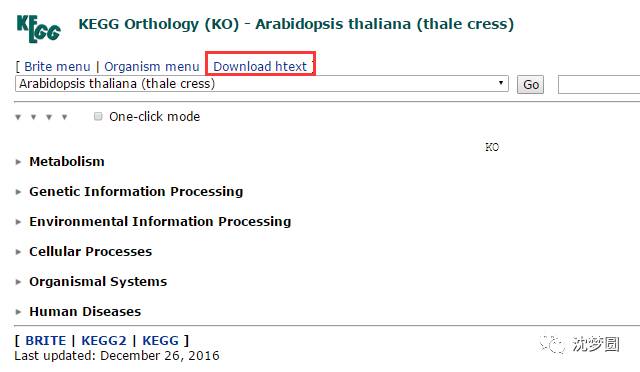
4 在跳转出的网页中点击“Download htext”,弹出下载窗口进行下载,国外网站有时会出现无法下载的情况,多试几次即可;
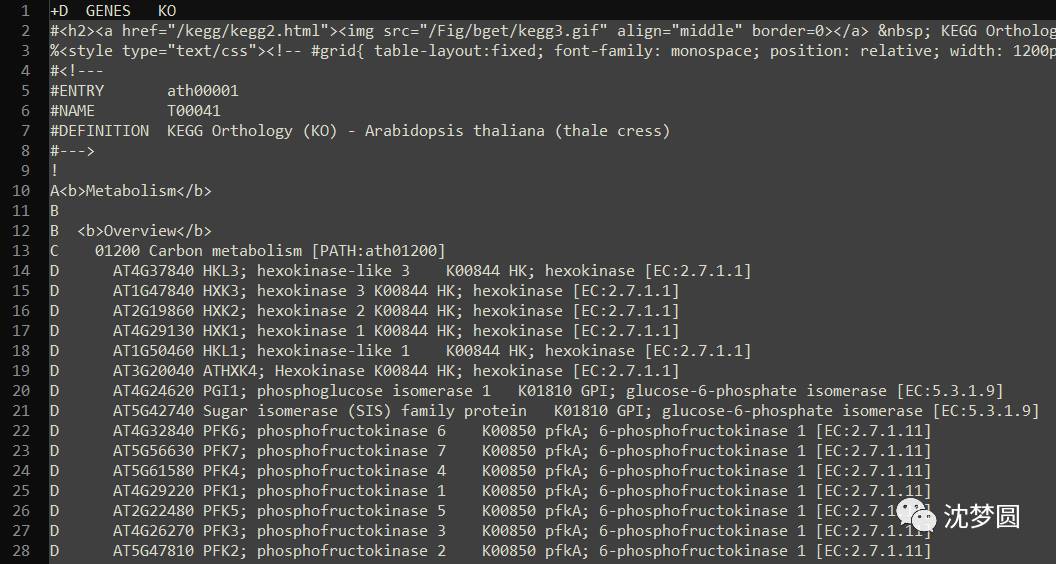
5 当然,下载好之后还没有结束。下载得到文本文件,可以看到里面的结构层次非常清楚,C开头的就是kegg的pathway的ID所在行,D开头的就是属于它的kegg的所有的基因。A,B是kegg的分类,总共是6个大类,42个小类。
6 我们要进一步把它转成可以直接使用的格式。在shell中输入如下命令(加粗部分为下载的原始文件,请修改成需要的名字):
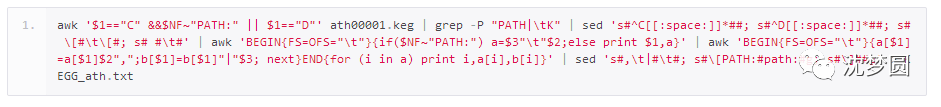
即可直接生成处理好的KEGG结果。第一列为拟南芥的基因名称,后面就是该基因的KEGG数据,一目了然。
需要说明的是,由于物种的不同,第一列也有可能出现GenBank等非基因名称的信息。
基因批量注释到KEGG通路并获得彩色的通路图
以下是一个简洁的批量将基因/蛋白map到KEGG通路中,并在通路中标记出输入的基因,并且将标记后的KEGG通路批量下载到本地的方法。
1.首先你需要有一个基因的ID列表/uniprot的ID列表/KO ID列表
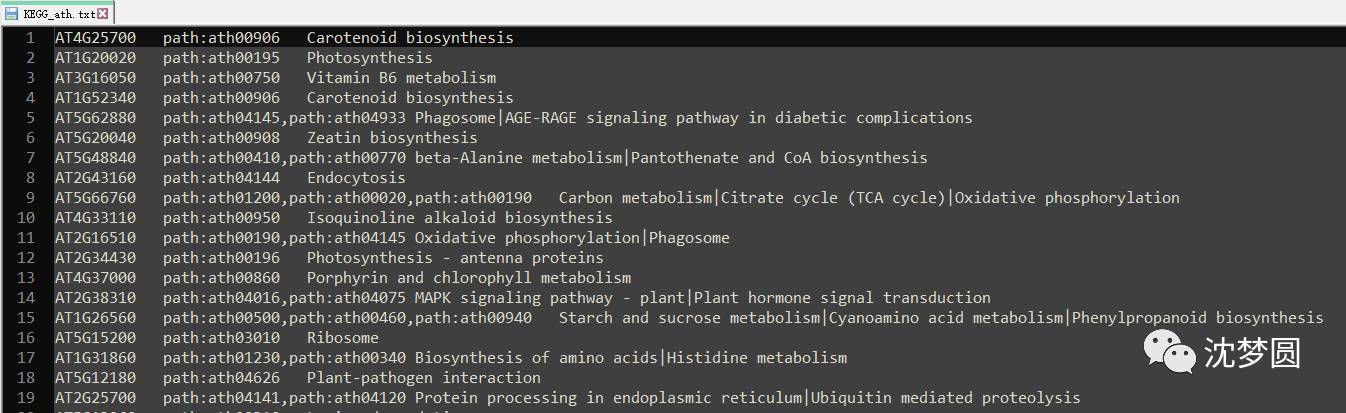
2.打开KEGG Mapper主页http://www.genome.jp/kegg/mapper.html,点击search&color pathway
(Mapping tools中提供了三种类型的pathway选项,①. 只对基因集合所在的KEGG通路进行搜索;②. 对基因集合所在的KEGG通路进行搜索,并将输入的基因进行着色标注。③.对选定的通路中目标基因进行着色(此选项是基于已经有感兴趣的通路,主要看参与感兴趣通路中的目标基因);本例以第二选项为例。 )
3.将ID输入框内,或者浏览上传文件也可
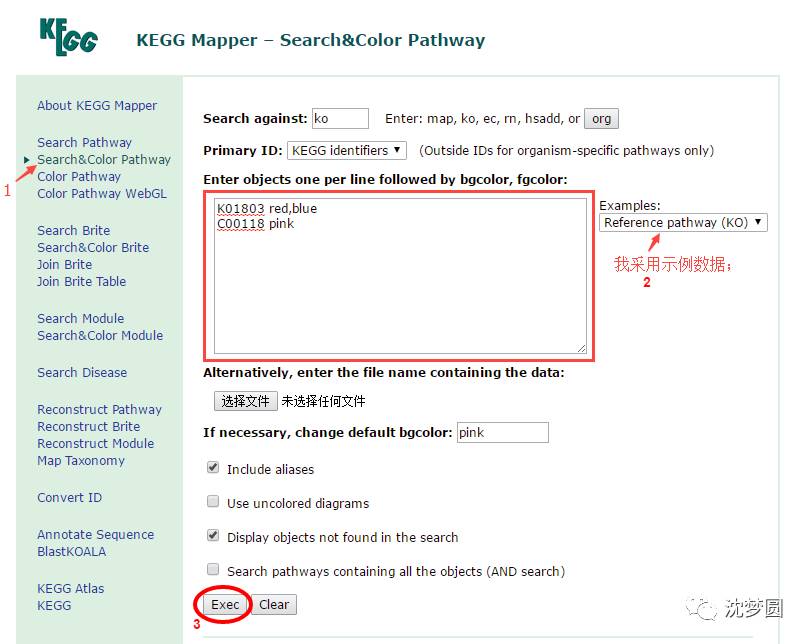
4.点击Exec后返回页面如下:

这时如果一个个点开下载会非常麻烦,尤其在基因数目较多的情况下。
5.在web页面的空白出点击右键→查看源代码,并将代码复制到本地,保存为文本”web.txt”。
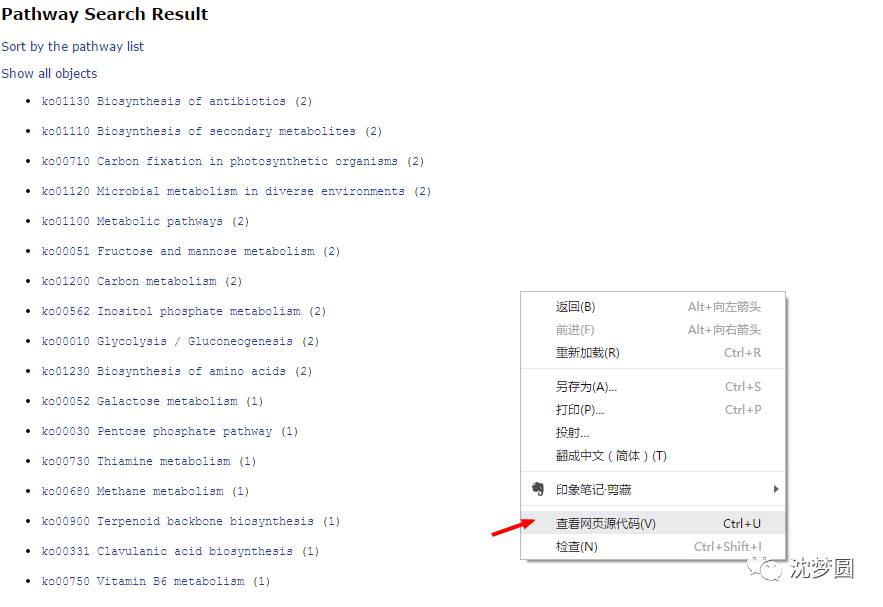
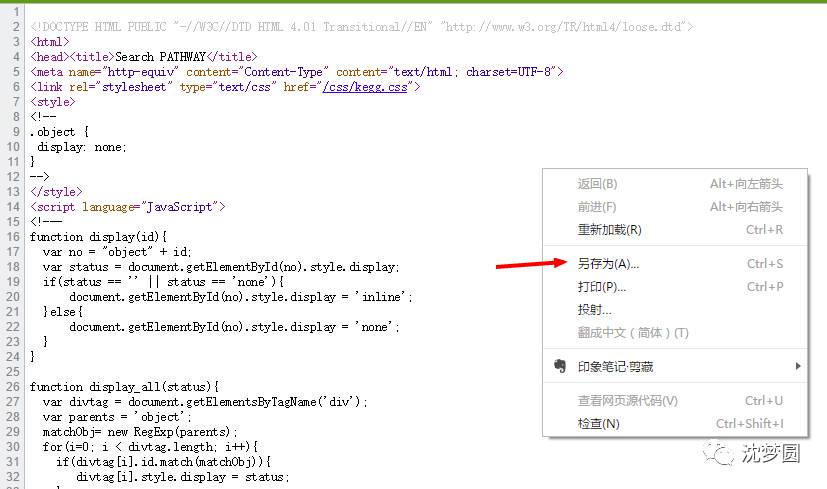
6.首先要获得上面图片中的pathway的url:

此时list文件内容:
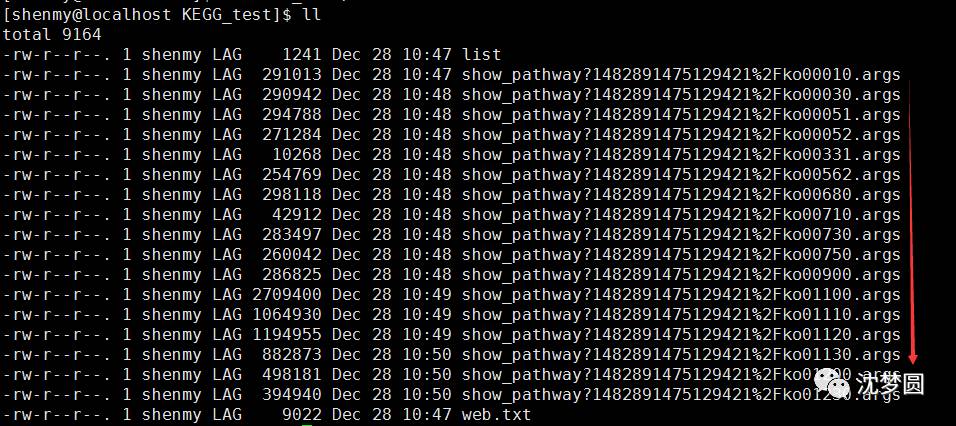
7.循环下载list文件中的链接;

然后我们会得到很多以show打头的文件,这些文件很关键
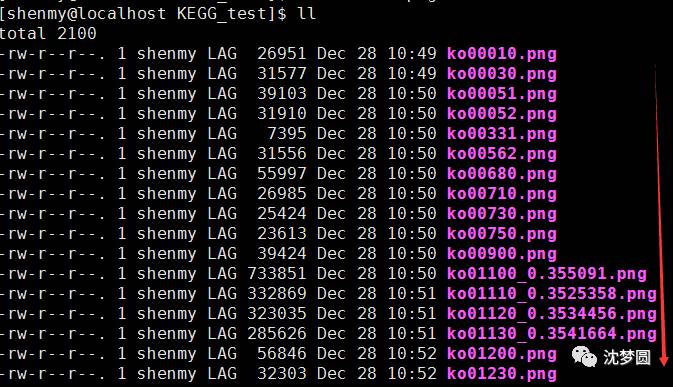
8.从下载到的文件中筛选png文件的地址,并将这些地址下载下来即可;

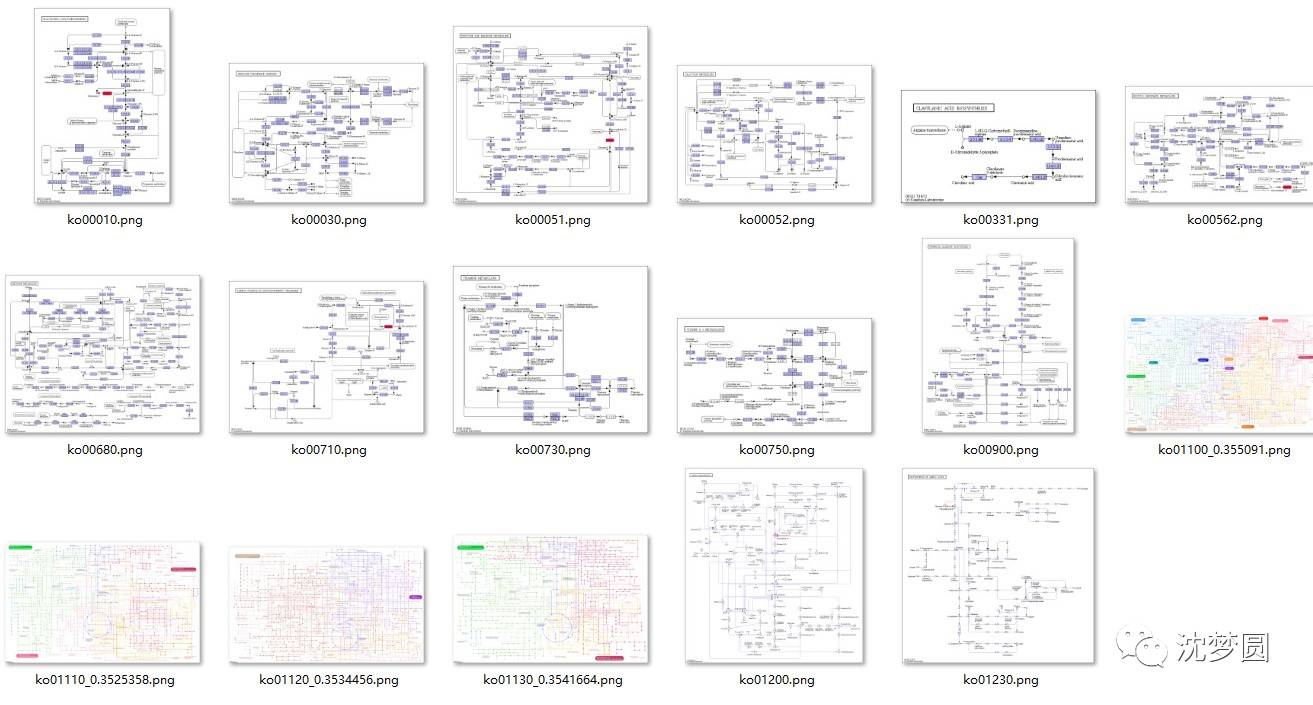
9.OK, 到这里大功告成了!
参考资料


生信菜鸟专栏
是生信技能树论坛的版主团队的专栏,团队成员生信技能背景丰富,文件格式,数据资源,软件使用,脚本技巧,统计绘图,组学实战均有对应人才。而本专栏将从基础到深入,为零基础的各位剖析生信技能。
科研路,不孤单!
^ ^
FS科研软件库,集合60+医学科研必备神器,现在统统打包分享
,
点这里
致敬Scihub|Freescience、生信人要一起做些很Cool的事儿




