话说当年我在暨大工作的时候,学生都很害怕汇报RT-PCR的结果,因为每次一讲到用了t test做了检验,底下某小老板,就一顿狂喷,说学生瞎搞,这不对那不对,但从没告诉正确的姿势,学生们都怕了他。于是我写了这篇文章,普及了一下相关的知识,RT-PCR那点东西,全在这里。
RT-PCR相对定量有两个模型,一是
Δ
Δ
C
t
模型,一个是扩增效率校正模型。这里我们先讨论简单的模型:
Δ
Δ
C
t
模型,在这一模型中,假定扩增效率为2,即每个PCR cycle,产物倍增,由以下公式给出:
R
a
t
i
o
=
2
−
Δ
Δ
C
t
其中ΔΔC
t
= ΔΔC
treated
- ΔΔC
control
, ΔΔC
treated
和ΔΔC
control
分别是treatment组和control组中目标基因和参照基因的Ct差,即ΔΔC
t
= ΔΔC
target
- ΔΔC
reference
.
扩增效率肯定是在(1,2)之间,理想状态下才是2,而理想状态通常是不存在的。这个模型的假设点其实是treatment组和control组的扩增效率一样,至于是不是2,比2小多少,并不影响后续的统计分析。
Data
我们来看以下一份数据,4组重复实验,用RT-PCR测了gene01和gene02的表达量,HK代表house keeping gene,即参照。以下数值为原始的Ct值。
ct sample = rep(rep(1:4, each=3), 2),
treatment = rep(c("Control","Treated"), each=12),
gene01 = c(23.22,23.34,23.12,24.06,24.15,24.15,23.18,23.13,
23.10,24.78,24.45,24.67,23.11,22.99,23.10,22.77,
22.99,23.06,23.73,24.01,23.80,23.73,23.83,23.73),
gene02 = c(29.08,29.04,29.39,28.23,28.01,28.12,28.79,28.43,
28.49,31.37,30.74,31.09,27.11,27.24,27.37,25.52,
25.72,25.52,27.43,26.73,26.65,27.96,27.84,27.98),
HK = c(19.68,19.69,19.80,19.95,19.93,19.97,19.93,20.02,20.27,
19.93,19.88,19.90,20.61,19.98,20.57,19.68,19.95,
19.85,20.27,20.08,20.07,20.10,20.07,20.10)
)
print(ct)
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
#
Data clean
每个sample,测了三次技术重复,我们使用平均值来提高精度。
require(plyr)
#
ct data.frame(gene01=mean(x$gene01),
gene02=mean(x$gene02), HK=mean(x$HK)))
print(ct)
#
#
#
#
#
#
#
#
#
Calculate statistical metric
按照ΔΔC
t
模型,我们先计算ΔC
t
,然后计算ΔΔC
t
,最终计算出Ratio。
delta.ct data.frame(gene01=x$gene01-x$HK,
gene02=x$gene02-x$HK))
print(delta.ct)
#
#
#
#
#
#
#
#
#
#
dd.ct select=c(gene01, gene02)) -
subset(delta.ct,
treatment=="Control",
select=c(gene01, gene02))
print(dd.ct)
#
#
#
#
#
ratio print(ratio)
#
#
#
#
#
这个ratio值计算出,经过扩增后,目的基因在处理组中的产量是对照组的多少倍。
Statistical Analysis
计算出ratio后,很多人就拿它来做t检验。这种做法是不对的,因为ratio的分布明显是右偏的。从理论上看,PCR的扩增可以分成以下三个阶段,指数扩增、线性扩增、平台期。
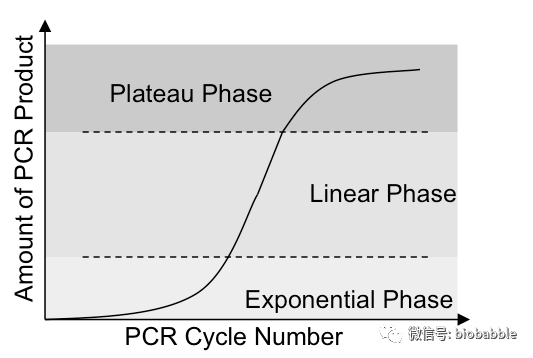
当试剂量足够的时候,就处于指数扩增阶段,只有当PCR试剂不足时,才进入线性扩增期,最后当试剂被耗尽时,产物不增长,位于平台期。
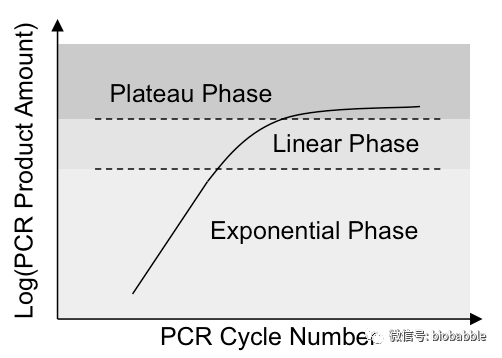
在我们的实验中,通常PCR反应是处于指数增长期。我们将其进行对数处理,指数增长就会变成线性增长,这样数据的右偏现象就会有明显减弱,当然我不敢说对数转换后,它就能呈正态分布,但起码对数处理后数据的分布比原始ratio数据更接近正态了。
T Test
只有偏态不明显的情况下,才可以用t检验进行分析。
#
t.test(log(ratio[,1], base=2), mu = 0)
#
#
#
#
#
#
#
#
#
#
#
#
t.test(log(ratio[,2], base=2), mu = 0)
#
#
#
#
#
#
#
#
#
#
#
Use −ΔΔC
t
directly
如果我们要使用T检验,就得做对数处理,细心的人应该会发现ratio的计算就是指数运算,ratio取对数之后,就是−ΔΔC
t
,所以我们可以直接使用它来做检验。
#
t.test(-dd.ct[,2])
#
#
#
#
#
#
#
#
#
#
#
ANOVA
另一种方法,使用ANOVA进行统计,这也是参数统计方法,在我们这种小样本的情况下,同样对数据分布有严格的要求,我们不能基于ratio做统计,要基于ΔC
t
值。
print(delta.ct)
#
#
#
#
#
#
#
#
#
summary(aov(gene01 ~ treatment, data=delta.ct))
#
#
#
summary(aov(gene02 ~ treatment, data=delta.ct))
#
#
#
#
#
ANOVA也是比较常用的,而且可以应用于多组实验和多个因素中。但是如果按照上面的代码来做,还不如用T test,因为实验是成对(tretated vs control)进行的,而这个信息在上面的ANOVA分析中,被扔了。
实际上 −ΔΔC
t
的单样本T检验,相当于ΔΔC
t
的双样本T检验。
t.test(gene02 ~ treatment, delta.ct, paired=T)
#
#
#
#
#
#
#
#
#
#
#
而这里用ANOVA,没有用到成对的信息,计算出来的p值和unpaired T test就基本相等。
t.test(gene01 ~ treatment, delta.ct, paired=F)
#
#
#
#
#
#
#
#
#
#
#
t.test(gene02 ~ treatment, delta.ct, paired=F)
#
#
#
#
#
#
#
#
#
#
#
做为成对T检验的扩展,使用方差分析进行多组实验的比较,需要用到的是重复测量方差分析(repeated-measures ANOVA)。
delta.ct[,1] = factor(delta.ct[,1])
delta.ct[,2] = factor(delta.ct[,2])
summary(aov(gene01 ~ treatment+Error(sample/treatment), data=delta.ct))
#
#
#
#
#
#
#
#
#
summary(aov(gene02 ~ treatment+Error(sample/treatment), data=delta.ct))
#
#
#
#
#
#
#
#
#
#
#
这样算出来的p值就和成对T检验几乎一样。
Wilcoxon Rank Sum Test
我们计算出来的ratio,是经过PCR扩增之后产物的比值,这是经过指数扩增的,而如果我们取对数,相当于消除了指数增长的效果,−ΔΔC
t
其实就是原始量的差值。
所以ratio的比较,相当于是扩增后的比较,而log
2
ratio 的比较,则相当于是扩增之前的比较。这两种数据当然都是可以拿来做统计分析的,只不过T test等参数检验方法对数据分布有要求,不适合于ratio值的统计分析而已。如果我们使用非参数检验,则不管是ratio值也好,log
2
ratio值也好,结果都是一样的。非参数检验并不使用数据的原始值,而是使用其排序值来做检验,我们并不能确定对数处理后的数据就呈正态分布,还有就是通常我们的实验重复次数是非常少的,在这种情况下,使用非参数检验,其实也是一种保守的、不容易犯错的方法。
#
wilcox.test( ratio[,2], mu=1)
#
#
#
#
#