
m6A RNA修饰是目前生物研究方面最热门和前沿的研究方向之一,也是CNS文章和国自然资助的热点。今天想和大家一起学习2021年3月发表在Frontiers in Cell and Developmental Biology(IF:5.201)上的文章。
雪球老师说“生信文章中,技术决定下限,思路决定上限”,那么,让我们一起从技术和思路两个方面来学习这篇文章吧。


题目
:
m6A Regulators Is Differently Expressed and Correlated With Immune Response of Esophageal Cancer
疾病
:
食管癌(Esophageal Cancer, EC)
分析策略
:
差异表达 + 临床意义 + 交互网络 + 功能聚类
m6A概念
:N6-methyladenosine也叫m6A,是指RNA的腺苷酸的第六位N处发生甲基化,主要调控RNA的转录、剪切、成熟、稳定、翻译等。
m6A调控因子
:m6A这种甲基化修饰是可逆化的,调控因子包括甲基化转移酶(writer)、甲基化阅读蛋白(reader)和去甲基化酶(eraser),具体见下表
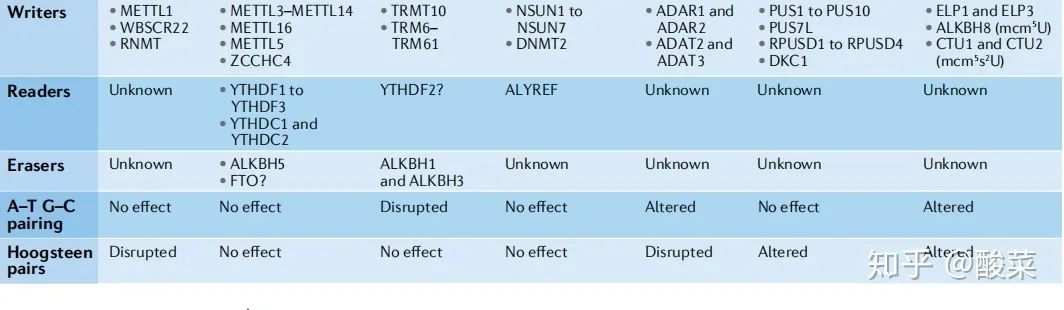
甲基化转移酶
(
writer
)
:能够让mRNA上的碱基发生m6A甲基化修饰。METTL3、METTL14、WTAP和KIAA1492都属于m6A甲基化转移酶的核心蛋白。这些蛋白会形成复合物(complex),共同行使催化功能。
去甲基化酶
(
eraser
)
:已经发生m6A修饰的碱基,在FTO和ALKBH这两种酶的作用下发生去甲基化。
甲基化识别蛋白
(
reader
)
:这些发生甲基化修饰的RNA碱基位点,需要特定的酶来识别,这时候就需要readers了。已知YTHDF家族包括YTHDF1、YTHDF2、YTHDF3以及Mrb1基因、Mmi1基因等都是readers类蛋白。这些酶能够识别发生m6A甲基化的碱基,发挥下游翻译、mRNA降解、加快mRNA出核速度等作用。
m6A的检测方法较为繁琐,所需费用也比较高,限制了m6A的研究进展以及临床应用,如果能从生物信息学挖掘出较为有价值的m6A及其调控基因的特征,能大大降低研究的成本和难度。
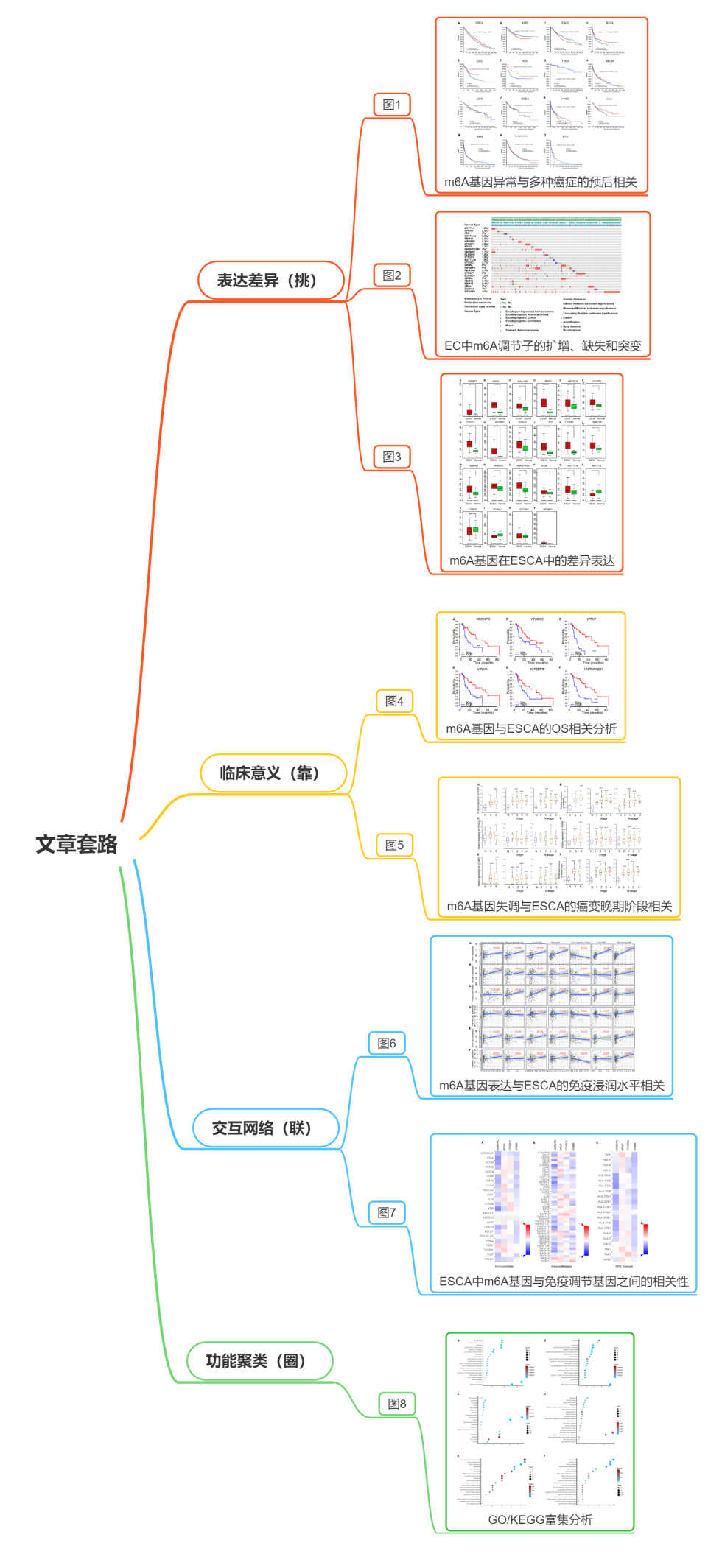
总体来看,本文一共有8张图,作者从TCGA数据库分析15种癌症中的23种m6A甲基化调控因子的突变与癌症患者生存时间之间的关系(图1),发现m6A调控因子的异常与乳腺癌、肾癌和食管癌(EC)患者的预后差显著相关。使用cBioPortal数据库检测了1,680例患者中m6A调控因子的遗传变异,发现大多数m6A调控因子在食管癌中扩增、缺失、突变,其中IGF2BP2的发生率最高(图2)。其中17种m6A调控因子在EC中表达增加,3种表达降低,3种无差异(图3)。Kaplan–Meier生存分析显示, 6种m6A调控因子与EC的较差愈后显著相关(图4)。与正常样品相比,EC中所有6种m6A调控因子(HNRNPC,YTHDC2,WTAP,VIRMA,IGF2BP3和HNRNPA2B1)均上调(图5)。在TIMER数据库中发现 m6A调控因子与EC中免疫细胞浸润水平的相关(图6)。为了进一步探讨m6A调控因子对肿瘤免疫反应的影响,计算了m6A调控因子和Immuno调控因子(免疫抑制剂,免疫刺激剂和MHC分子)的表达之间的相关性(图7)。并对6个m6A调控因子进行了GO和KEGG分析(图8)。
(https://www.xiantao.love/products)
(http://www.cbioportal.org/)
◆
UALCAN数据库
(http://ualcan.path.uab.edu/)
◆
TIMER数据库
(https://cistrome.shinyapps.io/timer/)
◆
TISIDB数据库
(http://cis.hku.hk/TISIDB/)
cBioPortal数据库显示EC中m6A调控因子的扩增、缺失和突变
以上2个图都是在
CBioPortal数据库
(http://www.cbioportal.org/)中完成。
进入CBioPortal数据库 → 【Esophagus/Stomach】 → 选择与EC相关的研究 → 【Query By Gene】
将23个m6A调控因子的基因名输入 → 【Submit Query】
得到的图片即为图2 → 【Download】 → 选择需要的格式下载
【Survival】 → 即可得到图1的EC survival → 选择需要的格式下载
图1的其他14种癌症的survival也用同样的方法,把所有图片拼接起来即可。
仙桃学术(https://www.xiantao.love/),点击【生信工具】
注:免费版和基础版都可以进行统计和可视化,由于高级版功能最全,这里选择高级版作为范例
【表达差异(挑)】 → 【表达差异】 → 【非配对样本】 → 【食管癌】 → 输入目的基因【IGF2BP3】 → 【确认】 → 可【保存结果】或【下载图片】
图3的其他22种基因也用同样的方法,把所有图片拼接起来即可。
【临床意义(靠)】 → 【预后分析】 → 【KM曲线图】 → 【食管癌】 → 输入目的基因【HNRNPC】 → 【确认】 → 可【保存结果】或【下载图片】
图4的其他5种基因也用同样的方法,把所有图片拼接起来即可。
进入UALCAN数据库(http://ualcan.path.uab.edu/) → 【TCGA analysis】
输入基因【HNRNPC】 → 选择【Esophageal carcinoma】 → 【Explore】
输入基因【HNRNPC】 → 选择【Esophageal carcinoma】 → 【Explore】
选择【Individual cancer stages】
选择【Nodal Metastasis status】,即为基因在不同N stage的分布
图5的其他5种基因也用同样的方法,把所有图片拼接起来即可。
进入TIMER数据库(https://cistrome.shinyapps.io/timer/) → 【Gene】
输入基因名【WTAP】 → 选择癌症类型【ESCA】 → 【Submit】 → 选择合适的格式下载即可
图6的其他5种基因也用同样的方法,把所有图片拼接起来即可。
进入TISIDB数据库(http://cis.hku.hk/TISIDB/) → 输入基因名【HNRNPC】 → 【Submit】
分别点击【Immunoinhibitor】或【Immunostimulator】或【MHC molecule】
例如【Immunoinhibitor】中,选择【ESCA】并截图
图7的其他3种基因也用同样的方法,把所有图片拼接起来即可。
原文作者是用DAVID数据库配合R语言来绘制的,但因DAVID数据库自2016年就未曾更新,所以我更推荐使用仙桃学术来绘制,但是结果与原文的略有差异
。
进入仙桃学术生信工具(https://www.xiantao.love/products/) → 【功能聚类(圈)】 → 【GO/KEGG】 → 【GO/KEGG富集分析】 → 将6个基因输入【分子list】 → 【确认】 → 【保存结果】并【表格下载】
小贴士:【保存结果】以便下一步可视化操作,【表格下载】可自行进行分析,这里两者都需要哦~
打开下载的GO/KEGG分析结果,筛选出pvalue<0.05的通路
进入仙桃学术生信工具 → 【功能聚类(圈)】 → 【GO/KEGG】 → 【GO/KEGG可视化】 → 选择刚刚保存的云端数据 → 选择【气泡图】 → 将从excel中复制的通路名粘贴到【ID List】 → 【确认】 → 【保存结果】或【表格下载】
图8的其他5种基因也用同样的方法,把所有图片拼接起来即可。
1.
从泛癌中筛选出与m6A相关的食管癌,进行后续分析,对于癌种的选择角度独特。
2. 结合m6A基因的突变数据和KM生存分析,从23种m6A调控因子中筛选出6种与食管癌相关的m6A调控因子进行后续分析,逻辑合理,论证紧凑。
3.结合了肿瘤免疫分析,使文章研究更深入。
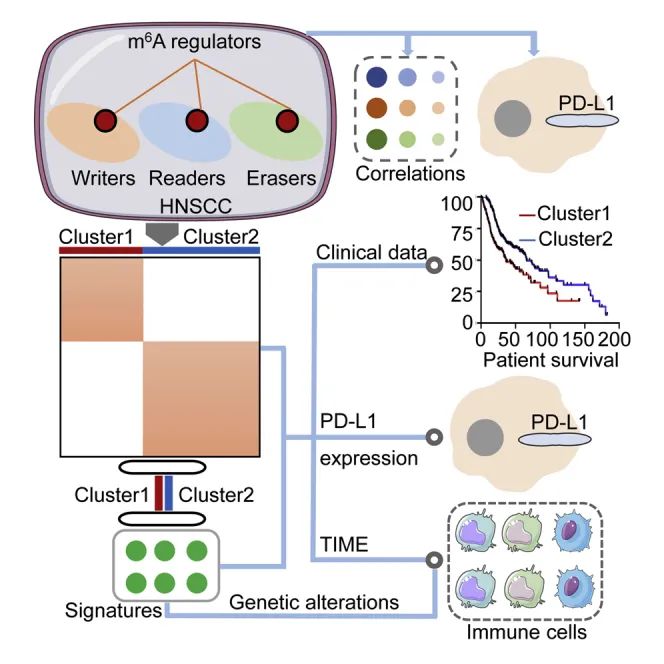
1.
可参考2020年发表于Mol Ther Nucleic Acids(IF:
7.032)上的“Comprehensive Analysis of the PD-L1 and Immune Infiltrates of m 6 A RNA Methylation Regulators in Head and Neck Squamous Cell Carcinoma”,对m6A调控因子进行聚类后,比较cluster之间的差异。
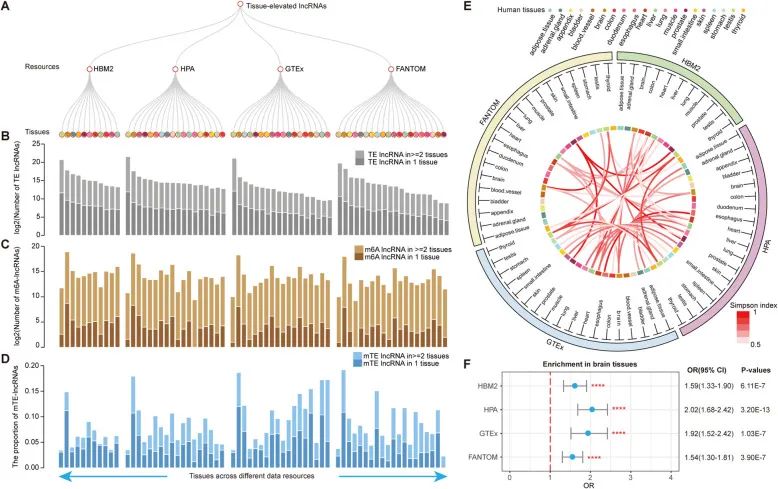
2.
可参考2
021年2月发表在Mol Cancer(IF:
15.302)上的“Pan-cancer characterization of expression and clinical relevance of m6A-related tissue-elevated long non-coding RNAs”对泛癌中m6A相关lncRNA进行分析。
3.
可利用m6A-Atalas数据库,www.xjtlu.edu.cn/biologicalsciences/atlas ,进一步挖掘m6A的表观基因信息。





