 点击蓝字“代谢组metabolome”,轻松关注不迷路
点击蓝字“代谢组metabolome”,轻松关注不迷路

生科云网址:
https://www.bioincloud.tech/
微科盟生科云 (https://www.bioincloud.tech)
旨在帮助客户
简化
分析流程,
助力
科研的基础上,不仅开发了
全自动化
的
一键式
批量
宏基因组
数据分析流程,还开发了
单个
分析
小程序
,可大大方便客户
灵活
分析。
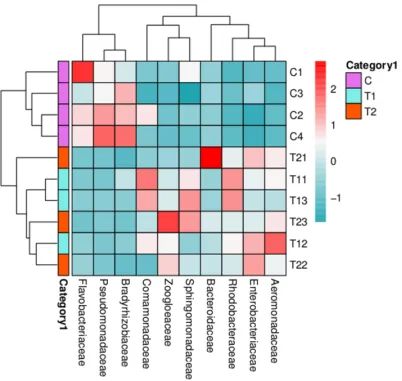
分组聚类热图
(又称“Heatmap 图”)是通过
颜色变化
与
相似程度
反映
二维矩阵
或表格中的
数据信息
。
它可以直观地将
数据值的大小
以定义的
颜色深浅
来表示,常根据需要将
数据
以样品间
丰度相似性
进行聚类,将聚类后数据表示在heatmap图上,可将
高丰度
和
低丰度
的物种、
基因
等分块聚集,通过
颜色梯度
及
相似程度
来反映
不同样品
或者
分组间
的
相似性
和
差异性
。
分析流程只需准备好
丰度表
和
分组信息表
两个输入文件,便可完成分组聚类热图的制作,更高效的挖掘基因的信息!
 结果展示
结果展示
接下来我们一起来看看我们的
视频
讲解吧!
微科盟生科云平台——分组聚类热图实操讲解
操作方法也可以看我们下面的
说明
哦~
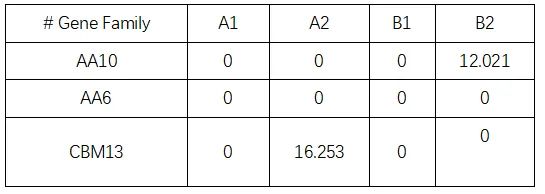
一、导入丰度表
制表符分隔的文本文件表格;第一行应包含
样本名
,唯一 (不可重复),与分组信息表第一列相对应 (顺序可以不一样);第一列为
组学feature名
(如微生物,代谢物,基因,表型名),唯一 (不可重复),推荐使用绝对丰度表,reads数表等,
总体数值过小(小于1)
可能导致
无法出图
,示例表格以功能丰度表为例:

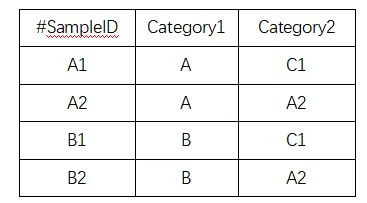
二、导入分组信息表
制表符分隔的文本文件表格;行名为
样本名
,列名为
分组方案
。
注:分组信息表中的
行名
(第一列) 必须和丰度表的
列名
相对应
,示例文件如下:
注:
1.输入文件不应该包含
特殊字符
,推荐使用字母数字下划线和点编辑表格;2. 输入文件行名 (第一列),和列名 (第一行)不能有
重复
的值;3. 表格中有
缺失值
的地方可以空着,但行名 (第一列) 和列名 (第一行) 以及最后一列不能有
缺失值
(不能空着),否则
无法
通过客户端检查。
三、选定分组方案
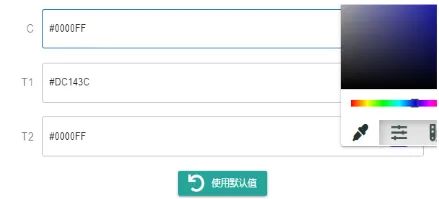
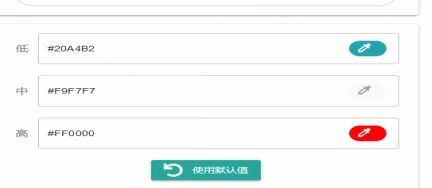
四、选定分组对应的颜色及格子颜色
可选择
默认值
,也可以按照
个人需要
进行调整
颜色
及
明亮
程度,示例如下:
分组对应颜色:
五、选定数值映射方式
数值映射方式有三种方式可选择,若选择log10转化,可
淡化巨大差异
,
维
持微小差异
,区分效果较好,例如输入绝对丰度表,其数值差异较大时可选择; 若选择
标准化
,可去除feature之间差异,突显样本间差异;若选择
无
:保留所有数据间差异。
六、是否使用分组均值绘制热图
若
不使用分组均值
绘制热图,则展示所有样本中组学features的组成情况;若
使用分组均值
绘制热图,则展示各样本组中组学features的组成情况;
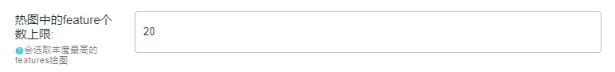
七、选定热图中feature个数上限
选择不同feature个数,可对丰度最高的feature画图,feature个数一般选择
默认值
即可,或按照
个人需要
进行调整,但超出上限以外的其余features丰度会合并为Others。
八、选择是否进行样本聚类
若选择
是
,结果图中样本会根据样本中组学features的丰度进行聚类;若选择
否
,结果图中样本顺序按照输入时顺序排列。
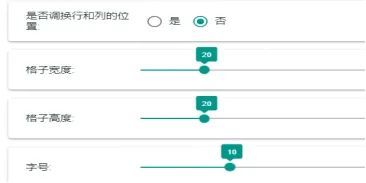
九、是否调换行列位置以及格子宽度、高度和字号大小
十、提交任务
点击
提交任务
,可选择任务完成时发邮件提醒,可避免等待运行时间,运行结束后可预览结果图,并下载结果文件。

 更多小工具和一键化程序上线
更多小工具和一键化程序上线
我们的云平台
持续升级
中,多个
组学
会陆续投入使用
,
请多多关注我们哦!
欢迎各位老师咨询使用方式,微科盟各组学老师联系方式如下:
注
:
已经与其他微科盟各组学老师是微信好友的则联系各自的老师,否则联系多组学老师8。
|
|
|
|
|
|

|
非其他微科盟各组学老师
好友的请添加多组学老师8为好友
|
|
|

|





