 点击蓝字“代谢组metabolome”,轻松关注不迷路
点击蓝字“代谢组metabolome”,轻松关注不迷路

生科云网址:
https://www.bioincloud.tech/
α多样性分析包括
稀释性曲线,α多样性指数计算及α多样性指数的组间显著性差异比较
。α多样性指数是对某个样品中物种多样性的分析,包含样品中的物种组成的
丰富度
和
均匀度
两个因素。比如Observed OTU指数是指样本中实际测定得到的OTU数量,衡量样品中OTU丰富度的指数;Shannon指数为多样性指数,它的计算考虑到样品中的分类总数,和每个分类所占的比例;Faith's Phylogenetic Diversity是基于系统发生树来计算的一种多样性指数;chao1指数是用来反映物种丰富度 (物种的种类数量) 的指标,它通过观测到的结果推算出一个理论的丰富度。
注:
在扩增子测序中,singleton通常是由于测序错误造成的,只会带来噪音,qiime2流程得到的OTU表通常会去除singleton,这意味着
某些根据singleton计算的指数将没有意义
,如ace,goods_coverage,chao1等。在得到整体的α多样性指数之后,结合分组信息来用Kruskal Wallis方法比较在各个样品分组之间α多样性指数是否有显著性差异。
β多样性分析是对
不同样品间的微生物群落构成进行比较,包括β多样性指数,β多样性组间显著性差异比较
。β多样性指数是基于Bray Curtis距离、Weighted Unifrac距离和Unweighted Unifrac距离来进行PCoA分析,并选取贡献率最大的主坐标组合进行作图展示,图中样品的距离越接近,表示样品的物种组成结构越相似。在得到整体的 β多样性指数之后,结合分组信息来用PERMANOVA方法和ANOSIM方法比较在各个样品分组之间的微生物组成结构是否有显著性差异。

赶紧打开我们下方的视频学起来吧!同时也可以登录我们
云平台链接免费注册
学习,详细的教程帮您轻松上手!



生科云平台在线网站(
https://www.bioincloud.tech
)
微科盟生科云平台——
Qiime2 α多样性
实操讲解
微科盟生科云平台——
Qiime2 β多样性
实操讲解
操作方法也可以看我们下面的说明奥
~
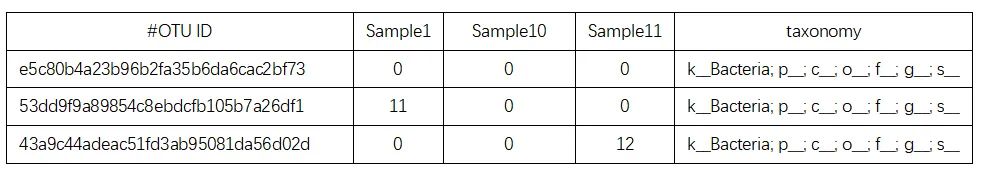
一、导入ASV (OTU) 丰度表
制表符分隔文本文件ASV (OTU) 丰度表的第一列应该为ASV或OTU的ID;中间列名应该是样本名;最后一列应必须为taxonomy(包含分类信息必须是普遍认同的七个分类水平,界门纲目科属种,分别以k__, p__,c__, o__, f__, g__, s__开头),示例表格如下:
二、导入ASV (OTU) 有根进化树
nwk格式的进化树文件以 “(” 开头,以 “;” 结尾;其中,有根进化树以 “root;” 结尾;进化树由ASV (OTU) 代表序列得到,可使用生信云的“构建进化树”工具生成,其流程如下:
① 导入同源序列文件:
文件格式必须是.fasta,.fa,.fna,.faa。第一行应为>开头的序列标识,唯一不可重复;第二行开始应为序列本身,可换行;接下来的序列也是如此,示例文件格式如下:

② 提交任务
点击提交任务,可选择任务完成时发邮件提醒,可避免等待运行时间,运行结束后下载结果文件查看。

③ 结果展示

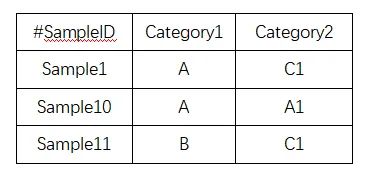
三、导入分组信息表
制表符分隔的文本文件表格;第一列为样本名,唯一 (不可重复),第二、三列为分组方案,其中,第一列的行名必须和丰度表的列名相对应,示例文件如下:
注:
1.输入文件不应该包含
特殊字符
,推荐使用字母数字下划线和点编辑表格;2. 输入文件行名 (第一列),和列名 (第一行) 不能有
重复
的值;3. 表格中有
缺失值
的地方可以空着,但行名 (第一列) 和列名 (第一行) 以及最后一列不能有
缺失值
(不能空着),否则
无法
通过客户端检查。
四、选定分组方案名
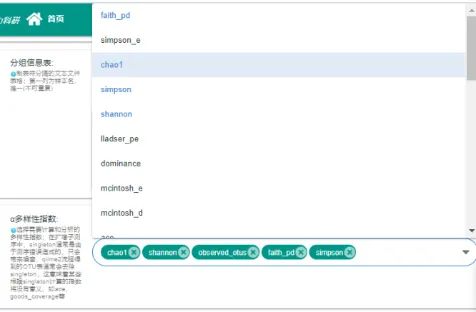
五、α多样性指数、抽样点个数和β多样性组间显著性差异比较方法
选择需要计算和分析的多样性指数,包括:chao1,shannon,observed_otus,faith_pd,simpson等,可选择任意几种进行分析,示例如下:
注:
在扩增子测序中,singleton通常是由于测序错误造成的,只会带来噪音,qiime2流程得到的OTU表通常会去除singleton,这意味着某些根据singleton计算的指数将没有意义,如ace,goods_coverage等。
选择不同的
抽样点个数
进行稀释性曲线的绘制,可选择默认值即可,示例如下:

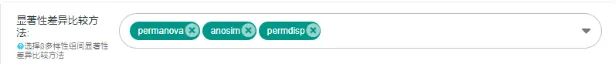
选择需要计算和分析的
β多样性组间显著性差异比较方法
,包括permanova,Anosim和permdisp,可随机选择任意一种或者几种进行分析,示例如下:
六、提交任务
点击提交任务,可选择任务完成时发邮件提醒,可避免等待运行时间,运行结束后可预览结果图,并下载结果文件查看。
 更多小工具和一键化程序上线
更多小工具和一键化程序上线
我们的云平台
持续升级
中,还会不断的加入更多组学的分析程序
,
请多多关注我们哦!
有如此详细的使用说明,您还会为作图烦恼吗?赶紧
点击
下方网址来我们云平台
免费
注册、
免费
体验,挖掘您想要的东西吧!



https://www.bioincloud.tech
欢迎各位老师咨询使用方式,微科盟各组学老师联系方式如下:
注
:
已经与其他微科盟各组学老师是微信好友的则联系各自的老师,否则联系组学老师47。
|
|
|
|
|
|

|
非其他微科盟各组学老师
好友的请添加组学老师47为好友
|
|
|

|
|
|
|

|
|

END

《代谢组metabolome》
是微科盟旗下专注于学术知识免费分享平台,内容主要包括
分享代谢组学最新文献,新闻,干货知识,视频课程和学术直播讲座
。
代谢组metabolome
仅用于学术成果分享与交流,不涉及商业利益。也严禁他人将本公众号的内容用于商业运营。





