生信草堂
将会与更多的优秀微信公众号合作,把最优秀的微信推文呈现给大家,希望可以帮助读者更多的了解生信技术,培养和提高读者的生信分析能力!
号外,号外,号外
你想和生信分析大神做好朋友么?
你想认识更多爱好生信分析的小伙伴么?
你想让自己的生信分析走上快车道么?
那就赶快加入我们的生信交流微信群吧!
正确加入我们的模式是:
添加我们的微信
Edison686868
或者
mly-1800
为好友
标注“
加入生信草堂交流群
”
在群里请大家注明自己
本名,单位,研究领域
便于小编管理
解释遗传关联分析结果的生物功能一直是一个很大的挑战,而这里要分享的文章则是一个成功的案例。通过
GWAS
发现的
SNP rs965513
作为
risk allele
对甲状腺癌易感性的机制。这篇文章的研究思路可以帮助我们思考遗传变异位点的潜在生物学机制。位于
enhancer
或者是非编码
RNA
中的变异可能很大程度上导致复杂疾病的遗传易感性。
乳头状甲状腺癌(
papillary thyroid carcinoma, PTC
)是最常见的甲状腺癌症。关联分析指出该疾病很大程度上受到遗传因素的影响。
GWAS
研究已经发现在
9q22
区域的单核苷酸多态性(
single nucleotide polymorphism, SNP
)
rs965513(
非编码
)
与
PTC
有关(
odds ratio~1.8
)
,
并且在不同人群中的到了独立重复验证。
SNP
(
rs965513
)位于
forkhead box E1 (
FOXE1
)
上游的
~60 kb
。
FOXE1
是一种已知的甲状腺转录因子,对甲状腺的功能,发育和分化至关重要,并且多次被报道与
PTC
的癌症发生有关。另一个功能性
SNP
(
rs1867277
)位于
FOXE1
启动子区域,也被报道与
FOXE1
转录调控有关。
最近,这个研究团队发现了一个甲状腺特异性的基因间长非编码
RNA
基因(
PTC susceptibility candidate 2 (
PTCSC2
)
。在人类基因组中,该基因位于
FOXE1
的反义链上:
PTCSC2
的
transcript isoform c
中的
exon 1
和
intron 1
与
FOXE1
启动子区域重叠,这说明
PTCSC2
和
FOXE1
可能共享同一个启动子或者转录调节机制。在甲状腺组织中,
PTCSC2
有剪切和未剪切两种转录本:剪切转录本有
11
个
exons
和一些不同的剪切亚型,而
SNP rs965513
正好位于未剪切转录本上(或者剪切的
intron
上)。另外,在
PTC
病人的正常甲状腺肿瘤,
rs965513
的有害基因型
[AA]
与
FOXE1
,
PTCSC2
,
和
TSHR
的表达降低显著相关。尽管,这些发现指出了
FOXE1
和
PTCSC2
与
PTC
的易感性和肿瘤发生的联系,但是
SNP rs965513
所产生效应的分子机制还是没有得到解释。
细胞中基因表达是受到
DNA
调节元件和序列特异性转录因子,以及染色质修饰的影响。因此,该团队推测在
rs965513
周围(
9q22
)存在
FOXE1
和
PTCSC2
转录的调节元件。于是,他们对
9q22
位点进行仔细分析和寻找导致
PTC
的功能性变异。最终,
他们根据研究结果暂时得出
9q22
诱发
PTC
是通过多种机制,而这些机制涉及到增强子,
TF
(
transcript factor
)结合位点,和两个及以上基因的转录变化。
接下来具体介绍该团队的研究过程:
重测序,
SNP
基因分型和
Imputation
,和
Haplotype
分析
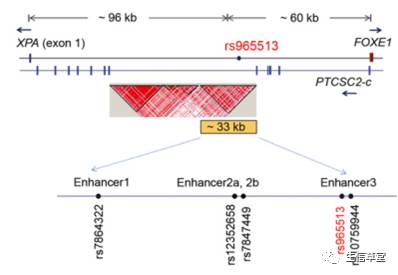
为了更好的找出
9q22
位点上的功能性
SNPs
和
PTC
相关的变异,该团队对
22
个
PTC
病人的染色体
9
号:
100455000–100622000
(
167 kb
)区域进行深度测序并进行
LD
分析。分析结果显示,在
PTC
病人和千人基因组计划
EUR
个体中,
GWAS SNP
周围都存在一个
33 kb
的
LD block
(图一)。
接着,为了在这
33 kb
区域中找出与
PTC
独立关联的
SNPs
,该团队在
1146 cases
和
1328 controls
中,进行
imputation
和
haplotype
分析。他们发现
33 kb
中,
只有
3
个
haplotypes
(
Hap1: OR=1.79
,
Hap2: OR=1.24
,
Hap3: OR=1.54
)包含
rs965513
的有害
allele[A].
另外,对
Hap1
的
diplotype
分析发现:杂合
Hap1
携带者的显示
OR=1.695
,而纯合显示
OR=3.423
。
总之,这些统计分析结果使他们把研究重点放到
~33kb
区域。
在甲状腺癌细胞系
KTC-1
和未感染甲状腺组织中,
9q22
富集了增强子组蛋白标记
考虑到
GWAS SNP
在
FOXE1
和
PTCSC2
中的位置,他们猜测
33kb
中的功能性
SNPs
是调节了重要的顺式转录调节元件,然后,利用公共数据库进行预测(
transcription factor (TRANSFAC) database
,
Encyclopedia of DNA Elements (ENCODE) ChIP sequencing data
,
和
HaploReg annotated information
),
他们发现有些区域包含了多个
TF
的结合位点(图二)。因此,他们选择了包含候选
SNPs
的
3
个区域,并根据它们的远程调控作用设计为
enhancer 1
,
enhancer 2
,和
enhancer 3
。
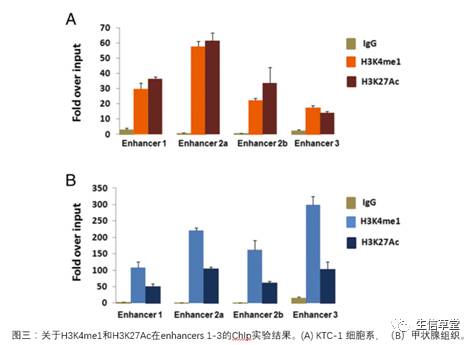
 接着,该团队就集中研究这三个
enhancers
。由于组蛋白是活跃型增强子标记,他们,在
KTC-1
细胞系和未感染的甲状腺组织中,对
H3K4me1
和
H3K27Ac
做了
ChIp
实验,并发现这两种组蛋白显著富集在
enhancer 1
,
enhancer 2a
,
enhancer 2b
,和
enhancer 3
(图三)。这一结果为
9q22
位点存在多种
vivo enhancers
提供了独立证据。
接着,该团队就集中研究这三个
enhancers
。由于组蛋白是活跃型增强子标记,他们,在
KTC-1
细胞系和未感染的甲状腺组织中,对
H3K4me1
和
H3K27Ac
做了
ChIp
实验,并发现这两种组蛋白显著富集在
enhancer 1
,
enhancer 2a
,
enhancer 2b
,和
enhancer 3
(图三)。这一结果为
9q22
位点存在多种
vivo enhancers
提供了独立证据。
3
个
enhancers
元件中增强子活性受到
SNP allele
的影响
。
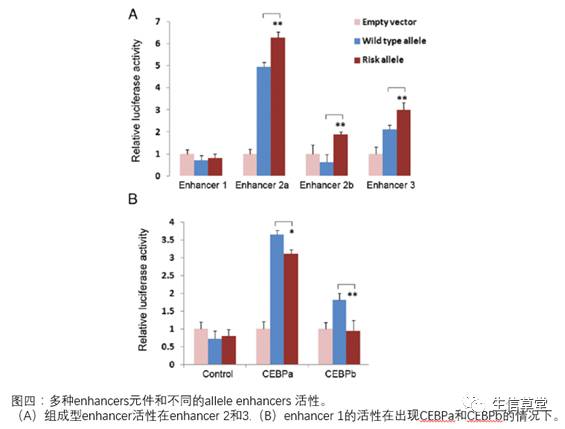
为了研究这
3
个
enhancers
的功能是不是受到
SNP allele
的影响,该团队进行了荧光素酶检测实验。他们对这
3
个
enhancers
的
risk allele
片段和
WT allele
片段分别进行扩增,并连接到荧光素酶报告载体上,然后,在
KTC-1
细胞系中进行检测。根据观察结果显示,
enhancer 2
中
rs12352658
的
[G] allele
和
rs7847449
的
[C] allele
,和
enhancer 3
中
rs10759944
的
[A] allele
都显示与
WT
相比显著增强荧光素酶的活性(图四)。由于
enhancer 1
没有观察到显著结果,于是,他们在做
enhancer 1
的荧光素酶实验时,同时转染了表达
CEBPa
或者
CEBPb
的质粒。结果,他们观察到,在出现
CEBPa
或者
CEBPb
时,
enhancer 1
中
rs7864322
的
risk allele [C]
显著降低荧光素酶的活性。
这部分实验结果指出,
在
9q22
位点中至少存在
3
个增强子元件并且它们的功能受到
allele
影响。总而言之这部分数据说明
GWAS SNP
附近有多种
allele
特异性的远程增强子元件(
long-range enhancer element
)。
针对
TFs
进行
ChIp
实验
为了进一步证明
3
个
enhancer
元件中的
SNPs
的功能,该团队利用
ChIp
实验技术,检测之前预测的
TFs
能否结合到
enhancers
上,和这种作用是否受到
allele
特异性的影响。于是,他们首先在
KTC-1
细胞系中检测
首先,他们在
KTC-1
细胞系中检测一系列预测发现的
TFs
,包括
CEBPa, CEBPb,STAT1, YY1, cJUN, ER,
和
TFAP2A
。
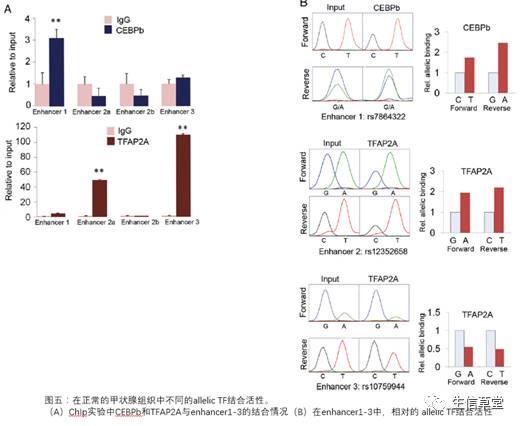
他们发现
CEBPa, CEBPb,
和
TFAP2A
有显著的结合作用。在正常的甲状腺组织中,
CEBPb
显著结合到
enhancer 1
上,但是没有结合到
enhancer 3
上(图五)。
TFAP2A
则显著结合到
enhancer 2a
和
3
上(图五)。接着,他们研究这些
TFs
与
enhancers
的相互作用是不是具有
allele
的特异性。在这里,他们用
SNaPshot
实验对
3