
本文来自作者
许玉龙
在
GitChat
上分享 「Java 操纵 Excel 文件数据实现复杂的项目需求」,
「
阅读原文
」
查看交流实录。
「
文末高能
」
编辑 | 哈比
一、明确功能需求
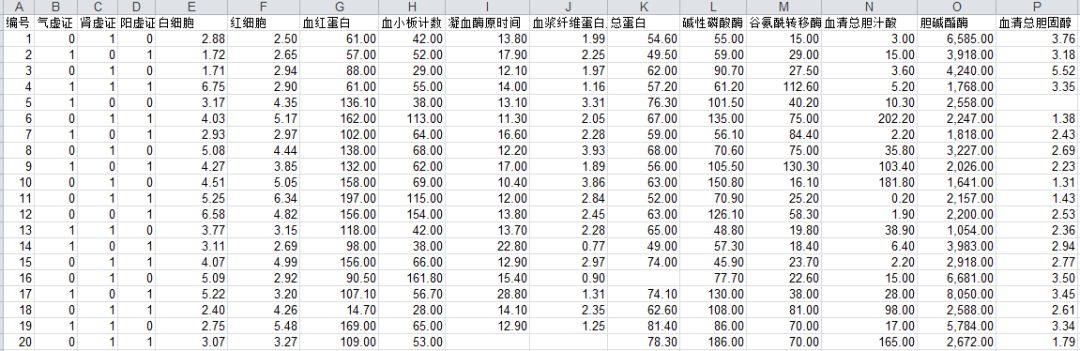
-
共有 3 个证候结果:气虚证、肾虚证、阳虚证,列序号分别为 1、2、3 列;值为 1 表示患者存在该证候,值为 0 表示不存在该证候,一个患者可以同时存在多个证候,证候起始和终止序号为 2-4。
-
共有 12 个西医指标:白细胞、红细胞、血红蛋白等,列序号分别为 4、5、6…,指标值为浮点数值。西医指标起始和终止序号为 5-16。
-
需求:
对所有数据,考虑每个证候,计算出存在和不存在该证候的两组数据,并对比该两组数据的均值、标准差、Ttest 检验、p 值,从而分析出西医指标对存在该证候的影响、相关性。
每个证候输出一个结果文件,文件中显示所有西医指标的计算结果值,按照 p 值从小到大排序,前几个指标即是对该证候影响较大的指标。
可使用任何语言解决,这里我们先介绍使用 java 解决,感兴趣的也可根据算法框架使用 python 实现,python 解决时代码更为简洁,也更简单。
二、设计算法
充分理解需求的情况下,设计算法框架。
要求遍历所有证候,每个证候单独输出一个文件; 在这个文件中 , 对所有西医指标进行考虑,即遍历所有西医指标;对于每个西医指标,要考虑所有行的记录(即遍历所有行), 分别计算证候为 0 和为 1 时的西医指标值。
通过上述分析,整体的算法框架需要三层循环 , 第一层为遍历每个证候 , 第二层为遍历每个西医指标 , 第三层为遍历每行记录,伪代码如下:
> For 证候 synIndex 1:6 // 假设有 6 个证候
> 创建证候文件 , 待输出数据
> For 西医指标 med 7:56 // 假设有 50 个西医指标
> 创建动态数组 , 存储指标值以及证候值
> For 每行记录 row
> 将西医指标值,根据证候值分别加入不同的动态数组
> End 每行记录 row
> 依据数组中的值 , 计算 p 值、t 值、均值等等 .
> 将该指标名称、p 值、t 值等信息作为一行,追加写入文件 .
> End 西医指标 med
> 关闭 csv 文件,考虑下一个证候
> End For 证候
三、设计细节
分组存储西医指标值,由于证候 0 和 1 的个数不确定 , 所以它们数组大小也不确定 , 需要使用动态数组分别存储证候 0 和 1 对应的西医指标值,可采用 vector 或 list 等数据结构来创建动态数组。
这里我们采用 vector 数据结构来创建动态 double 型数组,用于存储西医指标值。
读入及操作 xls 文件,采用导入 jxl 包调用函数读入 xls 文件数据,然后使用
Sheet readsheet = readwb.getSheet(0) 获取第一张 Sheet 表;
int rsColumns = readsheet.getColumns() 获取 Sheet 表中所包含的总列数;
int rsRows = readsheet.getRows() 获取 Sheet 表中所包含的总行数;
readsheet.getCell(j, i).getContents() 取得 j 列 i 行的元素值。并按照算法来操作数据,进行运算处理。
输出 csv 文件,采用 File csv = new File(OutFileName) 创建 CSV 文件,供写数据。
BufferedWriter bw = new BufferedWriter(new FileWriter(csv, false)) 写入新的数据行;需要导入 import java.io.BufferedWriter;计算 Ttest 分析的 t 值和 p 值。
采用函数 Ttest 如下 TTest myttest = new TTest();
t = myttest.t(OneSyndromeofMedArray, ZeroSyndromeofMedArray);
p = myttest.tTest(OneSyndromeofMedArray, ZeroSyndromeofMedArray);
其中,OneSyndromeofMedArray 是 double 类型数组,存储证候为 1 的西医指标值,ZeroSyndromeofMedArray 是 double 类型数组,存储证候为 0 的西医指标值。
需导入 import org.apache.commons.math3.stat.inference.TTest;
分别计算证候为 1 和 0 的对应均值和方差,需要导入 org.apache.commons.math3.stat.descriptive.moment.Mean;
import org.apache.commons.math3.stat.descriptive.moment.Variance;
然后声明 Mean mean = new Mean(); // 均值,Variance variance = new Variance();// 方差,计算结果值:
oneGroupMean = mean.evaluate(OneSyndromeofMedArray);
oneGroupStd = variance.evaluate(OneSyndromeofMedArray);
zeroGroupMean = mean.evaluate(ZeroSyndromeofMedArray);
zeroGroupStd = variance.evaluate(ZeroSyndromeofMedArray);
西医指标值存在为空情况,当空值的个数大于数据条数的一半时,不再进行计算处理,设特殊标记表示。
总体算法框架如下图所示:

四、编制代码
依据上述分析,按照算法框架,使用 java 编制代码,其中核心的实现代码如下:
public Boolean exeTtestCmd(){
jxl.Workbook readwb = null;
try
{
InputStream instream = new FileInputStream(filename);
readwb = Workbook.getWorkbook(instream);
Sheet readsheet = readwb.getSheet(0); // 获取第一张 Sheet 表 Sheet 的下标是从 0 开始
int rsColumns = readsheet.getColumns(); // 获取 Sheet 表中所包含的总列数
int rsRows = readsheet.getRows(); // 获取 Sheet 表中所包含的总行数
System.out.println(" 行数 " +rsRows + " 列数 " +rsColumns);
// Cell cell = readsheet.getCell(9, 13);
// System.out.println("9 列 13 行的值是 " +cell.getContents()); // 下标从 0 开始,转换到 excel 表格中是 10 列 14 行
for(int synIndex = synstart-1; synIndex < synend; synIndex++)//each syndrome
{
int medIndexNumber = 1;
String SyndromeName = readsheet.getCell(synIndex, 0).getContents();//output filename
String OutFileName = "F:/" + SyndromeName + ".csv";
File csv = new File(OutFileName); // 创建 CSV 文件,供写数据
BufferedWriter bw = new BufferedWriter(new FileWriter(csv, false)); // 附加 添加新的数据行
String nameoffirstrow = " 序号 "+ "," +" 西医指标 "+ "," + " 证候为 1 组均值 " + "," + " 证候为 1 组方差 "
+ "," + " 证候为 0 组均值 " + "," + " 证候为 0 组方差 " + ","+ "t 值 " + "," + " p 值 ";
bw.write(nameoffirstrow);
bw.newLine();
for(int medIndex = medstart-1; medIndex < medend; medIndex++){
int NoValueCountOfMed = 0;
double t, p, t2=0;
double oneGroupMean=0, oneGroupStd=0, zeroGroupMean=0, zeroGroupStd=0;
Vector ZeroSyndromeOfMed = new Vector ();
Vector OneSyndromeOfMed = new Vector ();
for (int row = 1; row < rsRows; row++){//for each line(row)
String tempMedIndex = readsheet.getCell(medIndex, row).getContents();
if((tempMedIndex!=null) && (tempMedIndex.length()>0)){//medicine value exist
if((readsheet.getCell(synIndex, row).getContents()).equals(String.valueOf(0))){//syndrome is 0
String tempvalue = readsheet.getCell(medIndex, row).getContents();
Double f;
if (tempvalue.length()>7){//length more than 7 will exist ",", such as "1,569.00"
String strDeleteComma = DeleteComma(tempvalue);
f = Double.parseDouble(strDeleteComma);
}else{
f = Double.parseDouble(tempvalue);
}
ZeroSyndromeOfMed.add(f);
}
else if((readsheet.getCell(synIndex, row).getContents()).equals(String.valueOf(1))){//syndrome is 1
String tempvalue = readsheet.getCell(medIndex, row).getContents();
Double f;
if (tempvalue.length()>7){//length more than 7 will exist ",", such as "1,569.00"
String strDeleteComma = DeleteComma(tempvalue);
f = Double.parseDouble(strDeleteComma);
}else{
f = Double.parseDouble(tempvalue);
}
OneSyndromeOfMed.add(f);
}
}else{//medicine value is not exist
NoValueCountOfMed++;
}
}//end for each line (row)
if (NoValueCountOfMed > 0.5*(rsRows-1)){ //Number of not exist medicine value more than half of rsRows
p = 100; //the more less the more significant. namely don't consider this medicine index
t = 0; // the abs(t) more bigger more significant. namely don't consider this medicine index
}else{// compute p and t according to ZeroSyndromeOfMed and OneSyndromeOfMed
int LenOne = OneSyndromeOfMed.size();
int LenZero = ZeroSyndromeOfMed.size();
double OneSyndromeofMedArray [] = new double [LenOne];
for(int i=0; i















