
原文以DNA's secret weapon against knots and tangles为标题
发布在2017年4月19日的《自然》新闻上
原文作者:Elie Dolgin
一个简单的过程似乎能解释庞大的基因组是如何保持有序的,但人们无法就这一过程由何驱动达成一致。
Leonid Mirny在办公椅上转了一圈,抓起自己笔记本电脑的电源线。他用手指绕出了一个甜甜圈大小的环,兴奋地坐立难安:“这就是马达蛋白不断挤压成环的动态过程!”Mirny说。他是美国麻省理工学院的一位生物物理学家。
Mirny兴奋的原因并不是将电脑配件收拾整齐。他讨论的问题是基因组的一个核心组织原则——约两米长的DNA是如何压缩到人体的几乎每个细胞中,而没有像去年的圣诞彩灯一样缠成一团乱麻的。
他认为,DNA不断穿过环状的马达蛋白,从而形成了环。这一过程被称为环挤压,它有助于将DNA局部区域维系在一起,与基因组的其它部分分开,甚至还协助了染色体形成一定的形状和结构。
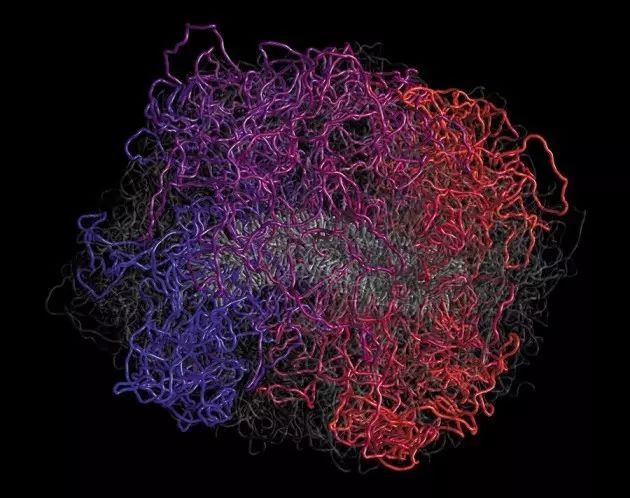
DNA环有助于将基因组局部区域维系在一起。
M. Imakaev/G. Fudenberg/N. Naumova/J. Dekker/L. Mirny
过去几十年来,科学家也探讨过相似的假说,但在基因组3D结构研究爆炸式发展的当代,Mirny的模型,以及美国贝勒医学院的遗传学家Erez LiebermanAiden提出的一个类似的模型,让这些假说在分子细节层面上升到了新的高度。这些模型巧妙地解释了一些知名研究项目的数据(这些项目的研究对象是基因组的各部分是如何在物理上相互作用的),并因此备受关注。
然而,这些简单的解释仍然存在争议。尽管
我们日益明确基因组成环会调控基因表达,并且可能与细胞发育和癌症等疾病相关,
但这些模型的预测已经超出了现有的实验观测结果的范围。
首先,成环的分子机制仍然是个谜。
如果主要的候选蛋白如同Mirny所预测的那样充当动力“马达”,那它就会以前所未见的速度消耗能量。“我的一个物理学家朋友告诉我,‘这就是你的领域中的希格斯玻色子’,”Mirny说;它能解释基因组生物学最深层的奥秘之一,但可能需要多年时间才能获得验证。
尽管Mirny的模型与Lieberman Aiden的模型极为相似(两者间的区别十分深奥难懂),但辨清谁才是正确的并不仅仅是个细节问题。如果Mirny是正确的,“这就将是DNA酶学界一场彻底的革命,”英国牛津大学的染色体前沿研究者Kim Nasmyth说。他补充说,
究竟是什么驱动了环的形成“是目前基因组生物学领域最大的难题”。

三十多年前,遗传学家就知道基因组可以形成环,使调控因子接近它们所控制的基因,但并不清楚这些环是如何形成的。
多年来,一些研究者分别提出了环挤压理论的不同版本。第一个提出类似观点的是美国希望之城贝克曼研究所的遗传学者Arthur Riggs,在一篇被忽视的1990年论文中,他率先提出被他称为“DNA成卷”的想法。但人们普遍认为首先提出这一概念的是Kim Nasmyth。
按照Nasmyth的说法,2000年的某一天,他在意大利阿尔卑斯山区登了一天的山后产生了这个想法。那时,他和他的同事刚刚发现环状的黏连蛋白,这种蛋白复合体的主要作用是在细胞分裂时帮助分离染色体拷贝。在摆弄自己的登山工具时,
Nasmyth突然意识到,染色体可能是主动穿过黏连蛋白、或者相关的复杂凝缩蛋白的,就像绳子绕过登山扣一样。
“这似乎解释了一切,”他说。
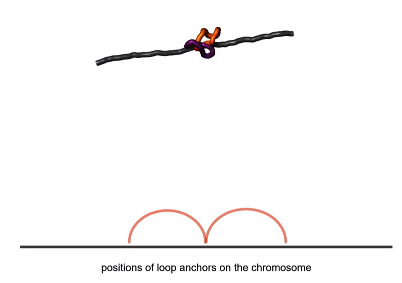
前期染色体通过环挤压进行压缩和分离:模型简示。
Mirny
Lab
在一篇长达73页的综述中,Nasmyth用几个段落描述了这个想法。“根本没人注意到它,”他说——就连美国西北大学的生物物理学家John Marko也没有对此提起注意——正是他在十多年之后建立起了与Nasmyth文字论证相补充的数学模型。
大约五年后,Mirny也加入了为DNA环建模的行列。他希望能解释生物学家Job Dekker编制的数据集。Dekker是他的一位长期合作者,就职于美国麻省大学医学院。Dekker一直在利用Hi-C技术寻找染色体不同位置间的物理相互作用,
在这一方法中,科学家测序相邻的小段DNA,生成每个染色体的图谱,通常以状如分形的“棋盘”形式:沿主对角线颜色最深的方块代表相互作用最密切的位置。
Dekker和他的合作者生成的Hi-C快照揭示出了明显呈不同区隔的环,其相互作用发生在20~100万碱基长的离散DNA片段间。
这些“拓扑关联结构域”
(TAD)
就像一列拥挤的火车上的车厢。人们能在同一节车厢中走动,接触其他乘客,但只有穿过车厢尽头的门才能与相邻车厢的乘客互动。人类基因组长达30亿核苷酸,但大多数相互作用发生在局部TAD区域内。
当时,Mirny和他的团队已经努力了一年多,试图利用计算机模拟来解释TAD的形成。随后,Mirny碰巧参加了一场学术会议,会上,Marko谈起了他当时还未发表的环挤压模型(Marko创造了这个术语,并沿用至今)。这正是Mirny想寻找的答案中的缺失一环。他们尝试了环挤压模型,发现它真的奏效。形成环的物理作用能让局部区域保持有序,模型也再现出了Hi-C图谱中的许多精细特征。
2015年8月,Mirny和他的同事在bioRxiv预印本平台上发布了他们完成的手稿,他们谨慎地使用了一个宽泛的术语,将该模型描述为“环挤压因子”。但文章并没有回避对其具体身份的猜测:分裂期细胞成环过程的驱动力是黏连蛋白,此时的染色体呈松散状。此后,他们在另一篇文章中提出,在染色体紧密缠绕的细胞分裂期间,凝缩蛋白发挥了同样的作用。
一个关键的线索是CTCF蛋白。
人们已经知道,它在未凝集染色体的每个环的基部与黏连蛋白相互作用。很久以来,研究者都认为环是在这些CTCF蛋白随机相遇并结合时在DNA上形成的。但如果任意两个CTCF蛋白都能配对,为什么这些环只形成于局部,而不会在相隔遥远的位点间形成?
Mirny的模型假设CTCF是黏连蛋白的一个终止标志。如果黏连蛋白只在正在形成的环的每一侧都遇到CTCF时才停止挤压DNA,蛋白就会自然而然地结合在一起。
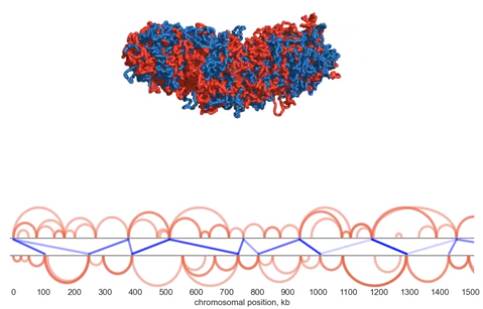
前期染色体通过环挤压进行压缩和分离:模拟黏连。
Mirny
Lab
然而,提出黏连蛋白发挥了驱动作用是“一个巨大的跨越”,生物物理学家Geoff Fudenberg说。他在Mirny的实验室完成了博士学业,现在就职于加州大学旧金山分校。
“
没有人曾在活细胞中,甚至体外观察到过这些马达蛋白发挥这样的作用,”他说。“但我们发现,利用这一原理,数据所呈现出的所有不同特征都能得到统一。”
举例来说,实验结果表明,细胞中黏连蛋白量的减少会导致形成的环减少。过度活跃的黏连蛋白则会产生大量的环,导致染色体被压成小蠕虫状的结构。
上述研究的作者在解释自己的结果时遇到了困难。然后,Mirny的文章在bioRxiv上发布了。“
有史以来第一次,一篇预印本真正改变了人们在这个领域思考问题的方式”
,英国医学研究委员会伦敦医学研究所的细胞生物学家Matthias Merkenschlager说。(Mirny团队的研究成果最终于2016年5月发表在了Cell Reports上。)

Lieberman Aiden说,他在2015年3月的一个电话会议中首次产生了环挤压的想法。在那时,他和自己前导师,美国博德研究所的遗传学家Eric Lander已经发表了当时分辨率最高、最为详细的Hi-C人类基因组图谱。
在电话会议中,Lieberman Aiden试图解释自己的数据中一个奇怪的现象。几乎所有锚定环的CTCF结合点都有着相同的方向。
他意识到,作为挤压的终止标志,CTCF具有固有的方向性。正如同司机不必理会交叉路口中与他们的前进方向不同的停车标志,环挤压因子也会一直通过CTCF位点,除非终止标志朝向的是正确的方向。





