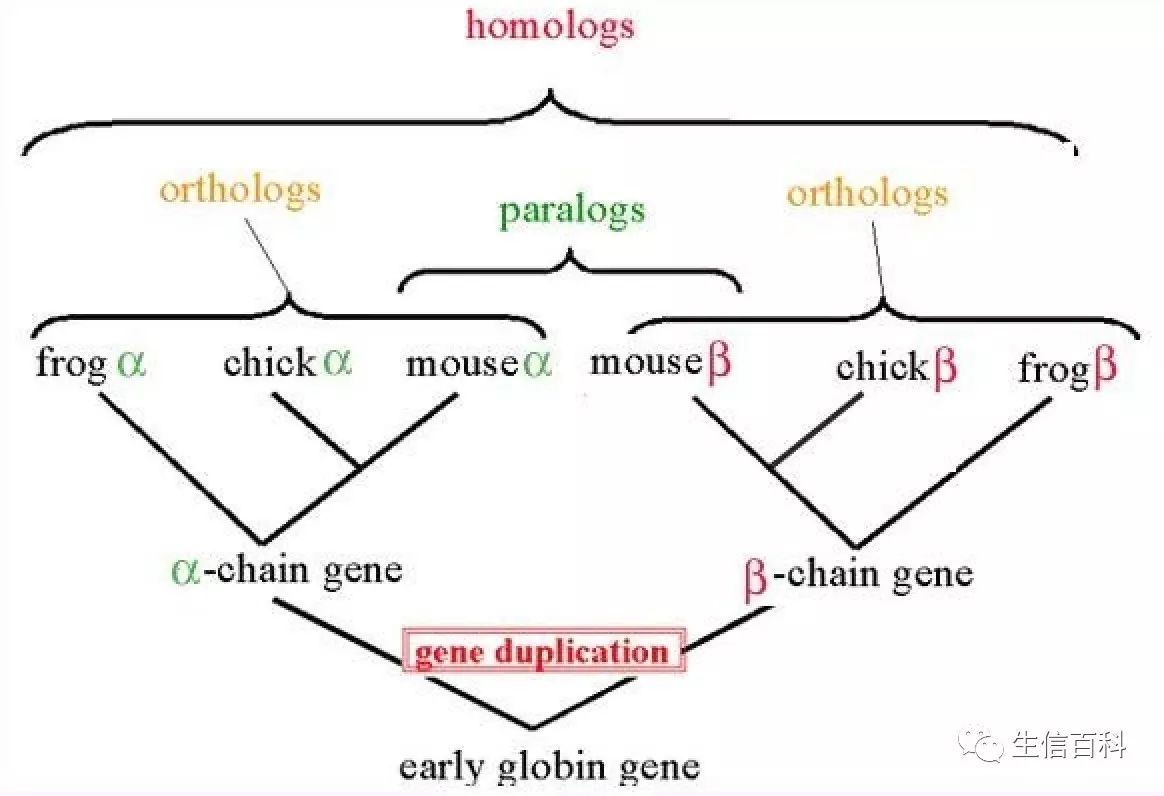
直系同源的基因通常具有相似的生物学功能,所以我们在进行一个未知序列的功能的鉴定的时候,本质上是在找功能已知的同源基因。对于两个或者多个物种的同源基因进行鉴定,是功能基因鉴定、比较基因组、功能基因分类、pathway预测等待的基础。
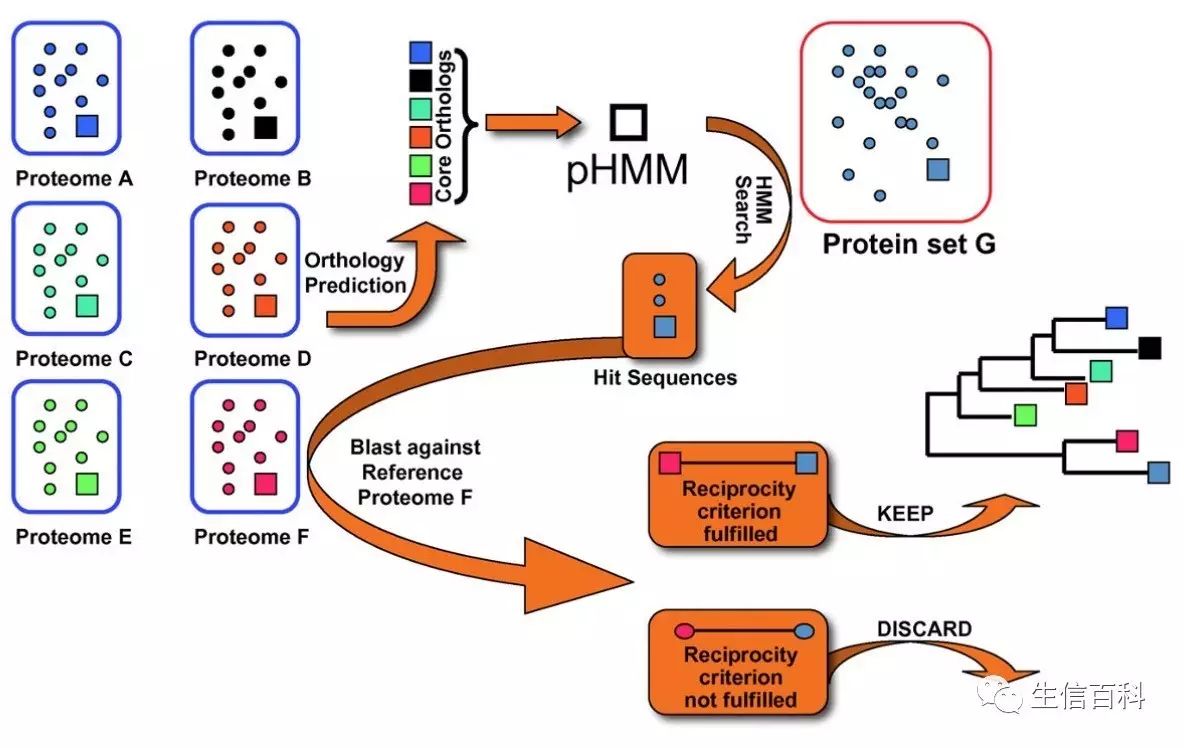
HaMStR是一款主要针对EST或者RNA-seq数据寻找直系同源基因的集成分析软件(网址:
http://www.deep-phylogeny.org/hamstr/
)。与Inparanoid和OrthoMCL等同样功能的软件相比,由于它自身集成了多种软件,HaMStR对数据的完整性要求不高,最后分析的结果更加全面可靠。HaMStR除了核心软件包以外,还提供了一个现有的模式植物的核心直系同源基因数据库,在大多数场景下使用非常有效。
在安装HaMStR之前,需要先安装一下四个集成的软件:
hmmsearch3:http://hmmer.janelia.org/
blastall:ftp://ftp.ncbi.nih.gov/blast/executables/release/
genewise:ftp://ftp.ebi.ac.uk/pub/software/unix/wise2/
clustalw2 :http://www.clustal.org/download/current/
这些软件安装过程相对简单,只需要按照说明操作来安装,并将每个软件的可执行程序加入到环境变量中。以blastall为例,下载解压后即可直接执行,执行如下命令就可以加入到环境变量中:(注意将BLASTHOME替换成blastall的实际目录)
export PATH=$PATH:BLASTHOME /bin
需要额外注意的一点是, genewise软件需要设置WISECONFIGDIR变量,可以用如下命令:(注意将$genewise2.2_HOME_PATH替换成实际目录)
export WISECONFIGDIR=$genewise2.2_HOME_PATH/wisecfg
HaMStR软件直接解压即可使用,它的可执行程序在bin目录下。
简要介绍HaMStR的使用。




