Ks可视化
我们在上一篇
如何用WGDI进行共线性分析(上)
得到共线性分析结果和Ks值输出结果的整合表格文件后,我们就可以绘制Ks点阵图和Ks频率分布图对共线性区的Ks进行探索性分析,从而确定可能的Ks峰值,用于后续分析。
Ks点阵图
最初绘制的点图信息量很大,基本上涵盖了历史上发生的加倍事件所产生的共线性区。我们能大致的判断片段是否存在复制以及复制了多少次,至于这些片段是否来自于同一次加倍事件则不太好确定。借助于Ks信息 (Ks值可以反应一定尺度内的演化时间),我们就可以较为容易地根据点阵图上共线性区域的颜色来区分多倍化事件。
首先,创建配置文件(这次是
-bk
模块,BlockKs)
wgdi -bk \? >> ath.conf
然后,修改配置文件
[blockks]
lens1 = ath.len
lens2 = ath.len
genome1_name = A. thaliana
genome2_name = A. thaliana
blockinfo = ath_block_information.csv
pvalue = 0.05
tandem = true
tandem_length = 200
markersize = 1
area = 0,3
block_length = 5
figsize = 8,8
savefig = ath.ks.dotplot.pdf
blockinfo 是前面共线性分析和Ks分析的整合结果,是绘图的基础。根据下面几个标准进行过滤
-
pvalue: 共线性区的显著性, 对应blockinfo中的pvalue列
-
tandem: 是否过滤由串联基因所形成的共线性区,即点阵图中对角线部分
-
tandem_length: 如果过滤,那么评估tandem的标准就是两个区块的物理距离
-
block_length: 一个共线区的最小基因对的数量,对应blockinfo中的length列
最后运行wgdi,输出图片。
wgdi -bk ath.conf
输出图片中的Ks的取值范围由参数area控制。图中的每个点都是各个共线性区内的基因对的Ks值。不难发现每个共线区内的Ks的颜色都差不多,意味着Ks值波动不大,基于这个现象,我们在判定Ks峰值的时候,采用
「共线性区的中位数」
就比其他方式要准确的多。在图中,我们大致能观察到2种颜色,绿色和蓝色,对应着两次比较近的加倍事件。
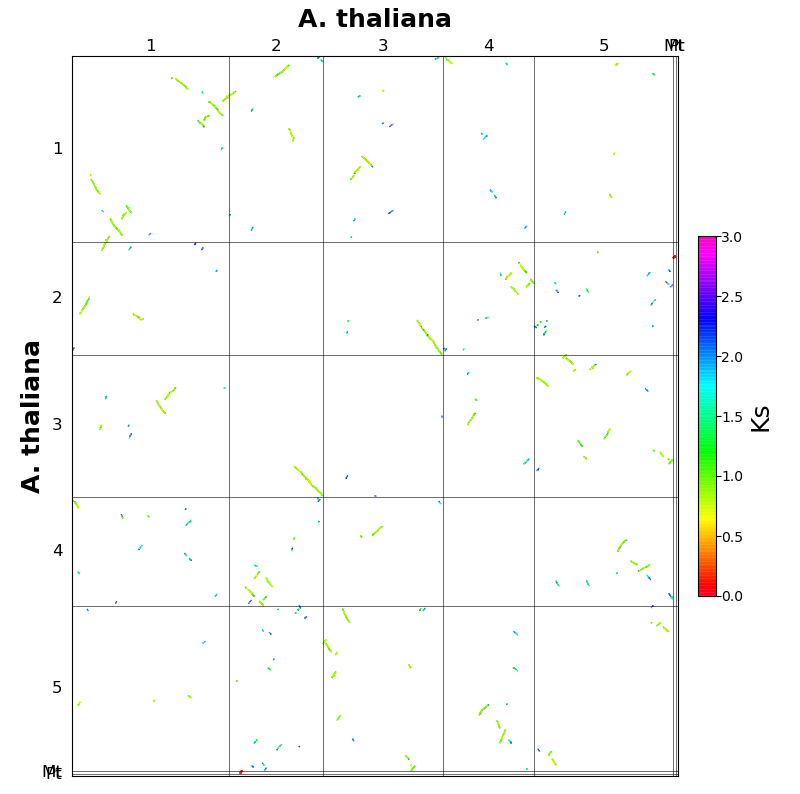 Ks点阵图
Ks点阵图
Ks频率分布图
除了用点阵图展示Ks外,我们还可以绘制Ks频率的分布情况。假如一个物种在历史上发生过多倍化,那么从那个时间点增加的基因经过相同时间的演化后,基因对之间的Ks值应该相差不多,即,归属于某个Ks区间的频率会明显高于其他区间。
首先,创建配置文件(-kp, ksPeak)





