DNA中的5-甲基胞嘧啶(5-methylcytosine, 5mC)是通DNA甲基转移酶在胞嘧啶环第5个碳原子上共价结合一个甲基产生的,广泛存在于不同组织中,在各种生物过程中发挥着重要作用。通过甲基化位点对相应的甲基化修饰进行研究是一种常用手段,因此,5mC位点的准确鉴定对深入理解其生物学功能至关重要。随着人工智能的飞速发展,深度学习已经成为了生物信息学的重要分析工具,越来越多的生物学问题通过深度学习得到解决。Transforme是一种基于注意力机制的深度学习模型,本研究基于第三代基因测序技术 Nanopore测序数据进行特征提取,通过Transformer编码器对特征进行编码,最后输入到双向长短期记忆网络(long short-term memory, LSTM)中以预测5mC位点。使用拟南芥(
Arabidopsis thaliana
)和水稻(
Oryza sativa
)对模型进行训练和测试,结果表明,本模型能够有效提取5mC位点的潜在特征,从而提高5mC位点的预测能力。

扫描下方二维码即可查看原文

亚热带农业生物资源保护与利用国家重点实验室、广西大学生命科学与技术学院
陈玲玲教授团队
在
《基因组学与应用生物学》
期刊第42卷第12期发表了题为“
基于Transformer编码器Nanopore数据的DNA 5-甲基胞嘧啶位点预测
”的研究论文。研究使用拟南芥和水稻的Nanopore测序数据对三种类型的5mC位点进行测试,其开发的Nanoformer能够有效提取5mC位点的潜在特征,在DNA 5mC位点预测中表现良好。该深度学习方法在未来可能进行更多物种和修饰类型的预测。
Nanopore测序中的电信号对DNA中的碱基修饰敏感,通过将甲基化5mC位点的原始电信号与已知序列的相同非甲基化位点的电信号进行对比,可以更高精度地测量特定基因组位置的DNA甲基化。目前,已有多种基于机器学习的算法使用Nanopore测序数据进行DNA甲基化检测。然而,之前的研究并没有充分利用到Nanaopore测序数据的序列和电信号特征,并且存在提取特征过程复杂、没有提取到位点的关键信息、模型预测功能不够强大等问题,通过改进这些方面有可能实现更好的预测性能。
本研究开发了一种从Nanopore reads中预测5mC所有类型甲基化位点的深度学习方法Nanoformer,为了尽可能保留5mC位点的原始信息,直接将Nanopore reads的原始电信号和DNA序列作为输入,通过Transformer编码器编码,然后使用Bilstm进行解码,最终输入到全连接神经网络中以预测甲基化状态。使用拟南芥和水稻基因组数据对Nanoformer进行了评估,最终的结果表明其在DNA 5mC位点预测中表现良好。
本研究构建了一个基于Transformer编码器和Bilstm的深度学习框架,命名为Nanoformer。如图1所示,模型的输入特征是每个motif位点的碱基序列和对应电信号结合而成的特征矩阵。输入维度是[batch,k_len,f_nums],其中,batch表示每一次输入样本的数量,k_len表示kmer长度,f_nums表示每个碱基的特征个数,默认为[256,41,12]。因为Transformer编码器只考虑了全局信息,不具备位置信息,所以在将数据输入到模型之前,需要在输入特征中加入位置编码(position encoding)。将进行位置编码后的数据输入到Transformer编码层中,Transformer编码层是由
n
个相同的块结构堆叠而成(本模型
n
=1),每个块结构又进一步分为两个子层。其中,第一层是一个多头自注意力层(muti-head attention),可以将输入信息映射到多个特征子空间,有助于模型捕捉来自不同子空间的特征关系,提取到不同信息之间更多的潜在关联;第二层是一个全连接前馈网络层(feed forward),由两个具有Relu激活函数的全连接层组成。为了防止梯度爆炸和消失,每一个子层之后都进行了残差连接(residual connection)和层归一化(layer normalization)。经过Transformer编码层后,再将编码后的数据输入到Bilstm中进行解码。Bilstm能够提取DNA序列两个方向的信息,并根据长短期记忆算法将正向输出与反向输出相结合以获得融合特征。最后经由两层全连接网络和一个softmax激活函数输出该位点为5mC位点的概率。
 图
1 Nanoformer
结构图
图
1 Nanoformer
结构图
Figure 1 The structure chart
of Nanoformer
本研究分别从拟南芥和水稻的Nanopor测序数据中随机选择了约9×和6×的Nanopore reads。表1为从水稻和拟南芥reads中提取到的基序位点数量信息。可以发现,在拟南芥和水稻中,高置信度非甲基化胞嘧啶的数量远远高于甲基化胞嘧啶的数量,尤其是在 CHG和CHH类型的基序中。对于每种类型的基序,随机选择出一百万条kmer作为模型 训练和测试的样本,不足一百万的按最大数量选择。在实验中,使用5折交叉验证来评估Nanoformer性能,采用6∶2∶2的比例来划分训练集、验证集和测试集。
本研究将Nanoformer和Deepsignal-plant进行了比较。Deepsignal-plant是目前为止最新发表的,也是表现最好的基于Nanopore数据检测基因组5mC位点的深度学习工具。为了比较的公平性,使用和训练Nanoformer相同的数据重新训练了Deepsignalplant,其准确率、精确率、召回率、F1值、AUC等5个性能指标对比如表2所示,Nanoformer在拟南芥和水稻的三种类型5mC位点预测中性能都要明显优于Deepsignal-plant的,其准确率、精准率、召回率、F1值、AUC在拟南芥中分别平均提升了4.53%、6.07%、3.35%、4.79%、2.78%,在水稻中分别平均提升了3.20%、4.15%、2.34%、3.26%、1.25%。
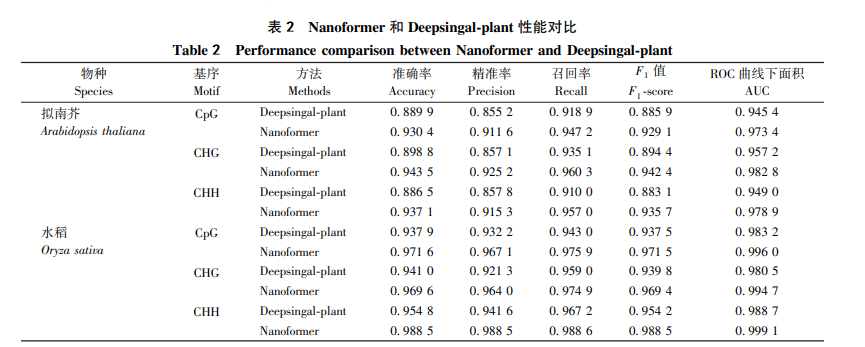
为了进一步证明Nanoformer的优势,分别使用9×的拟南芥和6×的水稻Nanopore reads在Nanoformer和Deepsingnal-plant上进行5mC甲基化位点预测,计算和BS-seq甲基化水平的皮尔逊相关性,结果如图2所示,Nanoformer在三种5mC甲基化位点预测的甲基化水平皮尔逊相关性 均要高于Deepsingnal-plant的,和表2的性能对比结果一致(
r
为皮尔逊相关系数)。
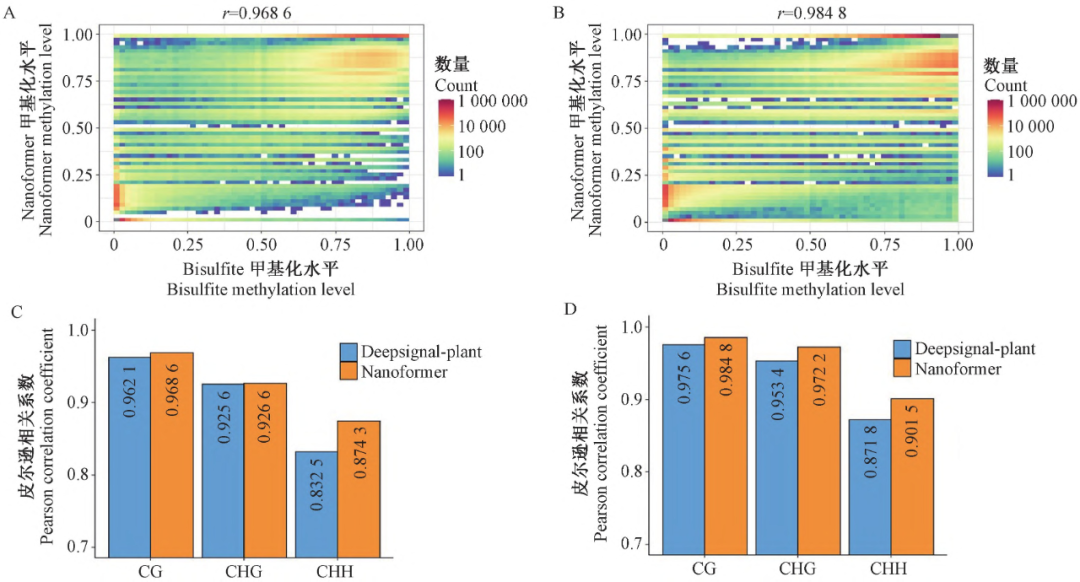
图
2
Nanoformer
与
Deepsignal-plant
预测
5mC
位点甲基化水平皮尔逊相关性对比
(
A)Nanoformer
预测拟南芥
CpG 5mC
位点
; ( B
)
Nanoformer
预测水稻
CpG
5mC
位点
; (
C)
拟南芥甲基化水平比较
; ( D)
水稻甲基化水平比较
Figure
2
Comparison of Pearson correlation between Nanoformer and Deepsignal-plant to predict the methylation level of 5mC sites
( A) Nanoformer prediction of
Arabidopsis thaliana
CpG 5mC sites; ( B) Nanoformer prediction of
Oryza sativa
CpG 5mC sites; ( C) Comparison of methylation levels in
Arabidopsis thaliana
;
( D) Comparison of methylation levels in
Oryza
sativa
为了进一步评估Nanoformer的跨物种性能,使用拟南芥和水稻数据对模型进行了交叉测试。测试结果如图3所示,三种类型5mC位点的AUC值在使用了不同物种的数据进行测试后均有一定程度的下降,不过下降程度有限。其中,使用水稻样本训练的模型来预测拟南芥5mC位点的AUC值均在0.939以上,使用拟南芥样本训练的模型来预测水稻5mC位点的AUC值均在0.976以上。这个结果说明Nanoformer具有一定跨物种检测5mC位点的能力。
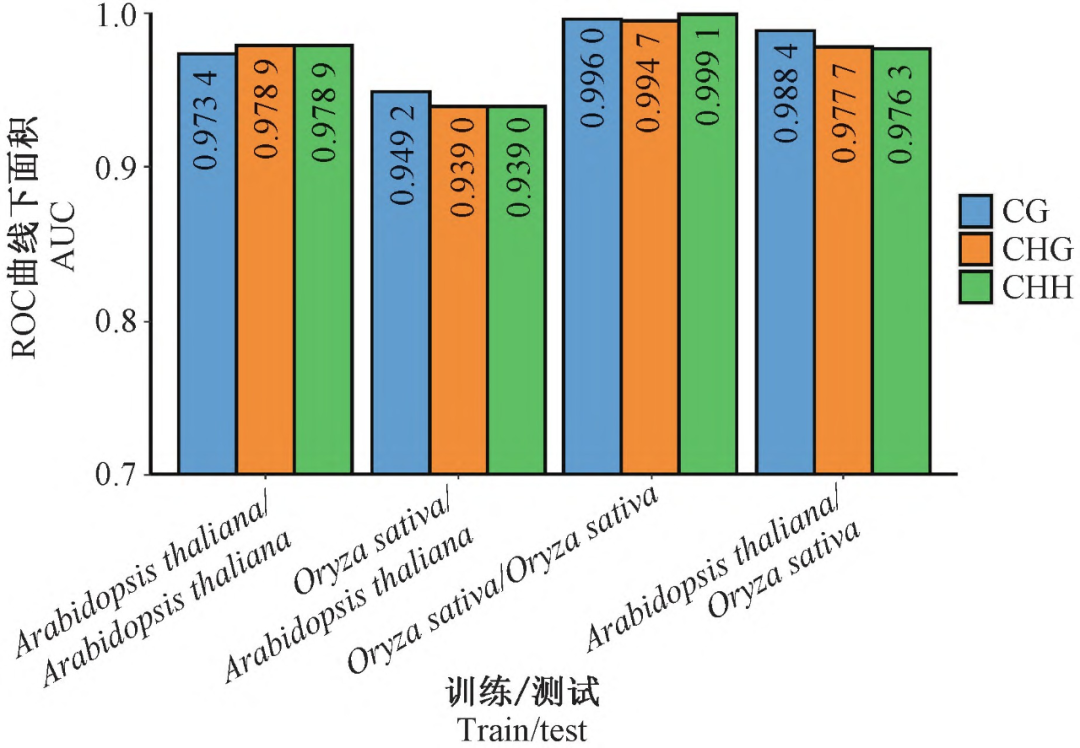
图
3 Nanoformer
跨物种性能测试
Figure 3 Cross-species performance testing of Nanoformer
DNA 5m甲基化是一种重要的表观遗传修饰方式,在许多生物的生长和发育过程中扮演着关键的角色。本研究提出了一种新的深度学习方法Nanoformer来进行DNA5mC位点预测。Nanoformer将Nanopore测序数据的原始序列和电信号结合后作为特征输入,这种融合特征相比于单独只用序列或者电信号来说,能够包含更多的5mC位点潜在信息,对模型性能有明显提升。此外,Nanoformer使用Transformer编码器进行特征编码,传统的循环神经网络在处理长序列时容易出现梯度消失或爆炸的问题,而Transformer编码器通过自注意力(self-attention)机制可以学习到序列中的长期依赖关系,从而提高序列预测的准确性。最后,Nanoformer使用Bilstm进行5mC位点的最终分类,Bilstm具有向前和向后两个方向的处理能力,这意味着它能够利用前后文的信息进行推理和预测,这对于包含5mC位点的DNA序列来说,使用Bilstm非常合适。
本研究使用了拟南芥和水稻的Nanopore测序数据对三种类型的5mC位点进行了测试,结果表明Nanoformer能够准确预测拟南芥和水稻中的5mC位点和非5mC位点,比现有的基于机器学习的方法取得了更好的性能。此外,对这两个物种的交叉验证表明,Nanoformer具有跨物种预测的能力。因此,Nanoformer可以用于全基因组表观遗传学分析,并在未来进行更多物种和修饰类型的预测。

陈玲玲,女,广西大学生命科学与技术学院教授,博导,亚热带农业生物资源保护与利用国家重点实验室副主任。国家万人计划科技创新领军人才,国务院政府特殊津贴获得者,广西自然科学基金创新研究团队负责人。主要从事生物信息学领域的研究工作,包括植物及微生物基因组多组学整合分析及蛋白质相互作用网络构建等研究方向。开发了植物CRISPR-P系列工具,是目前国际上通用的植物单链导向RNA设计工具。主导了多种农作物及园艺植物基因组解析,建立了作物及病原微生物代谢网络及蛋白互作网络,构建了多种植物多组学生物信息数据库。在















