我们有时候会有一个需求,就是从我们表达量矩阵里面挑一个单基因,来展现该基因在
两种或者多种不同处理下
的
时序性表达
。就像下面的图那样。
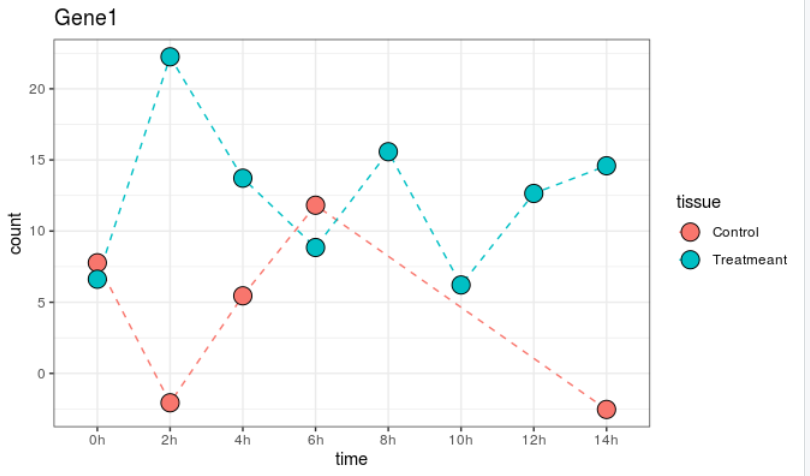
首先让我们构建下测试数据
# 测试数据
library(ggplot2)
library(tidyverse)
set.seed(1996)
test_data matrix(c(rnorm(5,mean = 5,sd = 5),rnorm(8,mean = 10,sd = 5)),
nrow = 1)
colnames(test_data) paste0(rep(c("Control_","Treatmeant_"),c(5,8)),
c(seq(0
,14,2)[c(1:4,8)],seq(0,14,2)),
"h")
rownames(test_data) "Gene1"
我这里只构建了一个Control下0,2,4,6,14h以及0,2,4,6,8,10,12,14h下的
一个基因
的表达矩阵。
> test_data
Control_0h Control_2h Control_4h Control_6h Control_14h Treatmeant_0h
Gene1 7.771877 -2.064757 5.451308 11.81829 -2.533594 6.618404
Treatmeant_2h Treatmeant_4h Treatmeant_6h Treatmeant_8h Treatmeant_10h
Gene1 22.24507 13.71656 8.846725 15.57643 6.213443
Treatmeant_12h Treatmeant_14h
Gene1
12.64404 14.58935
在得到这种数据之后,我们需要做下数据转换,把
宽数据
转换成
长数据
,才可以用ggplot2画图。
# 这里加上一列基因ID
test_data data.frame(test_data)
test_data$Gene rownames(test_data)
> test_data
Control_0h Control_2h Control_4h Control_6h Control_14h Treatmeant_0h Treatmeant_2h
Gene1 7.771877 -2.064757 5.451308 11.81829 -2.533594 6.618404 22.24507
Treatmeant_4h Treatmeant_6h Treatmeant_8h Treatmeant_10h Treatmeant_12h
Gene1 13.71656 8.846725 15.57643 6.213443 12.64404
Treatmeant_14h Gene
Gene1 14.58935 Gene1
# pivot_longer是最近的tidyverse套件里面宽数据转长数据的函数,当然你还是可以用gather
test_data_longer pivot_longer(data = test_data,cols = 1:(dim(test_data)[2]-1),
names_to = "Type",values_to = "count")
# 你也可以同样在cols后面写上"-Gene"
test_data_longer pivot_longer(data = test_data,cols = -Gene,
names_to = "Type",values_to = "count")
> test_data_longer
# A tibble: 13 x 3
Gene Type count
1 Gene1 Control_0h 7.77
2 Gene1 Control_2h -2.06
3 Gene1 Control_4h 5.45
4 Gene1 Control_6h 11.8
5 Gene1 Control_14h -2.53
6 Gene1 Treatmeant_0h 6.62
7 Gene1 Treatmeant_2h 22.2
8 Gene1 Treatmeant_4h 13.7
9 Gene1 Treatmeant_6h 8.85
10 Gene1 Treatmeant_8h 15.6
11 Gene1 Treatmeant_10h 6.21
12 Gene1
Treatmeant_12h 12.6
13 Gene1 Treatmeant_14h 14.6
但其实我们的Type里面包含了两个信息,即处理信息和时间信息,根据折线图的需求,我们到时候应该把颜色映射到处理类型,把X轴映射到时间上,
所以我们需要把Type拆成两列
。
# 用的是separate函数
plot_data test_data_longer %>%
separate(col = Type, sep = "_",into = c("tissue","time"))
# 这样就切割成了tissue和time两列了
> plot_data
# A tibble: 13 x 4
Gene tissue time count
1 Gene1 Control 0h 7.77
2 Gene1 Control 2h -2.06
3 Gene1 Control 4h 5.45
4 Gene1 Control 6h 11.8
5 Gene1 Control 14h -2.53
6 Gene1 Treatmeant 0h 6.62
7 Gene1 Treatmeant 2h 22.2
8 Gene1 Treatmeant 4h 13.7
9 Gene1 Treatmeant 6h 8.85
10 Gene1 Treatmeant 8h 15.6
11 Gene1 Treatmeant 10h 6.21
12
Gene1 Treatmeant 12h 12.6
13 Gene1 Treatmeant 14h 14.6
然后因为默认排序是并不是按照0,2,4,6,8,10,12,14h这样的,所以我们需要设置下因子顺序。
# str_sort可以帮助我们设置正确的数字顺序
> str_sort(unique(plot_data$time),numeric = T)
[1] "0h" "2h" "4h" "6h" "8h" "10h" "12h" "14h"
plot_data$time factor(plot_data$time, levels = str_sort(unique(plot_data$time),numeric = T))
你可以在这一步用subset或者filter挑选出你感兴趣的那个基因那部分数据,也可以在前面挑选。
然后就可以愉快地画图了。
ggplot(data = plot_data, aes(x = time, y = count,
group = tissue)) +
geom_line(aes(color = tissue),linetype = 2) +
geom_point(aes(fill = tissue),shape = 21,size = 5) +
theme_bw() +
ggtitle(label = unique(plot_data$Gene))

另附上批量画图
interest_gene_plot_list list()
for (i in interest_gene){
i_gene_data data[data$geneID %in% i,]
ggplot(data = plot_data, aes(x = time, y = count,
group = tissue)) +
geom_line(aes(color = tissue),linetype = 2) +
geom_point(aes(fill = tissue),shape = 21,size = 5) +
theme_bw() +
ggtitle(label = i) -> p
interest_gene_plot_list[[i]] p
}
pdf(XXX)
interest_gene_plot_list
dev.off()
这个批量画图是我临时造的,没搞测试数据……大家看看思路就行,反正很简单,就是for循环下,然后把图放list里面保存……





