磷是植物生长发育所必需的元素,其除了作为核酸和磷脂的关键成分外,还是信号通路传递和转录后调节的调节器
(Puga
et al.
, 2017, Ham
et al.
, 2018)
。植物吸收磷元素主要以无机磷的形式与土壤溶液一起被吸收,但是土壤中能够被植物吸收利用的可溶性磷有效率很低
(Holford, 1997)
。玉米作为我国第一大粮食作物,生物量高,需肥量大,缺磷势必会导致严重减产。因此通过遗传改良玉米磷吸收效率,培育并推广磷高效利用新品种,是解决这一问题的根本途径。虽然目前已经在植物中报道了一系列低磷响应基因,但对这些基因的研究维度还仅仅停留在一维和二维层面,为了得到更高分辨率的基因组构象及相应的调控关系,必须将相关性状的研究维度扩展到三维。
近日,四川农业大学玉米研究所
高世斌
教授团队在
The Plant Journal
发表了题为
“
Chromatin remodeling analysis reveals the RdDM pathway responds to low-phosphorus stress in maize
”
的研究论文,揭示了玉米低磷胁迫响应的新机制。

该课题组通过
Hi-C
测序结果发现,低磷条件下玉米染色质结构发生变化,并且全基因组甲基化测序初步证明
RdDM
水平与染色质结构变化密切相关。同时转录组和定量结果显示
RdDM
通路相关基因能够响应低磷胁迫。对
RdDM
途径的玉米突变体进行鉴定发现,
ddm1a
在低磷胁迫下能够通过缩短并加粗根系来增加其表层磷元素的吸收。关于玉米中的
ddm1
基因前人已有研究发现,玉米基因组中存在两个
DDM1
同源基因(
ZmDDM1A
和
ZmDDM1B
)
,
它们也分别被命名为
Chr101
和
Chr106
。
chr101
玉米突变体在叶片中
CHH
甲基化水平显著降低,
chr106
玉米突变体叶片中的
CHH
和
CHG
甲基化水平显著降低
(Li
et al.
, 2014)
。同时研究发现,
Zm
DDM1
对于
24-nt siRNA
的存在是必不可少的
(Long
et al.
, 2019)
。进一步的实验表明,
ZmDDM1
在体内与
RdDM
复合体相互作用,并且
ZmDDM1
靶向区域与开放性染色质区域密切相关,在很大程度上也与
ZmDDM1-ZmAGO4
结合的基因组位点重叠
(Long
et al.
, 2021)
。
因此,
ZmDDM1
作为
RdDM
途径的一个重要成员能够通过重塑染色质结构来响应低磷胁迫。
此外,通过结合
ChIP-seq
和
GWAS
分析显示
ZmDDM1A
能与磷和根发育相关基因互作(
ZmPT7
和
ZmSHR2-h
等),这也为后续继续扩展玉米低磷胁迫响应信号通路提供了非常有价值的信息。
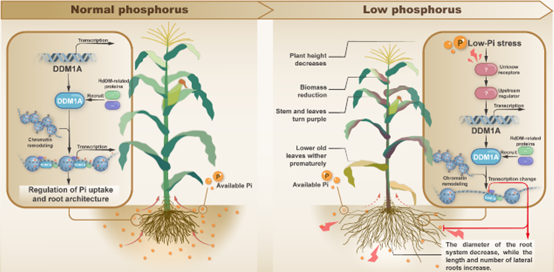
ZmDDM1A
参与玉米低磷胁迫响应的工作模型
四川农业大学玉米研究所青年教师
罗博文
博士和已毕业硕士生
张子琪
以及在读博士生
李滨洋
为论文共同第一作者
,高世斌
教授为通讯作者,
张啸
博士、绵阳农业科学研究院
马鹏
博士和瑞典农业科学大学
兰雨舟
博士等参与了研究工作。该研究得到了国家重点研发、国家自然科学基金、中国博士后基金、四川省科技支撑计划以及国家玉米产业体系等项目资助。同时感谢中国农业大学
贺岩
教授团队以及中国科学院分子植物科学卓越创新中心
龙金成
研究员提供的突变体材料和相关数据的分享,感谢赛乐基因提供的重测序数据处理加速服务(
BaseNumber DNA
测序数据分析系统)。
参考文献:
Ham, B.K., Chen, J., Yan, Y. and Lucas, W.J.
(2018) Insights into plant phosphate sensing and signaling.
Current opinion in biotechnology
,
49
, 1-9.
Holford, I.C.R.
(1997) Soil phosphorus: its measurement, and its uptake by plants.
Soil Research
,
35
, 227-240.
Li, Q., Eichten, S.R., Hermanson, P.J., Zaunbrecher, V.M., Song, J., Wendt, J., Rosenbaum, H., Madzima, T.F., Sloan, A.E., Huang, J., Burgess, D.L., Richmond, T.A., McGinnis, K.M., Meeley, R.B., Danilevskaya, O.N., Vaughn, M.W., Kaeppler, S.M., Jeddeloh, J.A. and Springer, N.M.
(2014) Genetic perturbation of the maize methylome.
Plant Cell
,
26
, 4602-4616.
Long, J., Liu, J., Xia, A., Springer, N.M. and He, Y.
(2021) Maize decrease in DNA methylation 1 targets RNA-directed DNA methylation on active chromatin.
Plant Cell
,
33
, 2183-2196.
Long, J.C., Xia, A.A., Liu, J.H., Jing, J.L., Wang, Y.Z., Qi, C.Y. and He, Y.
(2019) Decrease in DNA methylation 1 (DDM1) is required for the formation of (m) CHH islands in maize.
Journal of Integrative Plant Biology
,
61
, 749-764.
Puga, M.I., Rojas-Triana, M., de Lorenzo, L., Leyva, A., Rubio, V. and Paz-Ares, J.
(2017) Novel signals in the regulation of Pi starvation responses in plants: facts and promises.
Current opinion in plant biology
,
39
, 40-49.





