PRODIGY是一个
Haddock组
做的预测蛋白亲和力值的脚本,其同时也有在线服务,服务网址为:http://milou.science.uu.nl/services/PRODIGY/
该文章为2015年发表在Elife上,标题为
Contacts-based prediction of binding affinity in protein-protein complexes.
具体的详细算法可查看该篇文章。
在线介绍视频可以在百度网盘查看(英文):https://pan.baidu.com/s/1dFF0n9f
在线使用
我们可以看到在线界面非常简单,可选择的内容很少
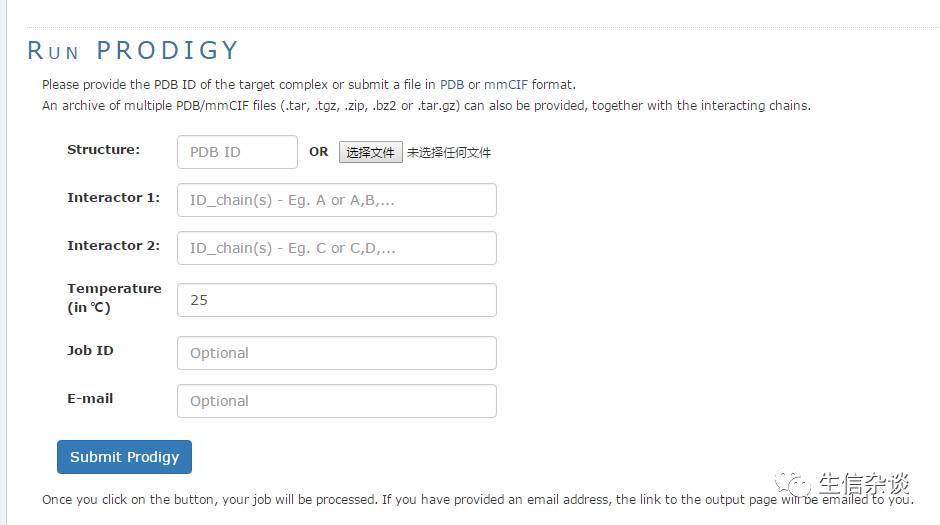
第一列为蛋白蛋白复合物的结构,可以上传
PDB
格式或者
mmCIF
格式,或者直接上传
PDB ID
,若有多个结构的话可以打包成压缩文件(.tar, .tgzi, .zip, .bz2 or .tar.gz)一次性上传。
Interactor
主要是就是输入相互作用的列啦,这个比较好理解,后面就是预测的相互作用温度,默认25度。
可能相较于设置可能大家更加关注于输出文件提供的内容,主要提供如下输出内容:
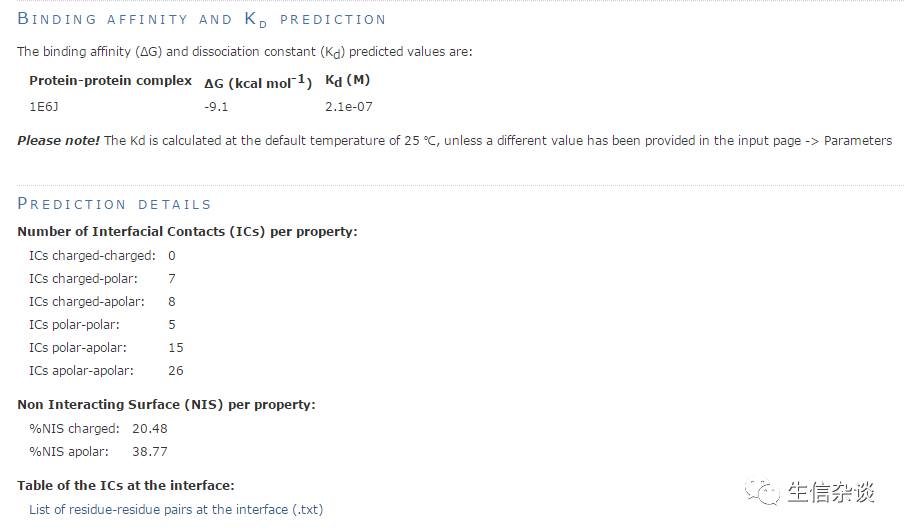
1.预测亲和力的值(ΔG)单位为kcal/mol
2.计算解离常数Kd,单位为摩尔M
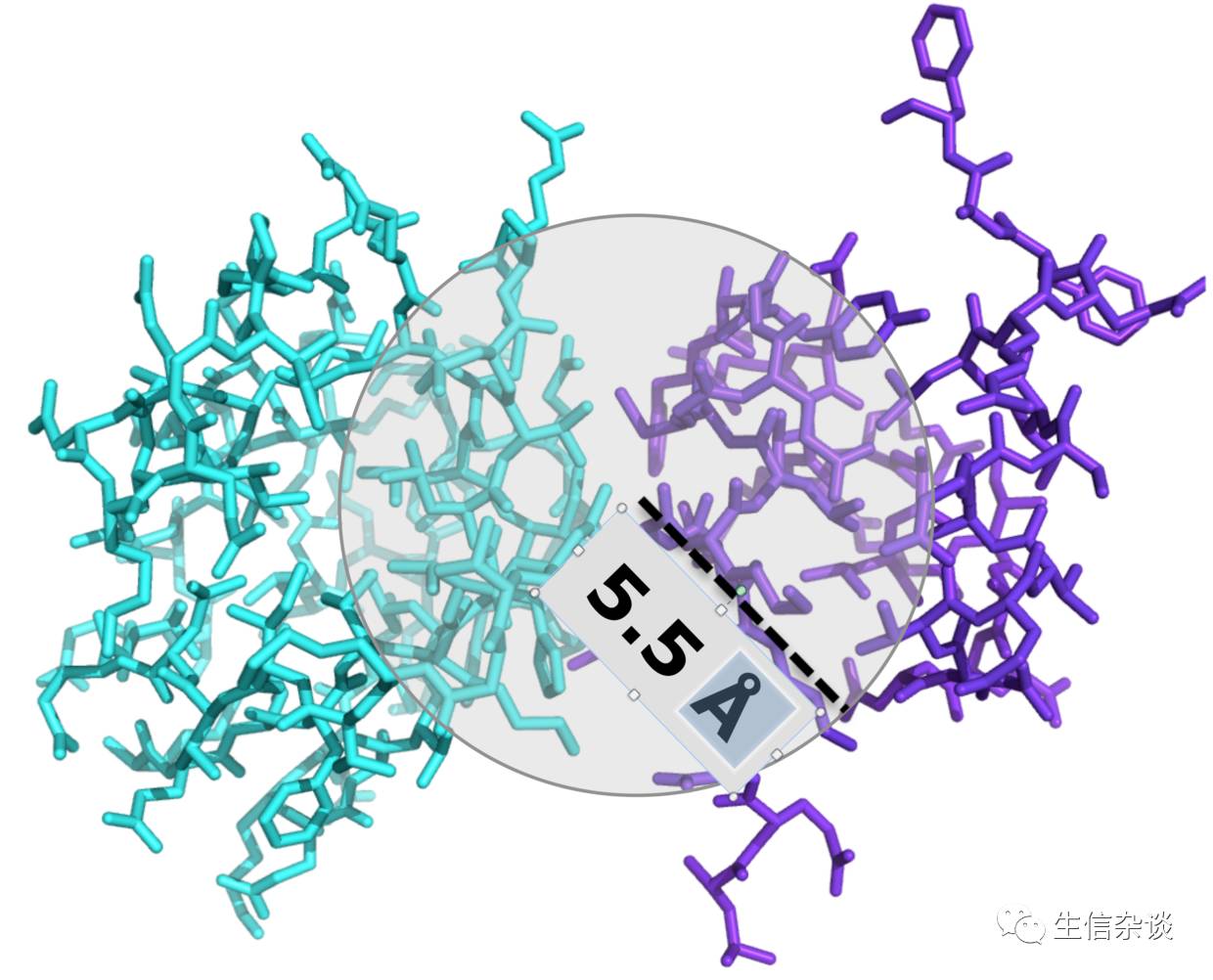
3.分子内的联系数量(ICs),阈值为5.5埃
4.复合物相互作用表面(NIS%)的电荷与极性百分比
5.txt的联系残基表以及Pymol显示文件
具体例子如下:
可以看一下其给的数据集与预测值的分析:
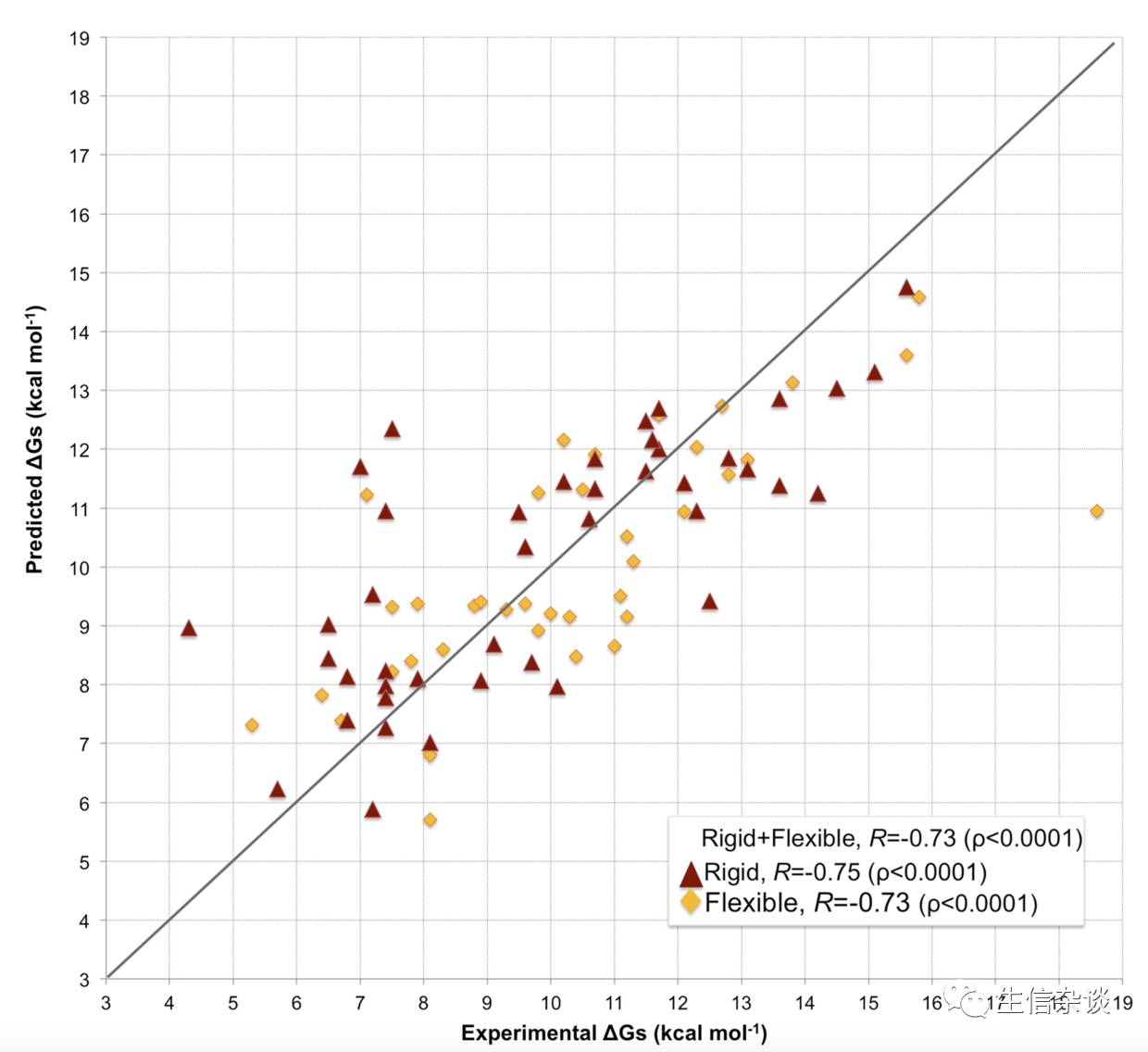
相关性达到了0.73,表现还是挺不错的,但是其并没有做无法结合的
两个蛋白之间的亲和力预测,是否有筛选作用
,个人猜测可能效果不理想。
表面联系的Pymol示意图:
若你想自己本地进行计算,可以下载安装
快速安装
git clone http://github.com/biopython/biopython.gitcd biopythonsudo python setup.py install
wget http://freesasa.github.io/freesasa-1.0.tar.gz
tar -xzvf freesasa-1.0.tar.gzcd freesasa-1.0./configure && make && make install
git clone http://github.com/haddocking/binding_affinity
使用
python predict_IC.py [--selection ]
可以输入
--help
的附加选项来查看可用的设置
值得注意的是该包的运行依赖于Biopython进行PDB结构验证以及计算原子间距离,依赖于freesasa进行BSA(buried surface area)计算。所以我们需要安装这两个包才可以使用,前面的安装中已经包含了这两个包的安装,当然也可以按照下面的方法分别安装
biopython快速安装
我们可以使用anconda进行安装,例如:
conda install biopython
FreeSASA快速安装
wget http://freesasa.github.io/freesasa-1.0.tar.gz
tar -xzvf freesasa-1.0.tar.gzcd freesasa-1.0./configure && make && make install
如果使用这些内容,记得引用如下文章:
Xue L, Rodrigues J, Kastritis P, Bonvin A.M.J.J, Vangone A.: PRODIGY: a web server for predicting the binding affinity of protein-protein complexes. Bioinformatics (2016)
Anna Vangone and Alexandre M.J.J. Bonvin: Contacts-based prediction of binding affinity in protein-protein complexes. eLife, e07454 (2015)
Panagiotis L. Kastritis , João P.G.L.M. Rodrigues, Gert E. Folkers, Rolf Boelens, Alexandre M.J.J. Bonvin: Proteins Feel More Than They See: Fine-Tuning of Binding Affinity by Properties of the Non-Interacting Surface. Journal of Molecular Biology, 14, 2632–2652 (2014).
更多原创精彩内容敬请关注
生信杂谈
:






