正文
火山图
火山图用于展示基因表达差异的分布,横轴为Log2 Fold Change,越偏离中心差异倍数越大;纵轴为(-1)*Log10 P_adjust,值越大差异越显著。一般横轴越偏离中心的点其纵轴值也会比较大,因此呈现火山喷发的形状。
一步绘制火山图
输入数据格式
火山图需要的数据格式如下 (本文用到的数据文件名为volcano.txt,文末有下载链接,此处截取一部分作为例子,也可用来画图,只是数据少,效果不明显)
id: 不是必须的,但一般的软件输出结果中都会包含,表示基因名字。
log2FoldChange: 差异倍数的对数,一般的差异分析输出结果中也会给出对数处理的值, 因此程序没有提供这一步的计算操作。
padj: 多重假设检验矫正过的差异显著性P值;一般的差异分析输出结果为原始值,程序提供一个参数对其求取负对数。
significant: 可选列,标记哪些基因是上调、下调、无差异;若无此列或未在参数中指定此列,默认程序会根据padj列和log2FoldChange列根据给定的阈值自动计算差异基因,并作出不同颜色的标记。
label: 可选列,一般用于在图中标记出感兴趣的基因的名字。非-行的字符串都会标记在图上。
id log2FoldChange padj significant label
E00007 4.28238 0 EHBIO_UP A
E00008 -1.1036 0.476466843393901 Unchanged -
E00009 -0.274368 1 Unchanged -
E00010 4.62347 7.37606076333335e-103 EHBIO_UP -
E00012 0.973987 0.482982440163204 Unchanged -
E00017 -1.30205 0.000555693857439792 Baodian_UP B
E00024 0.617636 2.78047837287061e-13 Unchanged -
E00033 1.48669 2.56000581595275e-60 EHBIO_UP -
E00034 -0.783716 0.00341521725291801 Unchanged -
E00036 2.01592 6.03136656016401e-06 EHBIO_UP C
E00040 -1.89657 4.73663890849056e-21 Baodian_UP -
E00041 -0.268168 0.563429434558031 Unchanged -
E00042 0.0861048 0.367700939634328 Unchanged -
E00043 -1.19328 1.42673872027352e-153 Baodian_UP -
E00044 -0.887981 2.43067804654905e-26 Unchanged -
E00047 -0.610941 5.51696648645932e-57 Unchanged -
使用significant列绘制火山图
sp_volcano.sh -f volcano.txt -x log2FoldChange -y padj -s significant -S "'EHBIO_UP', 'Baodian_UP', 'Unchanged'" -P TRUE -L top
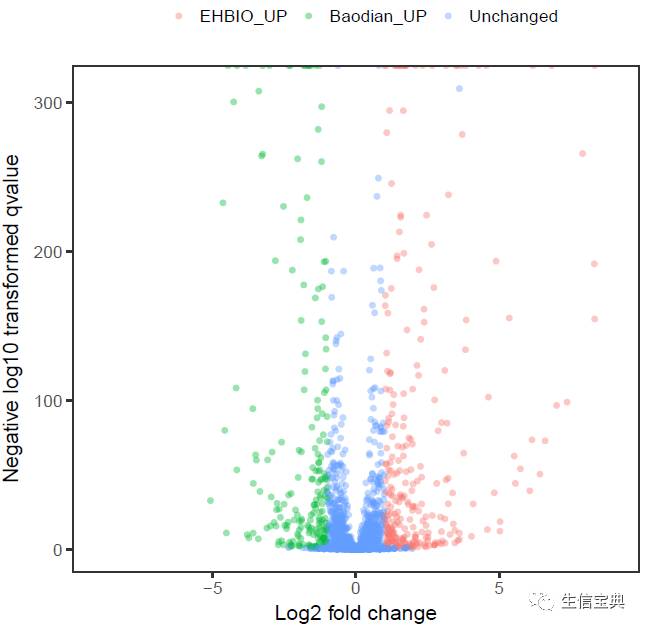
这个图看上去还可以,没有太大的问题。但有部分点与最顶端的线重合了,这些点的pvalue为0,取负对数后为负无穷。另外在一些情况下,会存在部分基因的pvalue极小,使得整张图呈现一个压缩的趋势,大部分点偏安于图的下方,中间大段空白,最上面零星几个点。为了避免这种情况,程序设置了参数-M用于设定pvalue的最大的负对数,所有大于给定值的数,都会视为给定值。
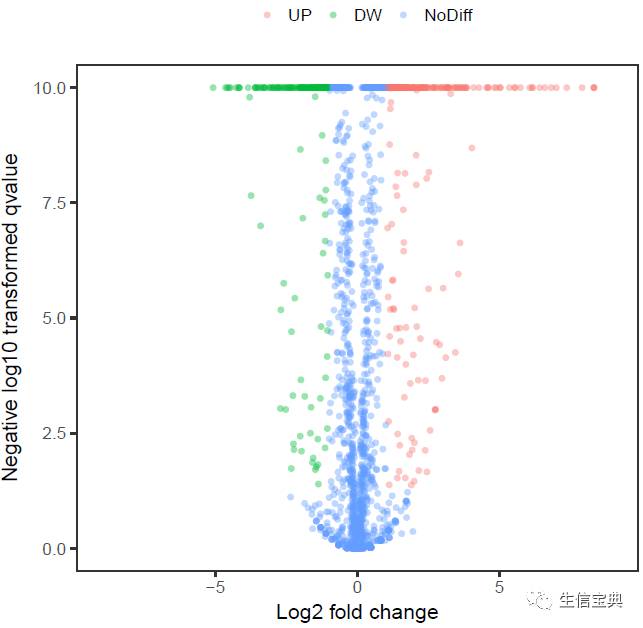
p_volcano.sh -f volcano.txt -x log2FoldChange -y padj -s significant -S "'EHBIO_UP', 'Baodian_UP', 'Unchanged'" -P TRUE -L top -M 10
注意看纵轴的变化,和最上面排成一条线的一堆点。

自动计算significant列绘制火山图
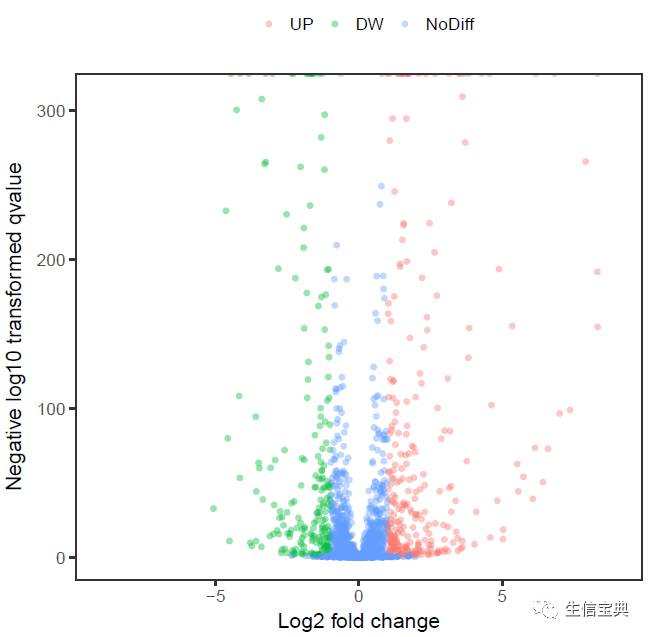
若不存在significant列,程序会根据-F指定的参数计算并标记差异基因。-F的默认值为"0.05,1"(引号是必须的), 第一个数表示pvalue或padj,对应于<-y>列;第二个数表示对数转换的差异倍数,对应于<-x>列。
sp_volcano.sh -f volcano.txt -x log2FoldChange -y padj -P TRUE -L top
sp_volcano.sh -f volcano.txt -x log2FoldChange -y padj -P TRUE -L top -M 10
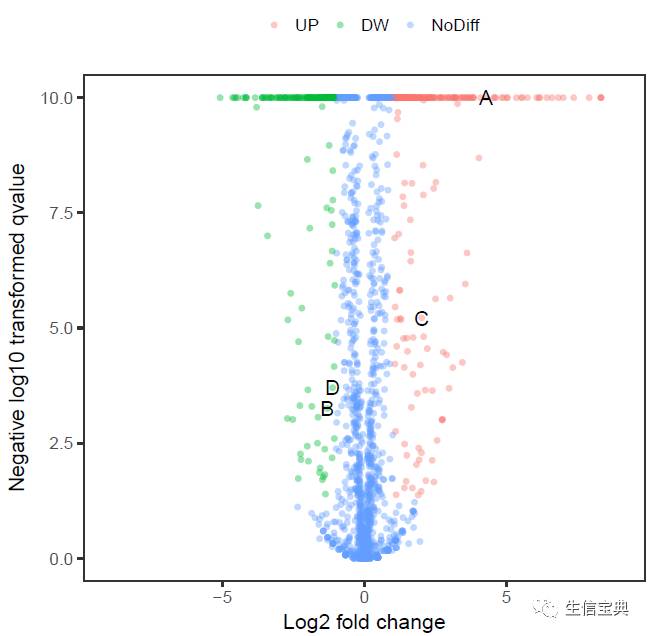
火山图中标记基因的名字
sp_volcano.sh -f volcano.txt -x log2FoldChange -y padj -P TRUE -L top -M 10 -l label
label列中非-的值都会标记在图上。
 今天先到这,前天提到的富集分析图,今天的火山图都是散点图的一种,后续介绍散点图时再对用到的R代码进行解读。
今天先到这,前天提到的富集分析图,今天的火山图都是散点图的一种,后续介绍散点图时再对用到的R代码进行解读。
需要绘图脚本的,还是请帮助转发下,谢谢。
测试文件的链接 http://pan.baidu.com/s/1i5LzDxr。
R绘图学习
R语言学习 - 入门环境Rstudio
R语言学习 - 热图绘制 (heatmap)
R语言学习 - 基础概念和矩阵操作
R语言学习 - 热图美化
R语言学习 - 热图简化
R语言学习 - 线图绘制
R语言学习 - 线图一步法
R语言学习 - 箱线图(小提琴图、抖动图、区域散点图)
R语言学习 - 箱线图一步法
R语言学习 - 富集分析泡泡图 (文末有彩蛋)
程序学习心得
生物信息之程序学习
如何优雅的提问
文章集锦
Linux学习 R统计绘图 Python教程 Perl学习
生信傻瓜 NGS持续更新中
(链接失效,点击菜单知识库查看)
关注生信宝典,换个角度学生信

关注宏基因组,再专业一点

Reference
http://blog.genesino.com/2017/07/volcanoPlot















