尽管抗肿瘤治疗获得了较多的进展,但是大部分病人最终因为肿瘤转移而治疗失败,这也不外乎小细胞肺癌SCLC,也让更多学者的关注点集中在肿瘤转移机制的研究上。今天分享的文献,讲述发现转录因子驱动染色质状态改变,促进了SCLC转移的机制研究。

文章
2016年7月发表在Cell的文章,IF=28.71,“Nfib Promotes Metastasis through a Widespread Increase in Chromatin Accessibility”,即“Nfib通过广泛增加染色质的易接近性促进转移”,作者首先发现SCLC转移灶的染色质相较于原位肿瘤细胞发生了显著的改变,染色质变得更容易接近,而这是由Nfib驱动的,随后带来的是调控多种促转移相关的神经元基因以及其他相关的表达,是SCLC转移所必须的。
思路
1.
发现SCLC存在两种不同染色质状态。
首选作者成功建立SCLC动物模型(图A-C),并分选出Tomato-
negative, GFP-positive标记的肿瘤细胞(包括原位肿瘤细胞和转移灶肿瘤细胞)。将获取的四组原位肿瘤细胞和肝转移灶肿瘤细胞,进行ATAC测序分析染色质的可接近性相关性,将肿瘤细胞分成了两种,一种为大部分包含原位肿瘤细胞,另一种为大部分包含转移细胞(图D)。

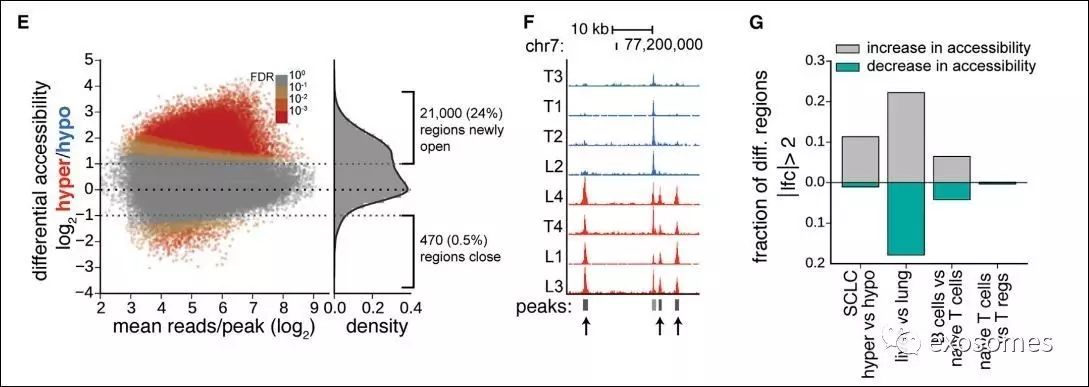
作者就上述两组进一步分析,发现以转移细胞为主的肿瘤细胞群中开放了更多的DNA可结合区,提示其染色质的可接近性显著增强(图E-F)。
初步暗示SCLC肿瘤细胞中存在两种不同染色质状态。
额外作者对比了转移灶之间,以及淋巴细胞亚群之间的染色质状态(图G),发现转移灶之间的差异不显著,而淋巴细胞亚群中存在差异,
这是否表明可通过染色质状态反映SCLC转移进程?
2.
状态改变的染色质有什么特点?
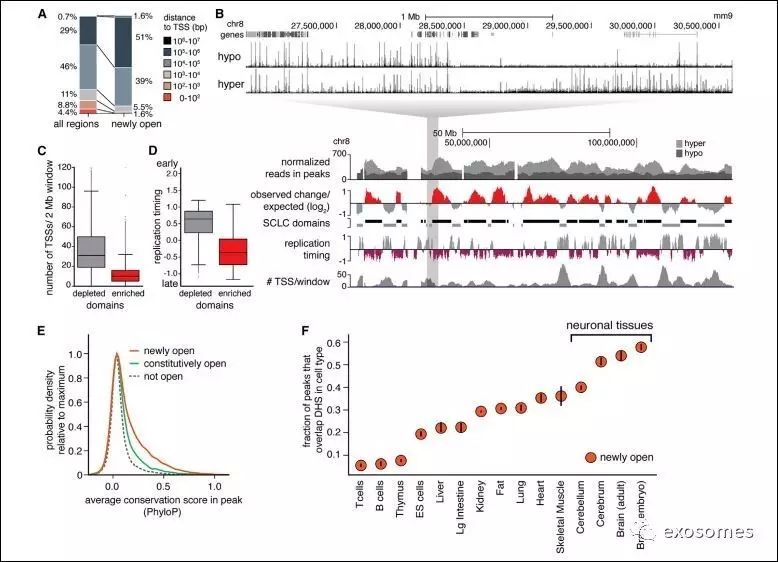
易接近的染色质区多远离转录起始点(图A),基因的密度较低
(图C),复制时间迟(图D),但是这些区的基因高度保守(图E)。为了判断这样的染色质状态是否仅存在与SCLC转移细胞内,作者还对其他组织细胞的染色质状态进行分析,发现脑组织细胞的染色质也多处于易接近状态(图F),而SCLC是一种神经内分泌肿瘤,其中是否有一定的联系?
3.
可是到底是什么驱动了染色质状态的改变呢?
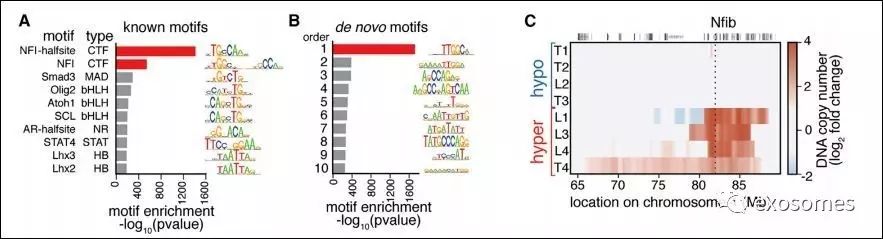
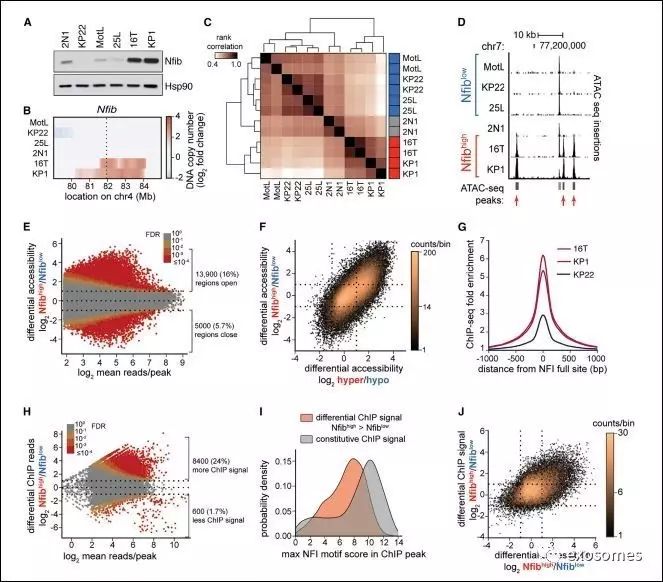
作者发现这些染色质新开放的区域富集了很多转录因子模体,其
中最多的为Nfi(核因子I)家族(图A),对比hyper-染色质可接近组与hypo-染色质可接近组,前者Nfib存在明显扩增现象(图C)。
那Nfib的表达水平是否与SCLC转移有关呢?动物肿瘤模型免疫组化结果提示SCLC晚期肿瘤发生转移后,Nfib高表达;在高侵袭性SCLC以及转移灶内Nfib也高表达;并呈现一种循序渐进的转移发生的进展过程,原位肿瘤细胞,DTC(游离肿瘤细胞),转移灶肿瘤细胞Nfib阳性细胞群渐渐增多。
均提示了Nfib在一定程度上可能启动了SCLC的转移。
返回上一层思考,Nfib是如何改变染色质的可接近性,重新开放了DNA结合区的?作者通过Tn5转座酶是基因片段化,测序分析转录因子印迹,作者观察到Nfib区结合了更多的转录因子,与核小体形成竞争关系(图G-I),
这可能诱导了染色质结构改变,或许促进了相关保守基因的转录。

4.
体内验证SCLC中不同的染色质可接近状态与Nfib相关。
6种鼠源SCLC细胞中KP1 ,16T Nfib在基因以及蛋白水平均高
表达,且Nfib-high细胞的染色质具有较高的可接近性,更多新开发的DNA结合区,可结合更多转录因子,更强的Chip信号。
5.
现象是观察到了,Nfib当真是控制染色质可接近性状态的关键?
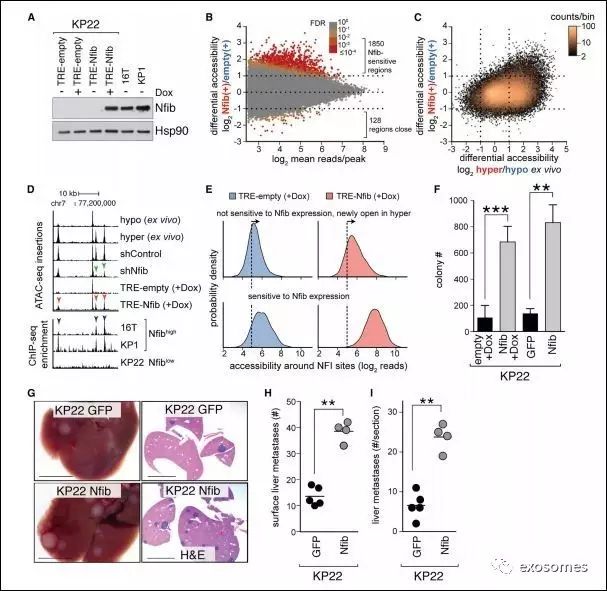
作者尝试敲除Nfib做了一系列验证实验,成功敲除Nfib后,新
开放的DNA结合区显著减少,染色质的可接近性降低,核小体与转录因子的结合增加,但动物模型中自发性肝脏转移仍然存在,不过转移灶显著减少,肿瘤细胞的克隆形成能力也降低,
提示Nfib是调控染色质状态,维持SCLC转移能力所必须的。

反向诱导添加Nfib表达相应实验,进一步
确定了Nfib是控制开放DNA结合区,改变染色质状态,以及介导维持SCLC转移能力的关键。
6.
上述提到在脑组织中也存在染色质高可接近性的现象,猜想是否Nfib介导开放了神经相关的基因促进了转移?
的确敲除或反向添加Nfib后,相应区域基因开放情况,基因表
达随之改变,而这些基因多与神经功能相关,包括轴突导向、突触组织和神经系统发育调节。

文章在这里结束了,其实就是Nfib让染色质结构变得疏松,促使更多转录因子有机会接近,促进高度保守的促进转移的神经元相关基因的转录表达,最终促进了SCLC的转移。

文章来源:
Denny SK, Yang D, Chuang C-H, Brady JJ, Lim JS, Grüner BM, et al. Nfib Promotes Metastasis through a Widespread Increase in Chromatin Accessibility[J]. Cell. 2016,166(2):328-42.