浙大生信博士团队倾力打造的一个科研人员学习交流的公众微信平台。我们致力于科研社区服务,分享最前沿的科技进展,提供生信分析方法,解读经典分析案例,公众数据库的挖掘和临床数据统计分析。在此我们欢迎各位的加入!戳
这里
生信草堂公众号原文,请多关注哦~
据WHO统计,目前吸烟每年会带走至少六百万人的生命。
照这个趋势下去,本世纪会有超过10亿烟草相关的死亡。从流行病学角度,吸烟与至少17种人类癌症相关,但直到现在,还没有人找到吸烟导致癌症发生的背后机制。
多年来,科学研究发现吸烟可以对身体不同器官造成的灾难性的基因损伤,并不断试图寻找吸烟导致疾病发生的病理机制。

下面我们向大家介绍一篇SCI论文(Georgiadis, Hebels et al. 2016),看它如何利用已有的数据,通过再次提出新的问题,并回答问题。
本文利用的组学数据包括
三种
:gene expression
(Agilent 4 × 44K human whole genome microarray platform), CpG
methylation (Illumina Infinium HumanMethylation450 platform), miRNA
expression profiling [Agilent Human miRNA Microarray (Release 19.0, 8 ×
60K), representing 2006 human miRNAs].
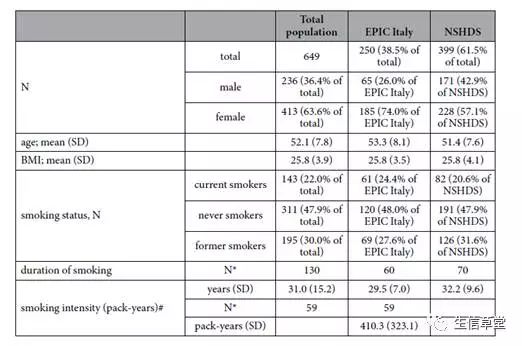
这些数据基于649个健康人的血液样本,详情请见图1,处理方法已经在已发表的文章描述过
(
Hebels, Georgiadis et al. 2013
,
Coonen, Theunissen et al. 2015
)。
之前的研究主要关注于发现与吸烟暴露相关的生物标记(Biomarker)。
本文整合这些已发表的数据,回答科学问题:基于健康者血液样本,这些发现的与吸烟相关的转录组和甲基化组改变是否与吸烟相关的疾病发生有关。
为了这一目的,他们进行一系列生物信息学分析如下:
一. 吸烟诱导基因表达、DNA甲基化及miRNA表达的改变
首先,作者们分别对基因表达、DNA甲基化和miRNA表达数据进行了常规的差异分析,发现了一些差异基因。
A.
吸烟者
VS.
非吸烟者
,作者们发现了350个差异表达转录本(FDR<0.1,其中231 FDR<0.05),这些转录本可以定位到271个差异表达的基因上,且大部分基因是低表达基因。
B.
吸烟者
VS.
非吸烟者
,作者们发现了1,273个差异CpG位点(FDR <0.05),这些差异甲基化位点在吸烟者中是低甲基化的,它们定位到了725个基因上。
其中最显著的基因是AHRR,它上面有27个CpG位点显著(FDR<0.05)。
这是很重要的结果,因为这个基因是吸烟甲基化研究中证据最多,结果最有力的。这里发现了它,可以有力的证明了结果的可靠性。不然就不好解释啦。
C.
吸烟者
VS.
非吸烟者
,作者们发现了34个差异表达的miRNA,其中26个高表达和8个低表达。
二. 疾病关联分析
第一部分的分析发现了一系列的与吸烟相关的差异基因,这一部分作者们想建立这些发现的差异基因与疾病之间的关系。
这里用到了一个很好用的数据库:The Comparative Toxicogenomic Database
(
http://ctdbase.org
)。
这个数据库包涵了许多信息:chemical-gene/protein interactions, chemical-disease 和 gene-disease关系(如图2)。
图2. The Comparative Toxicogenomic Database首页
通过这个数据库的疾病富集分析,可以找到差异基因富集的疾病List。这里作者们分别对差异表达基因集、差异甲基化基因集及它们的合并基因集进行了疾病富集分析(如图3)。
这里大家一定会意识到通过这样的疾病数据库分析,会有许多的疾病被富集,其中有一些疾病在流行病学研究中没有任何证据与吸烟相关。这个时候就要考虑到有可能是假阳性富集导致的这一现象产生。
所以作者们就想到了一个解决办法:
利用已有的流行病学知识进行有效排除,即对那些有充足流行病学证据的疾病作进一步研究。
这里他们用到
两个数据来源
:the US Surgeon General’ s report on the health consequences of smoking和the latest IARC Monograph on tobacco。
整合比较后找到一些较为可靠地富集疾病List
,如一系列吸烟相关的癌症 (详见图4)
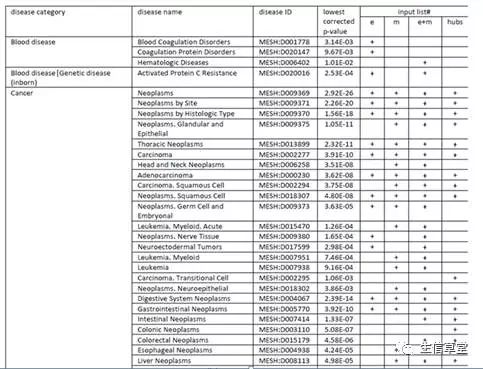
图3. 通过不同的基因集富集相关的疾病List
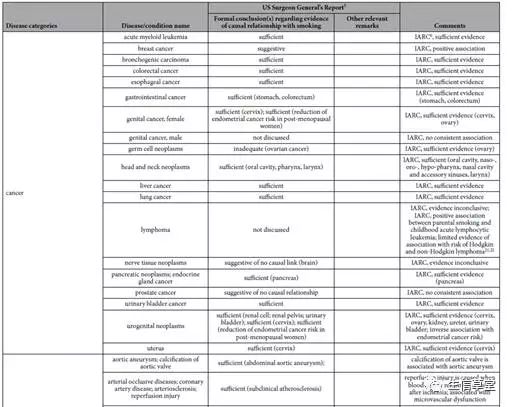
图4.
总结从the Comparative Toxicogenomic Database富集的疾病list与the US Surgeon
General’s Report on the health consequences of smoking和 the IARC
Monograph on tobacco比较的结果。
三. 生物信息学分析
1. 信号通路富集分析
为了进一步研究吸烟相关的这些差异表达(DEG)/甲基化(DMG)基因在吸烟相关疾病中发挥作用的分子机制,他们进行了通路富集分析。
这里使用的通路富集分析软件是
:
ConsensusPathDB
(
http://consensuspathdb.org/
)。
该软件是利用DEG基因集、DMG基因集和合集进行富集分析,总共有894个基因。
通过通路富集分析,发现了97个显著富集的信号通路(FDR<0.05),这些通路都是与多种疾病相关,包括癌症与心血管疾病(如图5)。
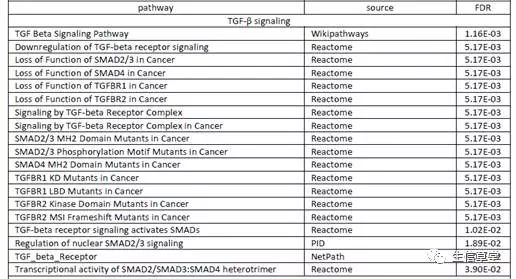
图5. 信号通路富集结果
2. hub基因的鉴定(identification of hub DEGs/DMGs)
因为作者们发现了大量的基因(n=894),很难进行重点研究





