
基因与基因之间的关系千丝万缕,
基因共表达网络(Gene co-expression network)
是用来分析基因间作用关系的一种常用手段。诚然,通过像STRING、PINA等数据库工具可以很容易的获取编码基因间的作用关系,但是只能获取已知的作用关系,并且结论可能会由于具体实验条件的不同而产生一定的偏差。此外,在预测非编码基因的靶基因时,除了各种靶基因预测工具,基因共表达网络分析也是一件利器。
其实基因共表达网络分析并非十分复杂的分析,用EXCEL就能搞定其中的数据处理。
先来说说
基因共表达网络分析的流程
:
1、获取基因表达数据,进行数据标准化;
2、筛选差异基因,构建差异基因的表达矩阵;
3、计算差异基因间的表达相关性系数;
4、利用Cytoscape构建网络,分析网络拓扑结构。
前两步的具体方法在前面的文章中讲过(
暴躁的EXCEL测序数据分析方法(前传),
暴躁的EXCEL测序结果分析方法
),这里就不赘述了,差异基因的表达矩阵利用
vlookup函数
即可构建。
今天实操过程中用的是GEO中
Series Matrix File(s)
格式的数据。
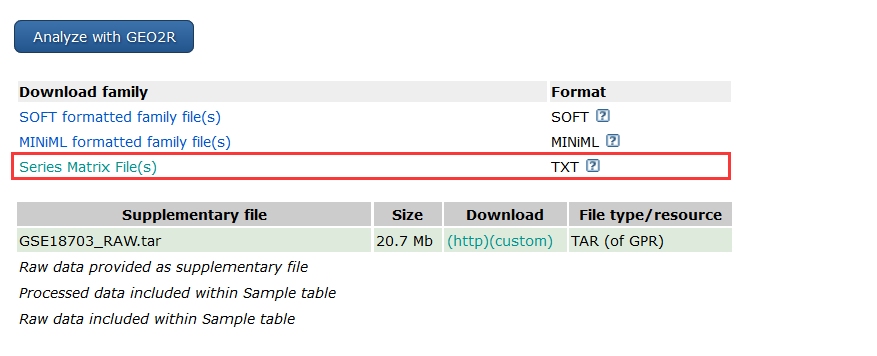
差异基因也没有筛选,只是随便选了50个基因进行分析演示
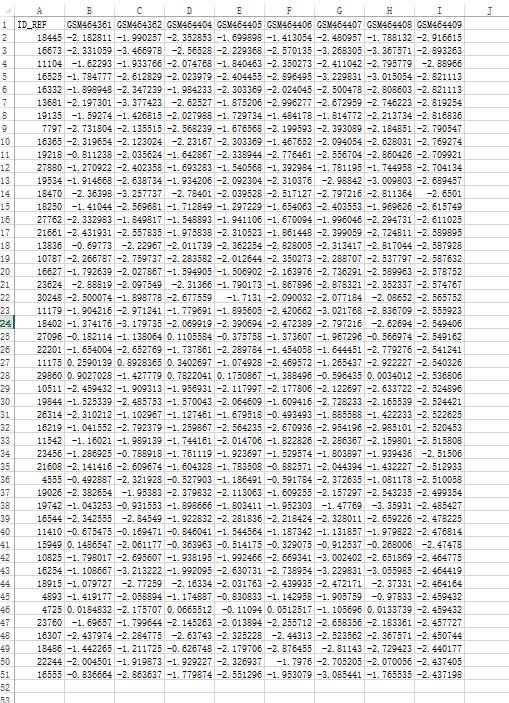
利用EXCEL(版本为2013)自带的工具分析相关系数。在左上角的“
文件
”里“
Excel选项
”“
加载项
”中,在“
非活动应用程序加载项
”中,找到“
分析工具库
”,然后在下方“
转到
”Excel加载项后,按“
确定
”,即可在“
数据
”选项中的分析下添加“
数据分析
”。
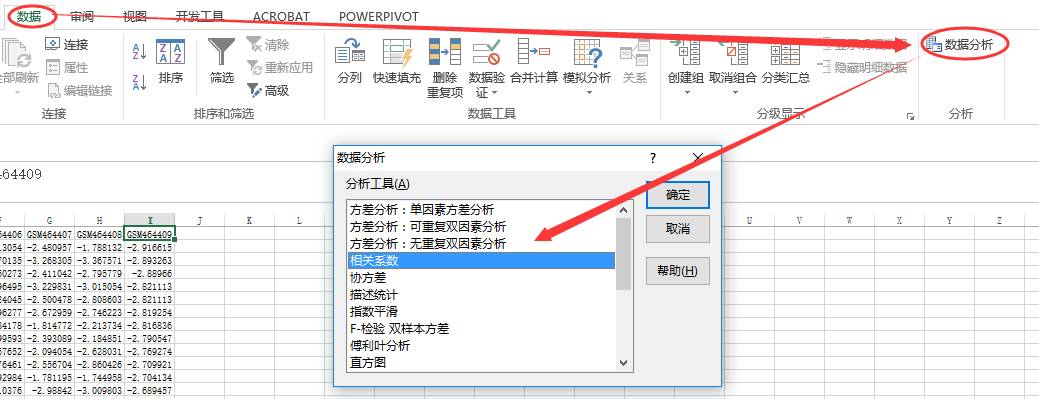
选择
逐行
分析,并将结果输出到新的表格中
然后把表头加上(
转置粘贴
的运用,见之前的微信文章)
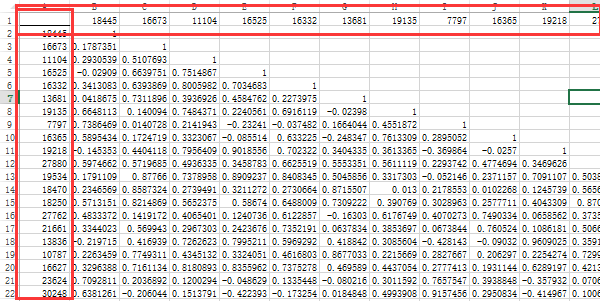
现在获取的是一个表达相关系数的矩阵,我们最后要生成网络图,这样的矩阵图是用不了的,必须得拆分成普通表格(如下图)。拆分矩阵我们需要用到这样的公式:
BA单元格:
=OFFSET($A$1,INT((ROW(A1)-1)/50)+1,0)
BB单元格:
=OFFSET($B$1,COLUMN(A1)-1,MOD(ROW(A1)-1,50))
BC单元格:
=OFFSET($B$2,INT((ROW(A1)-1)/50),MOD(ROW(A1)-1,50))
输入完成后,下拉填充即可。
公式中50这个数字是你的基因数量,本次演示用的是50个基因。
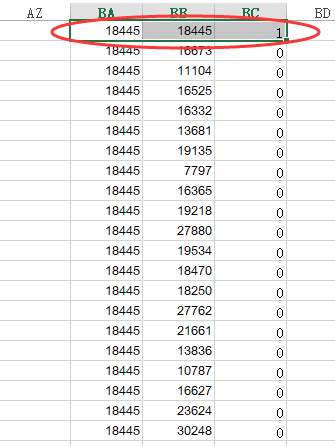
表达相关性的数据有了,建立筛选标准进行筛选(常用>0.98或如何用Cytoscape提升文章档次?)
讲完了~~听首歌消个愁吧~~

没看过瘾?那再给你们讲个EXCEL的高级用法——
定位
。
有没有傲娇的小童鞋说,我非要保留相关性系数的矩阵,我要把数据做成下图这样(
把大于0.9或小于-0.9的数据留下,其它全都清除
)。
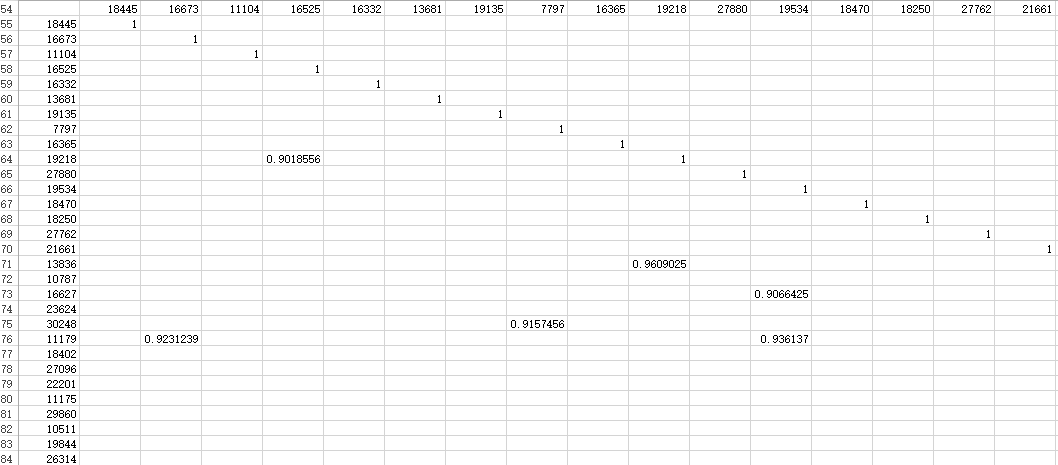
首先用
IF函数
做一个筛选
在B55单元格输入公式:
=IF(OR(B2>0.9,B2。
然后利用自动填充工具把整个区域都填充上数据。我们可以看到满足筛选条件的显示的是具体数值,不满足的显示是
FALSE
。

我们调出定位工具(快捷键为
F5
)
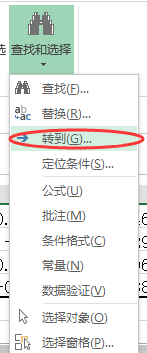
引用位置设置为数据区域
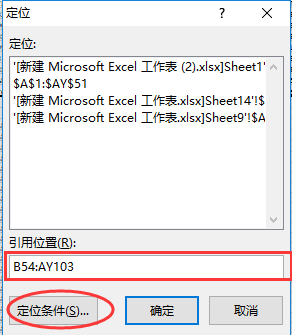
然后设置
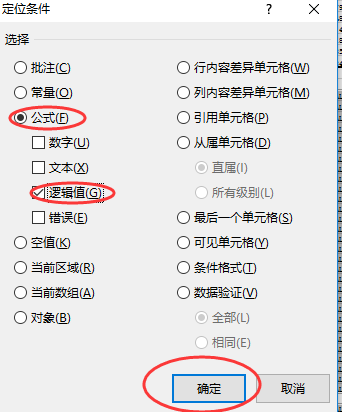
定位条件
,选择
公式
中的
逻辑值
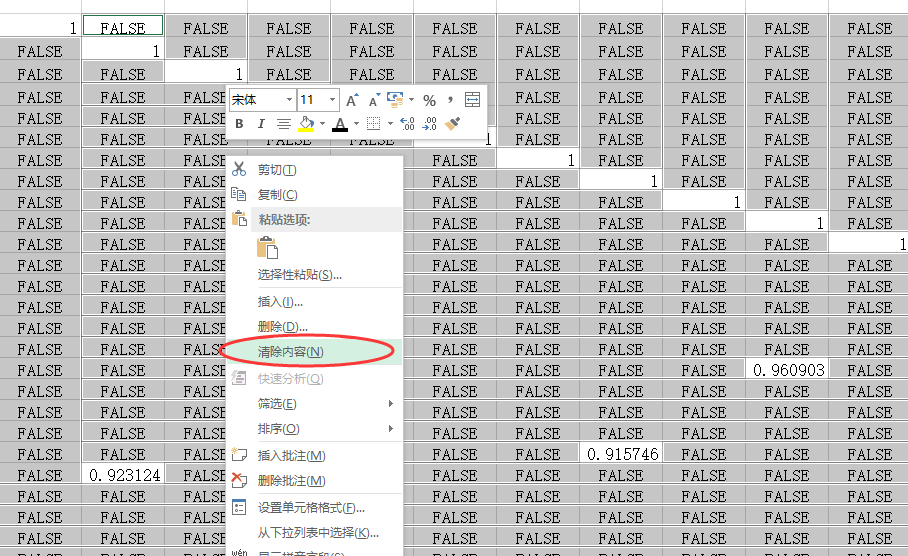
所有的FALSE就全部选中了,然后
清除内容
就大功告成了。
定位功能主要是利用条件格式来筛选出不规则的选择区域。常用的筛选标准还有
空值与其它
、
公式与其它
、
文本与其它
、
数字与其它
、
可见单元格与隐藏单元格
等。
定位的应用很多,比如
快速填充空值
。
下图中的左列如何做成右列?
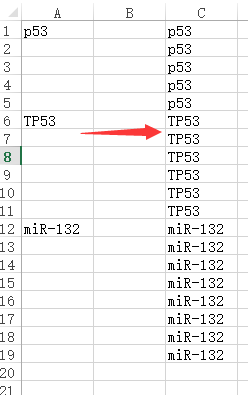
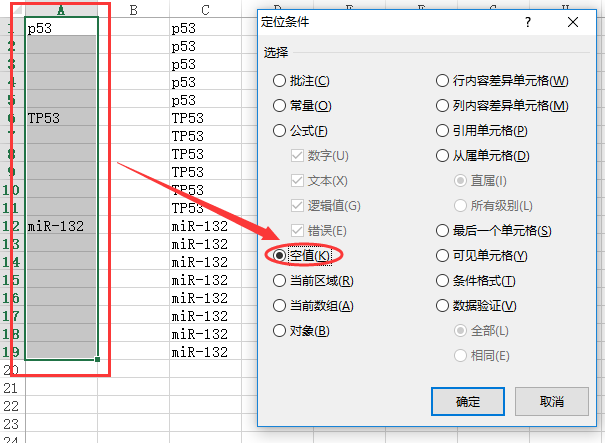
选中目标区域设置
定位条件
为
空值
在A2单元格中输入:=A1
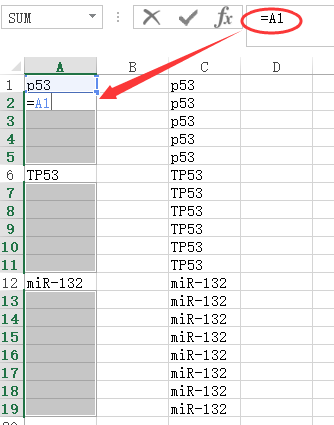
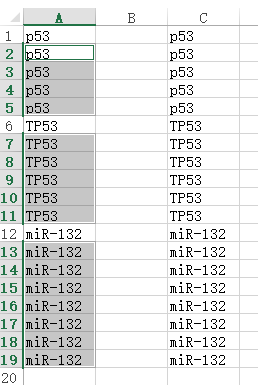
然后按
Ctrl+回车
即可完成填充。
今天演示的材料都上传到网盘中了,大家可以自己下载下来练练手















