MuTect&MuTect2
都是
GATK
下的一个子模块,
MuTect2
采用突变热点局部重比对和贝叶斯统计的方法,实现对
INDEL
的变异提取(
MuTect
不能提取
INDEL
),然后通过过滤
cosmic
数据库和
dbSNP
库、以及自己构建的
Panel of Normals (PoN)
(一般省略,很少有人自己构建),去除正常样本中的突变位点,最终得到高可信度的体细胞变异信息。
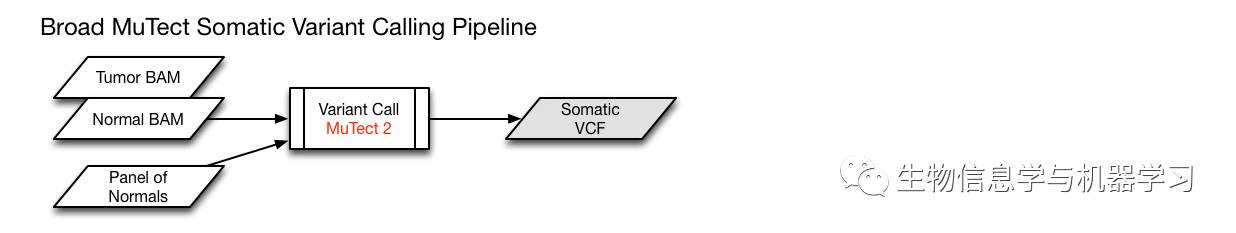
MuTect2
适用于
panel
和
WES
的肿瘤对照分析,不过速度相当慢,
WES(
白细胞
100
层
VS
组织
500
层
)
要
2
天的时间(因为只支持单线程,同
HaplotypeCaller
),
WGS
没跑过,估计更慢。对于小
panel
,深度测序,则需要调整参数,否则会得到空
VCF
文件。现在举一个小
panel
的例子对此说明。
之前本人做了
100
多个基因的
panel
,做的是
ctDNA
和白细胞对照测序,各
10000
层。自己搭建了
pipeline
,开始用默认参数,跑了
2
天流程,
VCF
文件还空空如也。此流程跑
500
层的
WES
却能正常工作。后来
google
研究,找到了原因,又去查官方文档,得到了验证。原来
MuTect2
默认参数设置很严格,
max_alt_alleles_in_normal_count
默认为
1
,也就是白细胞里只要发现
1
个变异,
MuTect2
就会认为该变异不属于
somatic
变异,并将其扔掉,试想
10000
层的深度,那还不全扔掉啊!这里推荐
max_alt_alleles_in_normal_count
参数设置为
1000
。同理,
max_alt_allele_in_normal_fraction
默认为
0.03
,建议改为
0.1
,这样就能保证即使
1000
起不到作用的时候(比如测序深度
<1000
层),也可以通过
0.1
过滤。
Variantfilt
官方暂时不推荐,因为在
GATK4.0
版会有新工具专门用于变异过滤,详情可参考
https://gatkforums.broadinstitute.org/gatk/discussion/9429/mutect2-and-vqsr-anyway-of-calling-vqslod-for-mutect2
10000X深度ctDNA测序,假阳性的变异多为:
C>A
与
G>T
突变(请读者思考为什么?)
对于
ctDNA
,面对这么多的假阳性(
oxidative DNA damage
,参考文章
Discovery and characterization ofartifactual mutations in deep coverage targeted capture sequencing data due tooxidative DNA damage during sample preparation
)我们就束手无策了吗?恐怕是的,暂时只能这样。
MuTect2
流程模板如下:
1
、创建
Panel of Normals (PoN)
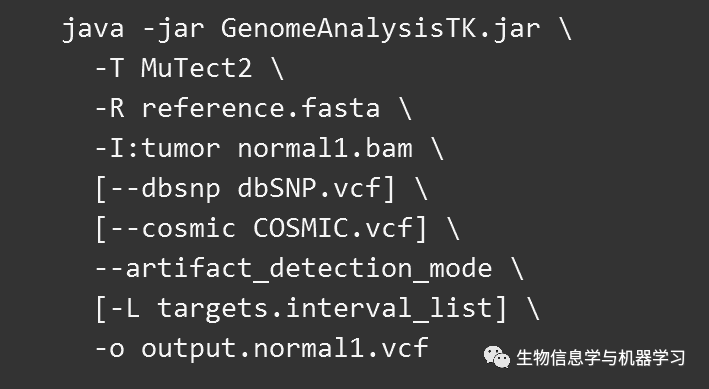
2
、
Tumor/Normal variant calling

--max_alt_alleles_in_normal_count 1000 \
--max_alt_allele_in_normal_fraction 0.1
(不要忘了最后
2
条)
后记:
多说一句,鉴于MuTect2运行速度过慢,小panel可以跑,WES勉强跑,WGS严重不推荐;或者
有
2
种(都是
GATK
开发的)办法可以改进,一种是通过
QUEUE
,还有一种是
WDL
。这里推荐用
WDL
,可以在本地、服务器、云平台上运行,是大趋势。





