正文
大家好,今天要和大家分享的是
2021
年
9
月发表的一篇文章:
“
Characterization of hypoxia response patterns identified prognosis and immunotherapy response in bladder cancer
”。
在本项研究中,作者使用无监督共识聚类方法综合评估了
1343
例膀胱癌
(BLCA)
患者的缺氧反应模式及其与基因组和临床病理特征的关联。确定了
5
种具有明显生存优势的缺氧反应模式
(HPXclusters)
,并使用最小绝对收缩和选择算子
(LASSO)
和
Cox
回归算法构建
HPXscore
以表示缺氧反应模式。低
HPXscore
组的特点是免疫激活和高
DNA
损伤修复,被称为免疫炎症表型。高
HPXscore
组中观察到基质相关通路的激活,被认为可能是免疫排斥表型。此外,文章还证明
HPXscore
是一个独立预后因素,可以作为
BLCA
免疫治疗结果的良好预测指标。
发表杂志
:
Mol Ther Oncolytics.
影响因子
:
7.201
明明是乏氧,为什么说是免疫分型呢?大家可以跟我们介绍过的两篇文章做对比。
6.6分纯生信免疫分型,换个肿瘤可重复
8.8分纯生信免疫分型,换个肿瘤可重复
其实这些文章的本质是:
基因集的单肿瘤分析
。不管是免疫基因,还是乏氧基因,以及还可以扩展的各种各样的基因集,都可以用这个思路。那么该模式就有很大的可扩展性。
需要进行该模式分析的朋友,可以联系小编咨询
请扫描下方二维码

研究背景
膀胱癌
(BLCA)
是指发生在膀胱黏膜上的恶性肿瘤,是泌尿系统最常见的恶性肿瘤。
BLCA
在组织学上被分为非肌肉浸润性
BLCA(NMIBC)
和肌肉浸润性
BLCA(MIBC)
。卡介苗
(BCG)
被批准为
NMIBC
治疗的一线免疫治疗药物,然而并非所有
NMIBC
患者都对
BCG
有反应,约
50%
的无反应者会复发或进展为
MIBC
,
5
年生存率
<50%
。肿瘤微环境
(TME)
包含多种细胞类型及分泌因子。在这样的微环境中,缺氧是细胞可塑性和肿瘤异质性的重要代谢因素。有研究报告指出,缺氧是通过激活缺氧
/HIF-1
α级联通路引发癌症侵袭和转移的重要触发因素。此外,化疗、放疗甚至免疫治疗敏感的癌细胞可能会转移到长期暴露于缺氧的耐药细胞中。
流程图
分析解读:
BLCA
缺氧反应相关基因的共识聚类
①
4
个微阵列数据集(
GEO
:
GSE13507
、
GSE32548
、
GSE32894
和
GSE48075
)和
TCGA
测序数据集(
TCGA-BLCA
)被合并为
meta-BLCA
队列。
②
UCSC Xena
收集
TCGA-BLCA
数据集的样本信息。
③分子特征数据库
(MSigDB)
下载缺氧反应相关基因集。
④
K-Means
聚类算法识别不同的缺氧反应模式
(HPXcluster)
。
⑤使用
R
软件“
limma
”包识别每个模式之间的
DEG
。
⑥
Kaplan-Meier
对队列中不同缺氧反应模式进行生存分析。
结果:
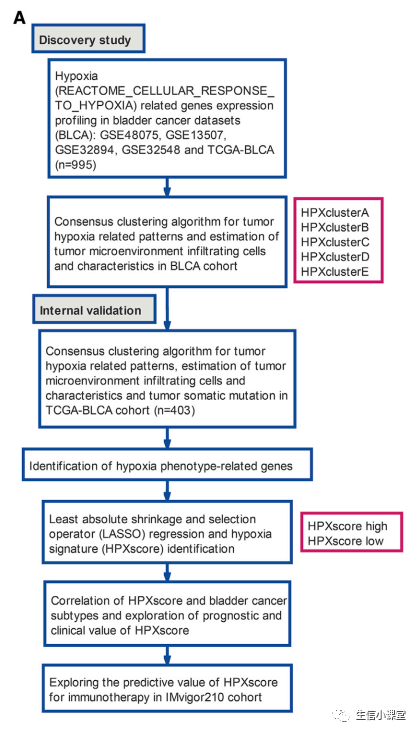
下图
B
:
meta-BLCA
队列(
GEO
:
GSE13507
、
GSE32548
、
GSE32894
、
GSE48075
和
TCGA-BLCA
)中患者的共识矩阵,
k=2-5
,使用
1000
次
K-Means
聚类算法确保稳定性。
下图
C
:
meta-BLCA
队列中缺氧反应相关基因的分层聚类。行代表缺氧反应相关基因,列代表
BLCA
样本。红色代表高表达,蓝色代表低表达。
下图
D
:使用
Kaplan-Meier
曲线对
meta-BLCA
队列中不同缺氧反应模式的生存分析。
HPXclusterE
显示出明显优于所有其他缺氧反应模式的
OS
(对数秩检验,
p=0.0133
)。
不同缺氧反应模式下的
TME
免疫细胞浸润和生物学过程特征
①通过单样本基因集富集分析
(ssGSEA)
计算
TME
免疫浸润细胞的相对数量。
②使用
R
软件中的“
GSVA
”包执行
ssGSEA
算法识别不同缺氧反应表型之间的生物过程差异。
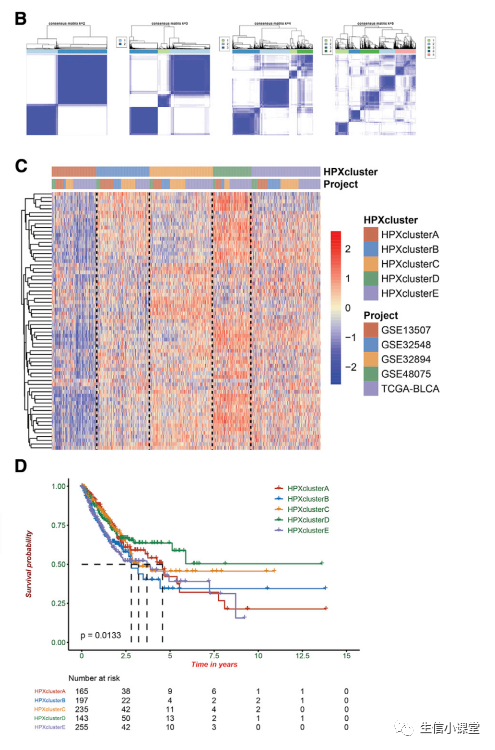
结果:
下图
A
:
meta-BLCA
队列中
995
名患者的
TME
免疫浸润细胞的分层聚类。
下图
B
:
ssGSEA
显示
meta-BLCA
队列中不同缺氧反应模式中生物途径的相对活性(包括免疫激活、抗原加工、错配修复和癌症通路等),热图用于可视化,红色代表通路相对激活,蓝色代表通路相对受到抑制。
下图
C
:
meta-BLCA
队列中
5
种不同缺氧反应模式之间的指示特征富集差异,以代表特定生物过程,包括基质激活相关特征、错配修复相关特征和免疫激活相关特征。
下图
D
:
meta-BLCA
队列中
5
种不同的缺氧反应模式之间
MHC
分子、共刺激分子和粘附分子的表达水平的差异。
TCGA-BLCA
队列中的缺氧反应模式
①在
TCGA-BLCA
队列中对上述分析结果进行验证。
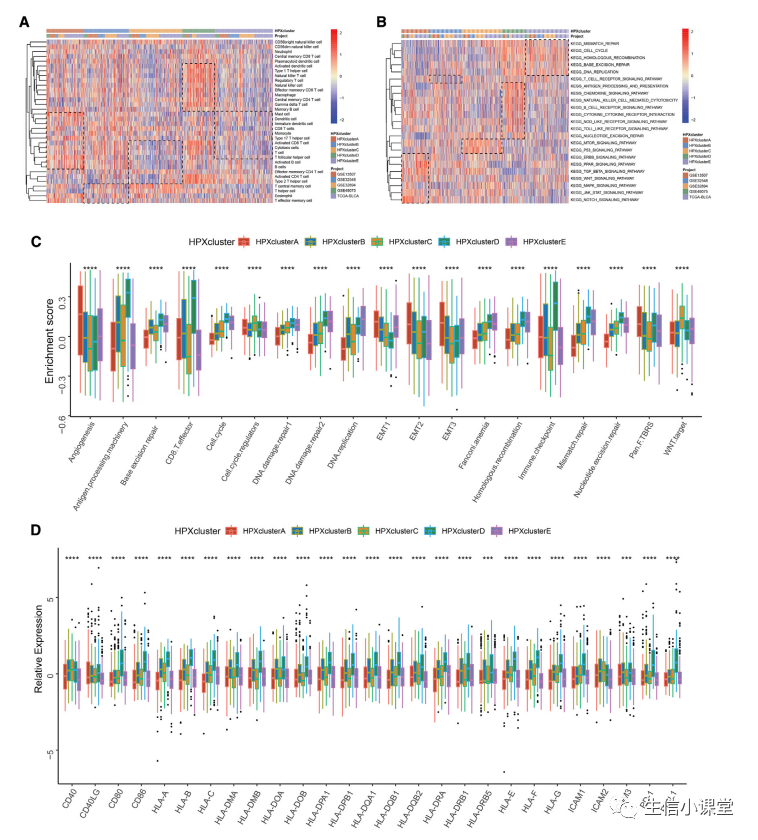
结果:
下图
A
:
Kaplan-Meier
生存曲线显示
TCGA-BLCA
队列中
5
种不同缺氧反应模式之间预后优势的差异(对数秩检验,
p=0.015
)。
下图
B
:
TCGA-BLCA
队列中
5
种不同缺氧反应模式中
403
名患者的
TME
浸润免疫细胞的分层聚类。
下图
C
:
TCGA-BLCA
队列中
5
种不同的缺氧反应模式之间
MHC
分子、共刺激分子和粘附分子的表达差异。
下图
D
:
TCGA-BLCA
队列中五种不同缺氧反应模式中免疫相关特征、基质相关特征和抗原加工机械相关特征的富集差异。
下图
E
:不同缺氧模式中
DEG
的基因本体论
(GO)
富集分析。
不同缺氧反应模式中的肿瘤体细胞突变
①使用
R
软件“
maftools
”和“
complexheatmap
”包研究
BLCA
中不同
HPXclusters
之间体细胞改变的分布差异。
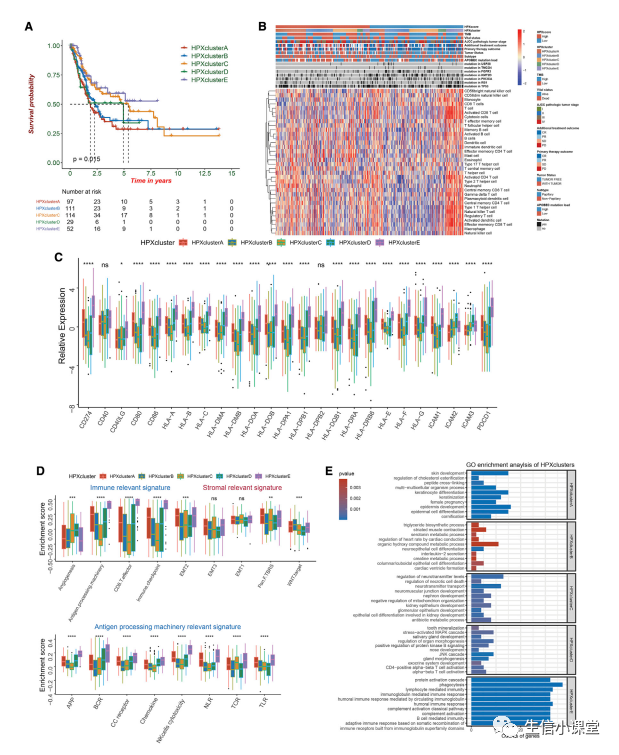
结果:
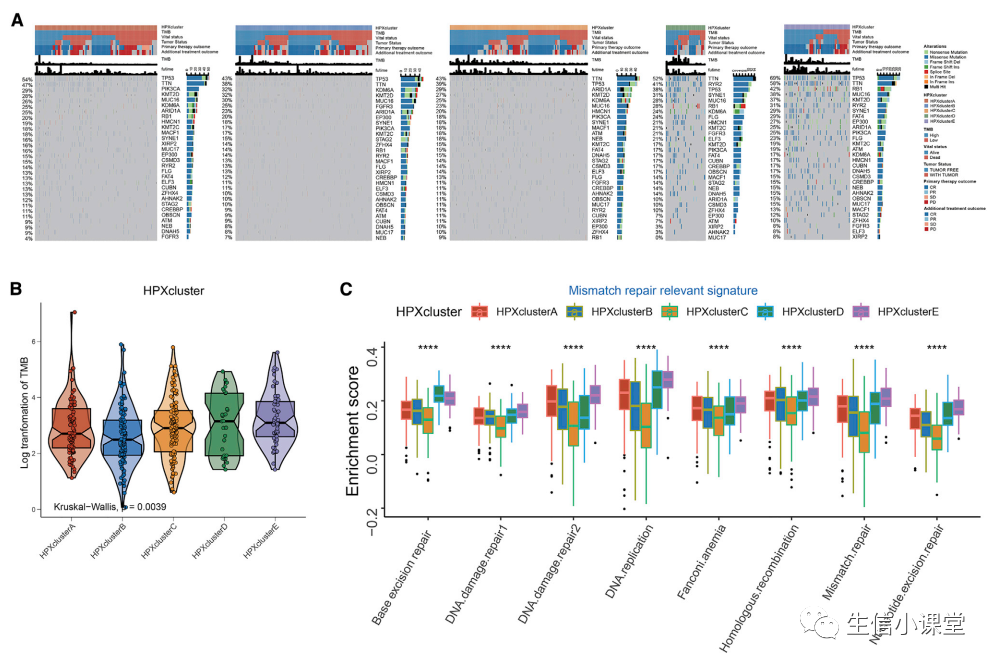
下图
A
:前
30
个突变基因在
BLCA
的
5
种不同缺氧反应模式中的分布。瀑布图注释中指示了遗传改变类型。上部条形图表示
TMB
和总生存
(OS)
时间。左右条形图上的数字显示了每个基因在不同缺氧反应模式中的突变频率。
下图
B
:不同缺氧反应模式之间
TMB
的差异。框的上端和下端代表值的四分位距。方框中的线代表中值。
Kruskal-Wallis
检验用于比较每个模式之间的统计差异(
p=0.0039
)。
下图
C
:
TCGA-BLCA
队列中
5
种不同缺氧反应模式中错配修复相关特征的富集差异。框的上端和下端代表值的四分位距。方框中的线条表示中值,黑点表示异常值。通过单向方差分析测试了五种不同缺氧反应模式之间的统计差异。∗∗∗∗
p<0.0001
。
缺氧表型相关基因特征(
HPXscore
)的转录组特征和生物学特征
①本文建立了一个评分系统,称为缺氧表型相关基因特征(
HPXscore
),以评估
BLCA
患者的缺氧反应模式。
②从不同的
HPXclusters
中识别出
DEGs
并合并为
meta-DEGs
。
③使用单变量
Cox
回归分析提取具有显著预后差异的
meta-DEG
作为候选
DEG
进行进一步分析。
④应用
R
软件中的
‘glmnet”
包的
LASSO-Cox
回归分析为
BLCA
构建最佳缺氧表型相关基因特征。
⑤每个样本的
HPXscore
由每个缺氧表型相关基因的相对表达及其相关的
Cox
系数定义。
HPXscore=
∑
ni=1(coefi
×
Expri)
,其中
Expri
是患者
i
签名中基因的相对表达,
coefi
是基因
i
的
LASSO-Cox
系数。
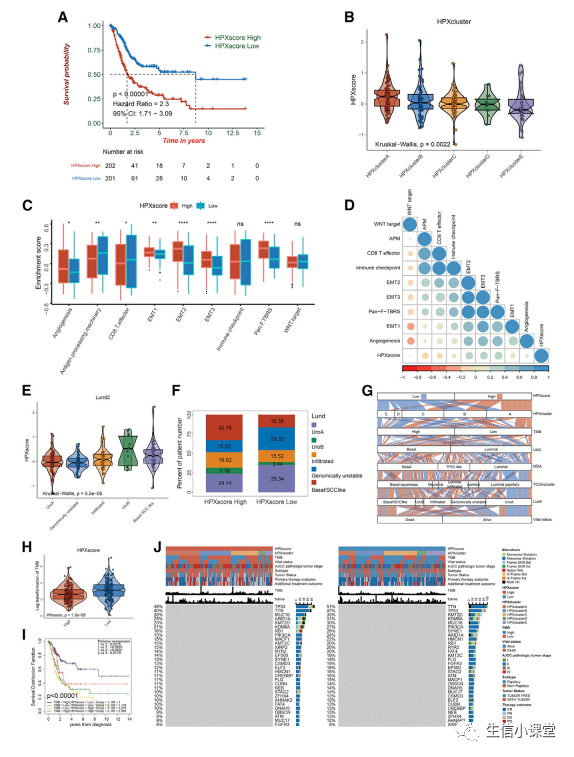
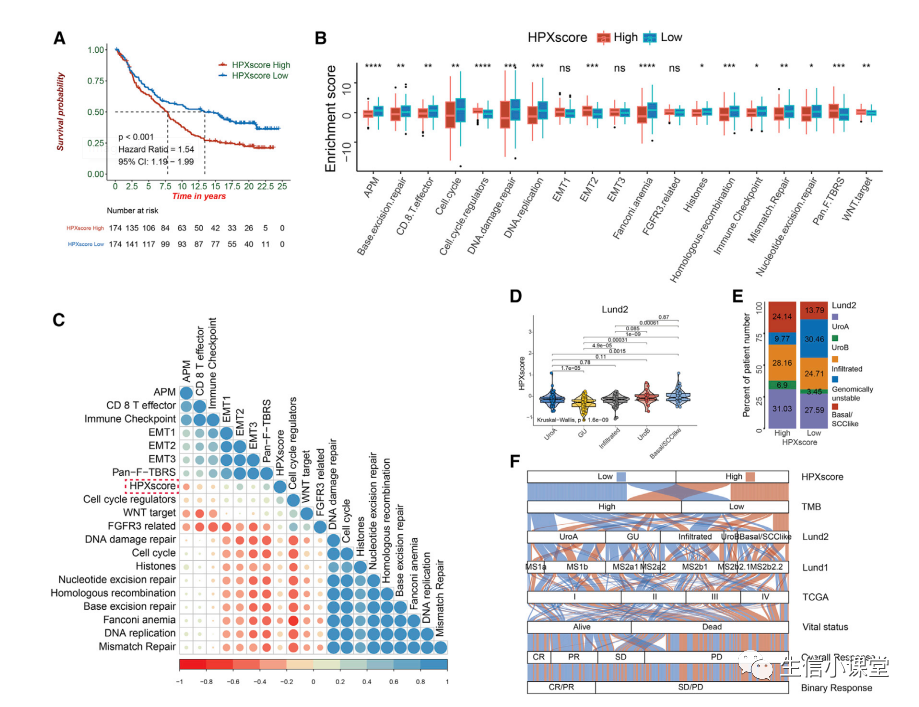
结果:
下图
A
:
Kaplan-Meier
生存曲线显示
TCGA-BLCA
队列中高和低
HPXscore
组之间预后优势的差异(对数秩检验,
p<0.00001
)。
下图
B
:
HPXscore
在
5
种不同缺氧反应模式中的分布。
Kruskal-Wallis
检验用于比较每个模式之间的统计差异(
p=0.0022
)。
下图
C
:
TCGA-BLCA
队列中高和低
HPXscore
组之间免疫激活相关特征和基质激活相关特征富集的差异。
下图
D
:
HPXscore
与
TCGA-BLCA
队列中与免疫和基质激活相关的基因特征之间的相关性。负相关用蓝色标记,正相关用红色标记。
下图
E
:
Lund
分类系统中不同分子亚型之间
HPXscore
的差异。
Kruskal-Wallis
检验用于比较不同分子亚型之间的统计差异(
p=5.5e-5
)。
下图
F
:
TCGA-BLCA
队列中高和低
HPXscore
组中
Lund
分子亚型的比例。使用
Fisher
精确检验测量统计差异(
p=0.004467
)。
下图
G
:冲积图显示
TCGA-BLCA
队列中
HPXscore
、
HPXclusters
、
TMB
、指示分子亚型和生命状态的变化。
下图
H
:
TCGA-BLCA
队列中高和低
HPXscore
组之间
TMB
的差异。使用
Wilcoxon
检验测量统计差异(
p=1.3e-5
)。
下图
I
:
Kaplan-Meier
生存曲线显示在
TCGA-BLCA
队列中由
HPXscore
和
TMB
分层的
4
组之间预后优势的差异(对数秩检验,
p<0.00001
)。
下图
J
:
TCGA-BLCA
队列中高和低
HPXscore
组之间的前
30
个突变基因的分布。上面的条形图表示
TMB
和
OS
时间。左右条形图上的数字显示了每个基因在不同缺氧反应模式中的突变频率。
HPXscore
可作为
BLCA
的独立预后因素
①通过单变量和多变量
Cox
回归分析确定
HPXscore
是否能作为
BLCA
患者的临床独立预后因素并构建列线图。
结果:
下图
A-B
:
HPXscore
和临床病理学特征的单变量
(A)
和多变量
(B)Cox
分析的森林图。
下图
C
:基于
5
个独立预后因素预测
3
或
5
年
OS
的列线图。∗∗
p<0.01
,∗∗∗
p<0.001
。
下图
D-E
:用于预测
3
年和
5
年
OS
的列线图校准曲线。
下图
F-G
:基于
3
年和
5
年风险的
5
个独立预后因素的列线图决策曲线分析
(DCA)
。

HPXscore
是
ICI
免疫疗法临床反应的预测生物标志物
①探讨
HPXscore
在
IMvigor210(mUC)
队列免疫治疗中的反应。
②
Kaplan-Meier
进行生存分析。
结果:
下图
A
:
Kaplan-Meier
生存曲线显示
IMvigor210(mUC)
队列中高和低
HPXscore
组之间预后优势的差异(对数秩检验,
p<0.001
)。
下图
B
:
IMvigor210(mUC)
队列中高和低
HPXscore
组之间指定基因特征的富集差异。
下图
C
:
HPXscore
与
IMvigor210(mUC)
队列中指示的基因特征之间的相关性。
下图
D
:
IMvigor210(mUC)
队列中
Lund
分类系统中不同分子亚型之间
HPXscore
的差异。
Kruskal-Wallis
检验用于比较不同分子亚型之间的统计差异(
p=1.6e-9
)。
下图
E
:
IMvigor210(mUC)
队列中高和低
HPXscore
组中
Lund
分子亚型的比例。使用
Fisher
精确检验测量统计差异(
p=0.005722
)。
下图
F
:冲积图显示
IMvigor210(mUC)
队列中
HPXscore
、
TMB
、指示分子亚型、生命状态、
ICI
总体反应和二元反应的变化。
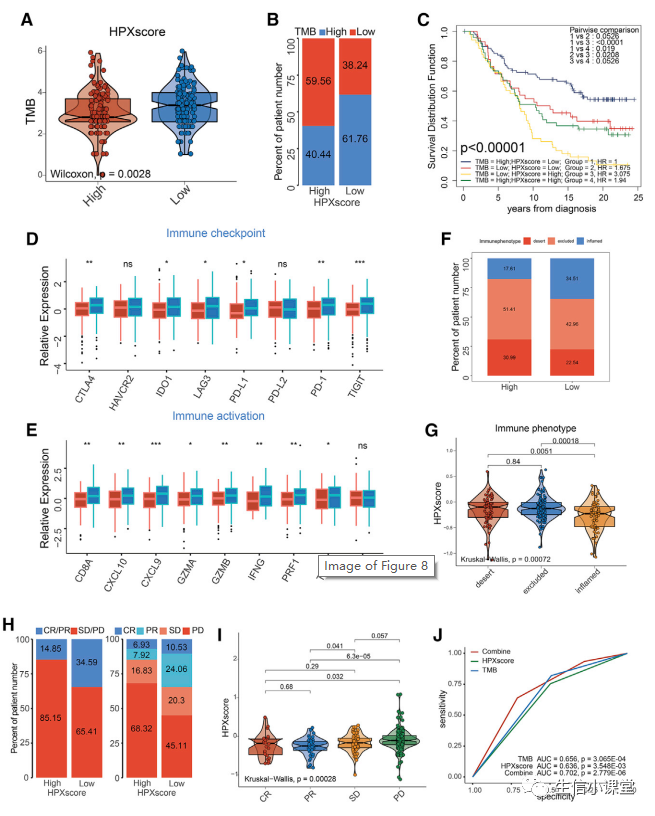
结果:
下图
A
:
IMvigor210(mUC)
队列中高和低
HPXscore
组之间的
TMB
差异。使用
Wilcoxon
检验测量统计差异
(p=0.0028)
。
下图
B
:
IMvigor210(mUC)
队列中高和低
HPXscore
组中
TMB
水平的比例。使用
Fisher
精确检验测量统计差异(
p=0.001859
)。
下图
C
:
Kaplan-Meier
生存曲线显示了在
IMvigor210(mUC)
队列中由
HPXscore
和
TMB
分层的
4
组之间预后优势的差异(对数秩检验,
p<0.00001
)。
下图
D-E
:
IMvigor210(mUC)
队列中高和低
HPXscore
组之间免疫检查点
(D)
和免疫激活相关基因
(E)
的表达差异。
下图
F
:
IMvigor210(mUC)
队列中高和低
HPXscore
组之间免疫表型的比例。使用
Fisher
精确检验测量统计差异(
p=0.033555
)。
下图
G
:
IMvigor210(mUC)
队列中不同免疫表型之间
HPXscore
的差异。
Kruskal-Wallis
检验用于比较不同分子亚型之间的统计差异(
p=0.00072
)。
下图
H
:
IMvigor210(mUC)
队列中低或高
HPXscore
组对
ICI
免疫疗法有反应的患者比例。
下图
I
:不同
ICI
免疫治疗临床反应组之间
HPXscore
的差异。使用
Kruskal-Wallis
检验测量统计学差异
(p=0.00028)
。
下图
J
:测量
HPXscore
、
TMB
及其组合在
IMvigor210(mUC)
队列中的预测值的受试者工作特征
(ROC)
曲线。
HPXscore
、
TMB
和
HPXscore
与
TMB
组合的
AUC
分别为
0.636
、
0.656
和
0.702
。
小结:
在这项研究中,作者通过对
1343
例
BLCA
患者进行综合评估,确定了
5
种具有明显生存优势、
TME
免疫细胞浸润、临床病理和基因组特征的缺氧反应模式
(HPXclusters)
。同时,作者还构建了一套量化个体缺氧反应模式的评分系统—缺氧表型相关基因特征
(HPXscore)
。研究表明,
HPXscore
能够可靠地全面评估
BLCA
个体的缺氧反应模式,并且与临床、细胞和分子特征相关,包括临床分期、组织学亚型、
TME
免疫细胞浸润、免疫表型、分子亚型、遗传变异和肿瘤突变景观。此外,
HPXscore
可以作为
BLCA
患者预后的独立预后因素,以及对
ICI
免疫治疗的临床反应的预测因素。
基因集泛癌分析文章列表
基因集泛癌分析,重磅来袭
想发高分纯生信文章,这也许就是你的机会
11.6分,最热复合物泛癌纯生信分析!想发高分纯生信不容错过
11分+SCI 基因集泛癌纯生信分析,两个多月接受,可重复!
快来看看最新的27分基因集泛癌分析是怎么做的
13+SCI 基因集泛癌分析纯生信 可重复
基因集泛癌分析
6.6分基因集泛癌分析纯生信 并没有很难
25天接受的基因集泛癌分析,先人一步的优势!
7.6分基因集泛癌纯生信分析,从投稿到接收仅一个月!
最新6.6分,45天接收的基因集泛癌纯生信 值得尝试!















