
欢迎点击「PaperRss」↑关注我们!
所有真核生物都有三个基本的细胞核多亚基RNA聚合酶,简称Pol I,Pol II和Pol III
。植物有两个其他的多亚基RNA聚合酶,分别是Pol IV和Pol V,是合成非编码RNA,协调RNA介导的DNA甲基化,用于沉默转座子和大量的基因。基于它们的亚基组成中,Pols IV和V很明显是Pol II的特化形式,但是它们的催化活性尚未确定。在这里,Pikaard研究组展示,Pol IV和V及Pol II在核苷酸掺入率,转录准确性和辨别力核苷酸和脱氧核糖核苷酸的能力彼此不同。 Pol IV 转录比Pol V及Pol II更容易出错。相比之下,Pol V展现出高保真的转录,与Pol II类似,表明需要Pol V 的转录本完全忠实于DNA序列,这序列恰好也是 siRNA–AGO沉默复合物募集的地方。

在所有真核生物中,有三个细胞核多亚基RNA聚合酶,对于生存力是至关重要的:RNA聚合酶I(Pol I),合成三种最大核糖核酸的前体;Pol II,转录数千mR
NAs和非编码RNA;以及Pol III,转录5S核糖体RNA和tRNA。 但是,植物不同于动物,植物还有两个额外的细胞核多亚基RNA聚合酶,Pol IV和Pol V各自由12个亚基组成【1】,其中至少七个与Pol II共享【2-5】。 在siRNA介导的DNA甲基化(RdDM)途径中,这主要是沉默转座子,病毒外源序列以及转基因序列,Pol IV同Pol V有非冗余的生物学作用。
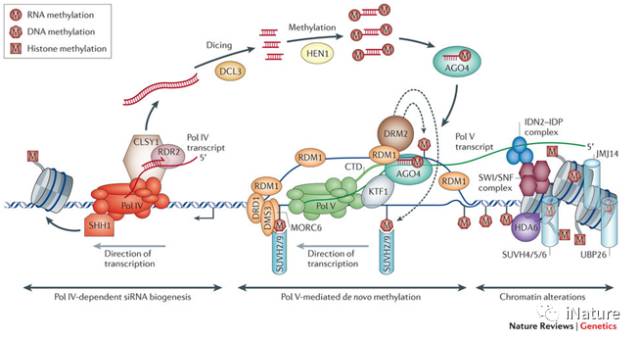
siRNA介导的DNA甲基化途径
Pol IV与RNA依赖的RNA聚合酶 2(RDR2)合作,产生双短链RNA,然后DCL3蛋白将其切成24nt siRNA【6-8】。 这些siRNA掺入Argonaute家族蛋白里面,主要是AGO4,并引导胞嘧啶甲基化和抑制性染色质修饰,定位到由Pol V转录的位点上【9-11】。 还有证据表明,PolV转录位点的RNA介导的DNA甲基化可以由来自转座子mRNA降解的21个核苷酸大小的siRNA引导【12-15】。
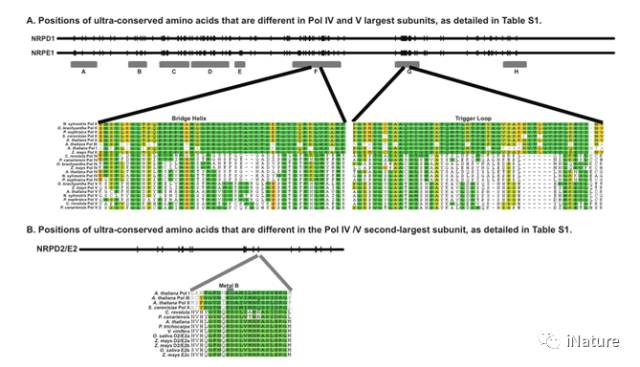
Pol IV和Pol V保守性分析
Pols IV和V显然对其进化的限制比其他聚合酶更少,允许其亚单位组成【5】及其催化亚单位发生巨大的变化,就比如氨基酸替换率比Pol II【3】高十到二十倍。在Pols I,II和III的催化亚基中不变的140个氨基酸位置中,在Pols IV和V是高度可变的【16】。这些包括被认为对聚合酶功能至关重要的氨基酸的取代和缺失,包括
trigger loop及 bridge helix结构域【17】。在Pol II和其他聚合酶中, trigger loop及 bridge helix 的构象变化导致从开放状态(其允许核苷酸三磷酸(NTP)进入催化中心)转变到NTP已经被关闭的状态的转变【18,19】。Ratchet-like的过渡在开放和封闭的构象之间被认为是与RNA易位相关,影响延长率以及精度(保真度)【20,21】。
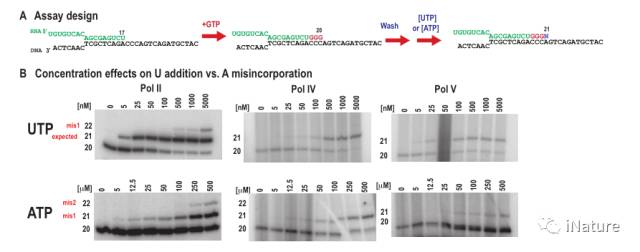
尽管在
trigger loop及 bridge helix结构域
和其他保守结构域内的氨基酸的缺失和置换,Pols IV和V是功能性的。并且已被证明在体外可以合成RNA【22】。 然而,相对于Pol II来说,它们的准确性和速度尚未得到研究。
在这里,
Pikaard研究组
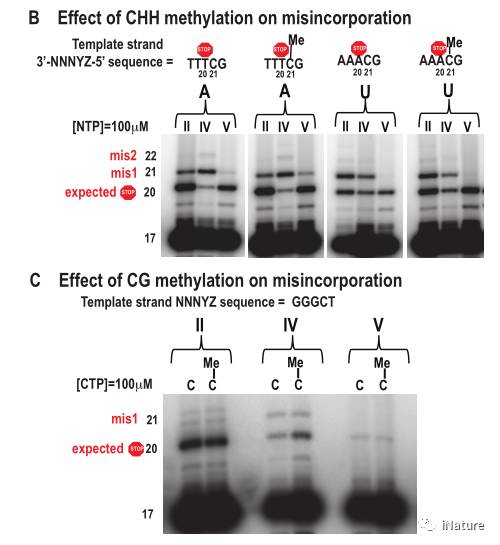
报告了Pol II,Pol IV和Pol II的几个参数的初步测量。显示Pols IV和V催化RNA合成比Pol II更慢
,Pol IV转录特别容易出错。 令人惊讶的是,Pol V比Pol II相比,更不容易出错,至少对于与DNA模板错配的核糖核苷三磷酸(rNTP)是这样的。 不过,相比于Pol II而言,Pol IV与Pol V表现出更差的区别核糖核苷和脱氧核糖核酸能力。
原文链接
https://academic.oup.com/nar/article/doi/10.1093/nar/gkx794/4103599/Catalytic-properties-of-RNA-polymerases-IV-and-V#96141091
通讯作者简介

Craig S. Pikaard
Craig S. Pikaard,现在是印第安纳大学布鲁明顿分校的教授,同时也是HHMI研究员,美国国家科学院院士(2017年入选)。Pikaard是一位生物化学家和遗传学家,主要研究基因转录,表观遗传学和基因沉默工作。他特别以表观遗传学现象,核仁优势以及两种植物特异性RNA聚合酶(简称Pol IV和Pol V)的研究而著称,对植物中RNA指导的DNA甲基化和基因沉默至关重要。
Pikaard出生在新泽西州,在百慕大开始小学,然后在宾夕法尼亚州西切斯特长大,参加了韦斯顿朋友学校。他于1980年毕业于宾夕法尼亚州立大学,获得园艺学学士学位,1985年毕业于普渡大学,获植物生理学博士学位。他在西雅图的Fred Hutchinson癌症中心进行了博士后研究,并于1990年加入了圣路易斯华盛顿大学的生物学系。2009年,他搬到布鲁明顿的印第安纳大学担任杰出教授与卡洛斯·米勒教授的同时任命(生物与分子与细胞生物化学系)。他于2011年被评为HHMI研究员,并于2015年被美国植物生物学家协会授予Martin Gibbs勋章。Pikaard于2017年当选为国家科学院院士。
Craig Pikaard的实验室对如何将特定基因的沉默感兴趣。一个重点是核糖体RNA(rRNA)基因的选择性沉默,真核生物有数百个rRNA基因,序列几乎相同。在早期发育过程中,几乎所有rRNA基因都是活性的。然而,后来在发育过程中,只有一部分rRNA的是活跃的。 Pikaard的实验室已经表明,非表达基因被DNA甲基化和抑制性组蛋白修饰失活,选择哪些基因沉默在某种程度上由基因的染色体位置决定,而不是它们的序列。了解选择机制的分子基础的研究正在进行中;实验室的第二个重点是了解核多重亚单位RNA聚合酶IV和V在RNA介导的DNA甲基化(RdDM)中的作用,这是沉默转座因子以及基因亚型所需的过程。使用体外进行的生物化学测定和体内遗传和基因组实验的组合,Pikaard的实验室正在努力了解Pol IV和Pol V转录如何启动,终止并与RdDM途径中其他酶的活性相结合。
Craig Pikaard在Nature,Cell,Science 等顶尖杂志发表了数百篇篇文章,其领导的实验室在RdDM途径以及基因的选择性沉默等方面的研究硕果累累,在国际上享有盛誉。
Craig Pikaard代表性文章
Jing Wang, Todd Blevins, Ram Podicheti, Jeremy R. Haag, Ek Han Tan, Feng Wang and Craig S. Pikaard (2017). Mutation of Arabidopsis SMC4 identifies condensin as a co-repressor of pericentromeric transposons and conditionally-expressed genes.
Genes and Development
(in press)
Jered M. Wendte, Jeremy R. Haag, Jasleen Singh, Anastasia McKinlay, Olga M. Pontes and Craig S. Pikaard (2017) Functional dissection of the Pol V largest subunit CTD in RNA-directed DNA methylation.
Cell Reports
19:2796-2808.
Michelle Marasco and Craig S. Pikaard (2017). Catalytic properties of RNA polymerases IV and V: accuracy, nucleotide incorporation and rNTP/dNTP discrimination. bioRxiv 117184; doi: https://doi.org/10.1101/117184
Todd Blevins, Jing Wang, Frédéric Pontvianne and Craig S. Pikaard (2017). Hybrid incompatibility caused by an epiallele.
Proc. Natl. Acad. Sci. 114:3702-3707.
Anastasia McKinlay,
Ram Podicheti,
Ross Cocklin,
Craig S Pikaard,
Douglas B Rusch (2017).RNA polymerases IV and V influence the 3′ boundaries of Polymerase II transcription units in Arabidopsis. bioRxiv preprint, first posted online Feb. 15, 2017;
Jered Wendte and Craig S. Pikaard (2017). The RNAs of RNA-directed DNA methylation.
Biochem. Biophys. Acta
. 1860:140-148;
Jered M. Wendte and Pikaard, Craig S. (2016). Targeting Argonaute to chromatin.
Genes & Development
30:2649-2650.
Gireesha Mohannath, Frederic Pontvianne and Craig S. Pikaard (2016). Selective nucleolus organizer inactivation in Arabidopsis is a chromosomal position-effect phenomenon.
Proc. Natl. Acad. Sci
. 113: 13426-13431.
Chinmayi Chandrasekhara, Gireesha Mohannath, Todd Blevins, Frederic Pontvianne and Craig S. Pikaard (2016). Chromosome-specific NOR inactivation explains selective rRNA gene silencing and dosage control in Arabidopsis.
Genes & Development
30:177–190.
Gireesha Mohannath and Craig S. Pikaard (2016). Analysis of rRNA gene methylation in
Arabidopsis thaliana
by CHEF-Conventional 2D gel electrophoresis. In:
The Nucleolus- Methods and Protocols
; a volume of the
Methods in Molecular Biology
series
.
Edited by Attila Nemeth. Humana Press, a part of Springer Science - Business Media, LLC.
Frédéric Pontvianne, Marie-Christine Carpentier, Nathalie Durut, Veronika Pavlištová, Karin Jaske, Sarka Schorova,_Hugues Parrinello, Marine Rohmer, Craig S. Pikaard, Miloslava Fojtova, Jiri Fajkus and Julio Saez-Vasquez (2016). Identification of nucleolus-associated chromatin domains in A. thaliana reveals a role for the nucleolus in 3D organization of the A. thaliana genome.
Cell Reports.
http://dx.doi.org/10.1016/j.celrep.2016.07.016
Todd Blevins, Ram Podicheti, Vibhor Mishra, Jing Wang, Doug Rusch, Haixu Tang and Craig S. Pikaard (2015). Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding
de novo
DNA methylation in Arabidopsis.
eLife
2015, Oct 2;4. pii: e09591. doi: 10.7554/eLife.09591.
Ream, Thomas S., Jeremy R. Haag, Frederic Pontvianne, Carrie D. Nicora, Angela D. Norbeck, Ljiljana PašaTolić and Craig S. Pikaard. (2015). Subunit compositions of
Arabidopsis
RNA polymerases I and III reveal Pol I- and Pol III-specific forms of the AC40 subunit and alternative forms of the C53 subunit.
Nucleic Acids Research
43: 4163-4178.
Craig S. Pikaard and Ortrun Mittelsten-Scheid (2014). Epigenetic Regulation in Plants. In:
Epigenetics
; edited by David Allis, Thomas Jenuwein and Danny Reinberg. Cold Spring Harb Perspect Biol. 2014 Dec 1;6(12). pii: a019315. doi: 10.1101/cshperspect.a019315. Review.
Haag, Jeremy R., Brent Brower-Toland, Elysia K. Krieger, Lyudmila Sidorenko, Carrie D. Nicora, Angela D. Norbeck, Andre Irsigler, Huachun LaRue, Jan Brzeski, Karen McGinnis, Sergey Ivashuta, Ljiljana Pasa-Tolic, Vicki L. Chandler, and Craig S. Pikaard (2014). Functional diversification of maize RNA polymerase IV and V subtypes via alternative catalytic subunits.
Cell Reports
9: 378-390.
Todd Blevins, Frédéric Pontvianne, Ross Cocklin, Ram Podicheti, Chinmayi Chandrasekhara, Satwica Yerneni, Chris Braun, Brandon Lee, Doug Rusch, Keithanne Mockaitis, Haixu Tang and Craig S. Pikaard (2014). A two-step process for epigenetic inheritance in Arabidopsis.
Molecular Cell
54:30-42.
Pikaard, Craig S. Nucleolar Dominance. In: eLS 2014, John Wiley & Sons Ltd: Chichester http://www.els.net/ [DOI: 10.1002/9780470015902.a0005976.pub2]
Thomas Ream, Jeremy Haag and Craig S. Pikaard (2014). Plant multisubunit RNA polymerases IV and V. In: Nucleic Acid Polymerases, edited by Katsuhiko Murakami and Michael Trakselis. Springer-Verlag, Berlin Heidelberg.
Frederic Pontvianne, Todd Blevins, Chinmayi Chandrasekhara, Iva Mozgova, Christiane Hassel, Olga M.F. Pontes, Sarah Tucker, Petr Mokros, Veronika Muchova, Jiri Fajkus, and Craig S. Pikaard (2013). Subnuclear partitioning of rRNA genes between the nucleolus and nucleoplasm reflects alternative epiallelic states.
Genes and Development
27:1545–1550.





