本来我想放男人装的封面作为题图,但是这样子估计就没人看教程了,所以还是放弃了。
这篇里面所有的教程和数据都在上一篇讲完了,没准备好的回去看
当你拿到测序数据后,就可以按照如下几步处理数据。第一步是
数据质控控制
,这一步对于组装而言非常重要,处理前和处理后的组装结果可能会天差地别;第二步,根据经验确定
起始参数
,如K-mer和覆盖率;第三步,使用不同软件进行组装;第四步,评估组装结果,如contig N50, scaffold N50, 判断是否需要修改参数重新组装。
尽管目前的测序技术已经非常成熟,公司提供的数据一般都可以直接用于普通的项目(特殊项目如miRNA-seq除外)。但由于
de novo
组装对数据质量比较敏感,因此需要通过质控来降低偏差。原始数据质量控制分为四个部分:
-
了解数据质量: 了解质量这一步可以暂时忽略,基本上基因组测序的结果都能通过FastQC的标准。
-
去接头和低质量reads过滤: 去接头和低质量reads过滤可供选择的软件非常之多,如NGSQCToolkit, Trimmomatic, cutadapter, 似乎都是国外开发的软件,但其实国内也有一款很优秀的工具叫做
fastp
-
去除PCR重复: 去重一般都是在比对后根据位置信息进行,没有基因组的话只能根据PE的reads是否完全一样进行过滤。从理论上说,测序相当于是从基因组上随机抽样,不太可能存在完全一摸一样的两条序列。不过貌似只有FastUniq能做这件事情,后来有一个人写了sequniq。
-
reads修正: 除了过滤或修剪低质量的reads外,一般而言,还需要对reads中的错误碱基进行修正。尤其当测序的覆盖度比较高时,错误的reads也就越来越多,会对
de novo
组装造成不良的影响。工具有BLESS2, BFC, Musket等,其中
BLESS2
的效率最高,效果也不错。
去接头和低质量reads过滤这一步推荐
fastp
,主要是因为它基于C/C++,运行速度块。
# 使用, 项目文件夹下
mkdir -p clean-data{lib1,lib2}
~/opt/biosoft/fastp/fastp -i raw-data/lib1/frag_1.fastq.gz -I raw-data/lib1/frag_2.fastq.gz -o clean-data/lib1/frag_1.fastq.gz -O clean-data/lib1/frag_2.fastq.gz
效果非常的惊人,直接干掉了90%的reads,从原来的1,294,104条变成77,375,一度让我怀疑软件是否出现了问题,直到我用同样的代码处理现在Illumina的测序结果以及看了FastQC的结果才打消了我的疑虑,没错,以前的数据质量就是那么差。
注
,除非是去接头,否则不建议通过删除序列的方式提高质量。
质控另一个策略是对短读中一些可能的错误碱基进行纠正,测序错误会引入大量无意义的K-mers,从而增加运算复杂度。此处使用
BFC
对测序质量:
~/opt/biosoft/bfc/bfc -s 3m -t 16 raw-data/lib1/frag_1.fastq.gz | gzip -1 > clean-data/lib1/corrected_1.fq.gz
~/opt/biosoft/bfc/bfc -s 3m -t 16 raw-data/lib1/frag_2.fastq.gz | gzip -1 > clean-data/lib1/
corrected_2.fq.gz
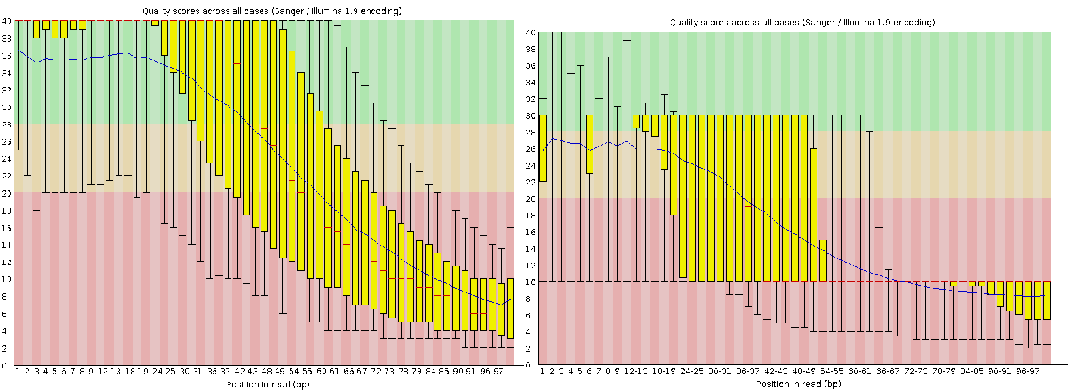 处理前后
处理前后
总之,质控的目标是在不引入的错误的情况下尽量提高整体质量,这一步对后续的组装影响很大,所以尽量做这一步,除非组装软件要求你别做,那你就不要手贱了。
二代组装可供选择的工具很多, 但是主流其实就那么几个, 所以组装的时候选择3~5个工具运行比较结果即可,比如说IDBA-UD, SOAPdenovo2, Abyss, velvet和Spades。当然一旦你选择一个软件准备运行的时候,你就会遇到参数选择问题,比如怎么确定k-mers,组装软件最基础也是最核心的参数。这里有几条原则值得借鉴:
-
k要大于log4(基因组大小),如果数学不好,无脑选择20以上
-
尽量减少测序错误形成的k-mers, 因为这是无意义的噪音, 也就是要求k不能过大
-
当然k也不能太小,否则会导致重复压缩,比如说ATATATA,在2kmers的情况下,就只有AT了
-
测序深度越高,K值也就可以选择的越大
但是说了那么多,你依旧不知道应该选择什么样的K,如果你的计算资源无限,那么穷举法最简单粗暴。如果穷举法不行,那么建议先用k=21, 55,77 组装一下contig, 对不同参数的contig N50有一个大致的了解,然后继续调整。此外还有一个工具叫做
KmerGenie
可以预测一个初始值。总之,让我们先运行第一个工具--SPAdes,可通过bioconda安装。
SPAdes全称是圣彼得堡基因组组装工具,包含了一系列组装工具处理不同的项目,如高杂合度的dipSPAdes,宏基因组的metaSPAdes。官方文档中以大肠杆菌为例运行整个流程,花了将近1个小时。我们的数据集比较小,速度会更快
# 项目根文件夹下
mkdir assembly/spades
spades.py --pe1-1 raw-data/lib1/frag_1.fastq.gz --pe1-2 raw-data/lib1/frag_2.fastq.gz --mp1-1 raw-data/lib2/shortjump_1.fastq.gz --mp1-2 raw-data/lib2/shortjump_2.fastq.gz -o assembly/spades/
你会发现之前说的k-mers在这里根本没出现,而且用的也是原始数据,这是因为
spades
.
py
有一个组件
BayesHammer
处理测序错误,并且它是多K类组装工具(multi-k assembly), 也就是说它会自动选择不同的K运行,从而挑选比较合适的k值,当然你还可以自己设置,比如说
-
k
21
,
55
,
77
。最后结果为纠正后的短读数据,组装后的contig, 组装后的scaffold, 不同格式的组装graph。
同样运行多k-mers运行后比较的工具还有IDBA,它也有一系列的工具。IDBA是基础版,IDBA-UD适用于宏基因组和单细胞测序的数据组装,IDBA-Hybrid则是基于相似的基因组提高组装结果,IDBA-Tran是专门处理转录组数据。对于无参考基因组组装,作者推荐使用IDBA-UD。
IDBA-UD工具要求将两个配对的短读文件合并成一个,我们的原始数据需要先用它提供的fq2fa先转换格式
# 项目文件夹下
mkdir -p assembly/idba_ud
~/opt/biosoft/idba/bin/fq2fa --merge zcat clean-data/lib1/corrected_1.fq.gz
) zcat clean-data/lib1/corrected_2.fq.gz) assembly/idba_ud/lib1.fa
~/opt/biosoft/idba/bin/fq2fa --merge zcat clean-data/lib2/corrected_1.fq.gz) zcat clean-data/lib2/corrected_2.fq.gz) assembly/idba_ud/lib2.fa
idba_ud
和k-mers相关参数为--mink,--maxk,--step, 通过
--
read_level_x
传入不同大小的文库,也提供了短读纠正的相关参数
--
no_correct
,--
pre_correction
~/opt/biosoft/idba/bin/idba_ud -r assembly/idba_ud/lib1.fa --read_level_2 assembly/idba_ud/lib2.fa -o assembly/idba_ud/ --mink 19 --step 10
运行结束后在
assembly
/
idba_ud
下会生成一系列的文件,其中结果文件为
contig
.
fa
和
scaffold
.
fa
。
最后介绍一个要手动运行不同k-mers的工具,如ABySS, 它有一个亮点,就是能够可以使用多个计算节点。我们使用k=31进行组装
mkdir -p assembly/abyss
# 增加 /1,/2
sed 's/^@SRR.*/&\/1/' zcat raw-data/lib2/shortjump_1.fastq.gz) | gzip > raw-data/lib2/s1.fq.gz
sed 's/^@SRR.*/&\/2/' zcat raw-data/lib2/shortjump_2.fastq.gz) | gzip > raw-data/lib2/s2.fq.gz
~/opt/biosoft/abyss-2.0.2/bin/abyss-pe -C assembly/abyss k=31 n=5 name=asm lib='frag short' frag='../../raw-data/lib1/frag_1.fastq.gz ../../raw-data/lib1/frag_2.fastq.gz' short='../../raw-data/lib2/s1.fq.gz ../../raw-data/lib2/s2.fq.gz' aligner=bowtie
注意,首先ABYSS要求双端测序的reads命名要以/1和/2结尾,其次第二个文库才37bp, 所以比对软件要选择bowtie,否则你运行一定会遇到
histogram xxx
.
hist
is
empty
的报错。当然到最后,这个问题我都没有解决掉,所以我
放弃
了。
虽然看起来abyss用起来很简单,但其实背后的工作流程还是比较复杂,如下是它的流程示意图
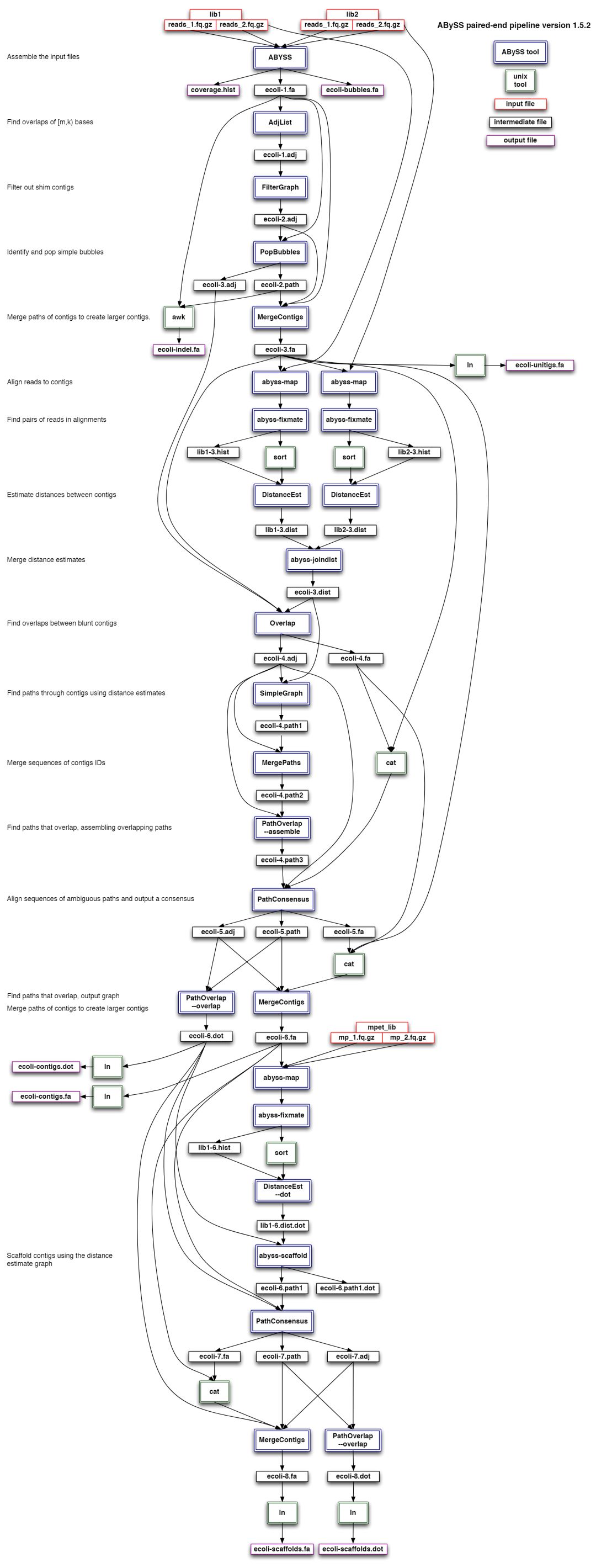 flowchart
flowchart
小结一下,这里用到了spades, idba,abyss三种工具对同一种物种进行组装,得到对应的contig结果,
核心
就是k-mers的选择。下面则介绍如何对组装结果进行评价。
基因组组装,简单也复杂
到底要不要组装基因组,没有调查就没有发言权
基因组组装之环境准备
顺便说一句,组装这个领域我也是刚进入,所以知识更新比较快,欢迎加入我的知识星球和我讨论






