上期
我们已经从GTEx下载提取了Liver的119个样本的表达谱文件,本期转换miRNA ID并将TCGA的样本与GTEx的进行合并.
PS:文末有福利.
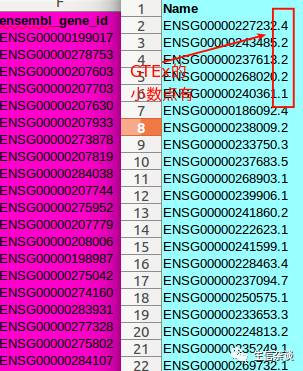
我们已经得到的GTEx的表达谱文件部分内容如下:
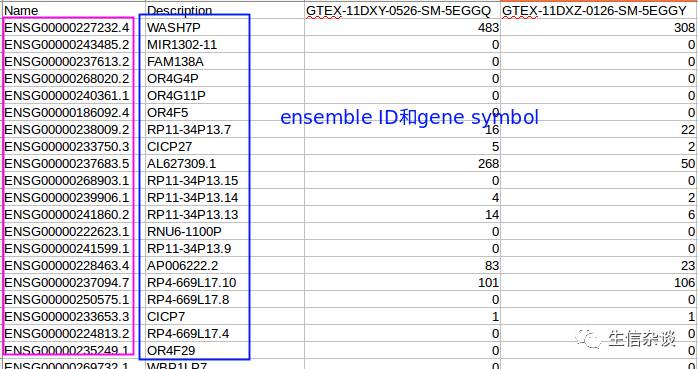
而我们之前得到的TCGA的表达谱格式是这样的:
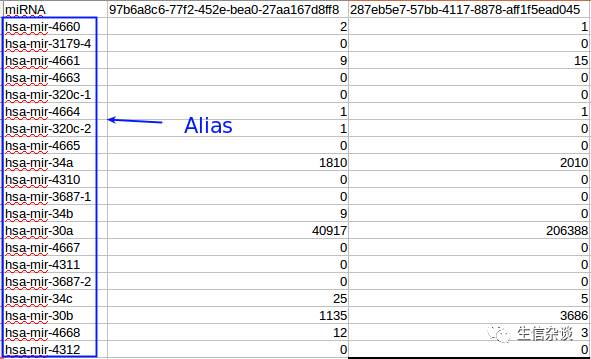
为了使两个数据库的ID对应起来,我们得找名称对应关系才行.这个可以在
人类基因命名委员会(HGNC)
(http://www.genenames.org) 找到.这个库包含了人类基因所有的命名,别名和ID,可以说是天下命名,无出其右.
可以直接在下载页面 (http://www.genenames.org/cgi-bin/statistics) 底部下载所有的命名关系的一个大表,但这次我们只需要处理miRNA的ID转换,那只下载miRNA的就行:
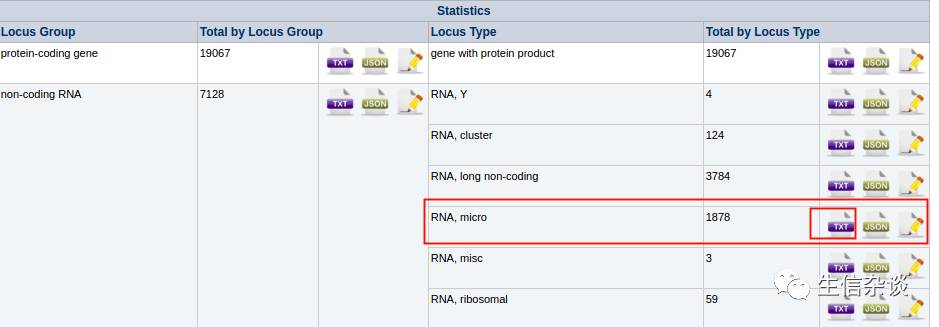
下载txt格式的文件后打开是这样的:
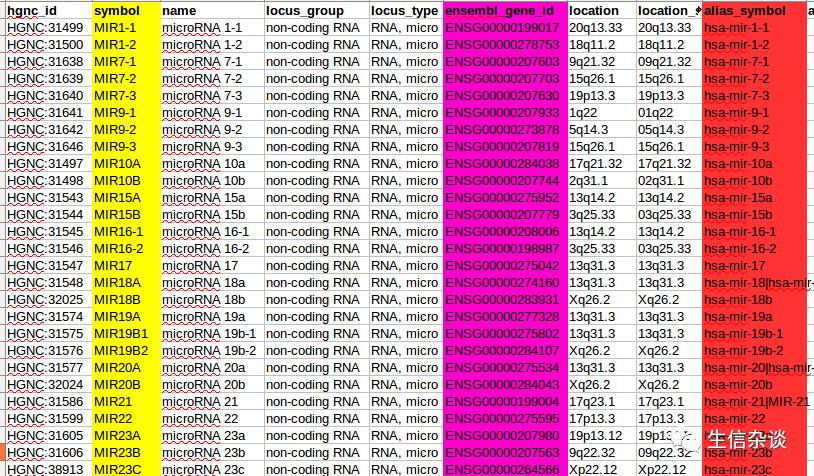
上色的三列是本次ID转换我们所需要的,我们将根据上面这个表把
GTEx的symbol
和
ensemble ID
转为和GTEx中的
alias
,但我们仔细看HGNC中的
ensembel_gene_id
与GTEx里的有点不同,GTEx里的每个ID后面还有
小数点
,而HGNC的并没有,这是由于ensemble版本造成的,只需看小数点前面的ID就可以了,
小数点前面ID的一致就是同一个miRNA
.这个问题也会在TCGA基因表达谱ID转换时出现,同理只需管小数点前面的ID就可以了.
R实现转ID的过程比较方便,R代码如下:
rm(list=ls())
library(data.table)
library(stringr)
GTEx_liver_path"./GTEx_Analysis_v6p_RNA_Liver2.tsv"
GTEx_reads"\t",header =T)
names(GTEx_reads)[1:2]"ensembl_gene_id","symbol")
GTEx_reads[,ensembl_gene_id:=str_split_fixed(GTEx_reads$ensembl_gene_id,"[.]",2)[,1]]
HGNC_mir_path"../HGNC/RNA_micro.txt"
HGNC_mir"\t",header =T)
HGNC_mir"ensembl_gene_id","symbol","alias_symbol")]
上面的代码完成了读入文件和统一列名和去除GTEx
ensemb_gene_id
中的小数点.但在合并时我们有三种选择:
GTEx_reads_merged_id"ensembl_gene_id"))
GTEx_reads_merged_symbol"symbol"))
GTEx_reads_merged_both"ensembl_gene_id","symbol"))
-
只匹配
ensembl_gene_id
进行合并;
-
只匹配
symbol
进行合并;
-
匹配
ensembl_gene_id
和
symbol
进行合并;
三种方法都尝试了下发现差异很大:
a1a2a3
结果发现通过两个条件(
ensemb_gene_id
和
symbol
)合并得到的结果最少(
1113条
),只通过
symbol
得到的最多(
1249
条).但不管是
1113
还是
1249
条,都远少于miRNA总数
1881
,ID转换率低于
66.4%
.我搜了几条发现这应该是由于
版本差异
导致的.GTE应该用的是
GRCh37.p13
,而HGNC上面是最新版的
GRCh38.p10
.我们通过一个两个例子来说明.
同一个ensemble ID:
ENSG00000211563
在不同版本
gencode
的结果
:
|
Source
|
Ensembl_gnee_id
|
Symbol
|
|
Gencode_v19
|
ENSG00000211563.2
|
MIR338
|
|
Gencode_v23
|
ENSG00000211563.3
|
AC115099.1
|
|
Gencode_v26
|
ENSG00000211563.4
|
MIR3065
|
|
HGNC
|
ENSG00000211563
|
MIR3065
|
|
GTEx
|
ENSG00000211563.2
|
MIR338
|
同一个
miRNA
hsa-mir-338
在不同版本
gencode
的结果
:
|
Source
|
Symbol
|
Ensembl_gnee_id
|
|
Gencode_v19
|
MIR338
|
ENSG00000211563.2
|
|
Gencode_v23
|
None
|
None
|
|
Gencode_v26
|
MIR338
|
ENSG00000283604.1
|
|
HGNC
|
MIR338
|
ENSG00000283604
|
|
GTEx
|
MIR338
|
ENSG00000211563.2
|
可以看出同一个
ensembl_gene_id
在三个不同版本的
Gencode
所对应的Symbol中都是不一样的,而同一个
Symblol
在不同版本的
Gencode
中的
Ensembl_gene_id
也不同.
但还是可以发现:
HGNC
与最新的
Gencode
保持一致,而
GTEx
与
Gencode_v19
版本保持一致,也就是说,ID转换率低是由于新旧版本不匹配导致的,如果要在新旧版本间选个标准,最好还是选择新版本,因为新旧版本的差异是由于新版本对就版的更正所导致的.也就是说新版本对”基因”的注释更加准确,但转换率会比较低.














