在2015年备受瞩目的测序黑马当属10X Genomics无疑,通过将来自相同DNA片段(10-100kb)的reads加上相同的barcode,然后在illumina平台上进行测序,最后通过barcode将read划分到每一个DNA片段上,最后以每一个DNA片段进行单独组装,获得相对较长的的DNA片段。其原理类似于构建BAC 文库,用Illumina测BAC,然后进行单个BAC 组装。但是其存在一个barcode不一定对应一个DNA片段的问题,唯一性相比较BAC,肯定会逊色不少;目前该技术受10X Genomics垄断,国内公司有代理此公司产品。
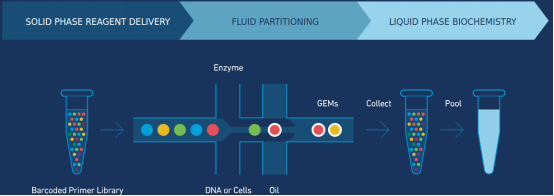
图1 10X Genomics的文库构建
首先说一下Hi-C辅助组装的原理:那细胞核内包含同源染色体在内每条染色体的都占据着一个独特区域,称为染色体疆域(Territory),这就导致基因组各区段(Locus)在同一染色体上的交互频率高于不同染色体的交互频率,染色体内部不同Locus之间的交互频率与Locus之间的线性距离一般近似服从幂次定律(Power Law)。因此通过Hi-C就可以将不同的染色体进行划分以及同一条染色体排序。

图2
Chicago技术原理图
Chicago技术可以说是一种体外Hi-C技术,主要解决Hi-C技术中不同染色体端粒区域存在交互带来的组装问题。他的核心点是通过人工构造一定长度的染色体片段(各片段长度分布信息见图3),利用同一个片段上的交互信号强,不同片段的交互信号弱,从而也实现了单DNA片段组装,提升Scaffold组装指标,但不能组装到染色体水平。相对于10X,他也是单DNA片段组装,只是利用的是片段之间交互信息作为Link的关键信息,他的文库DNA片段长度相对于10X有了较大的提高。
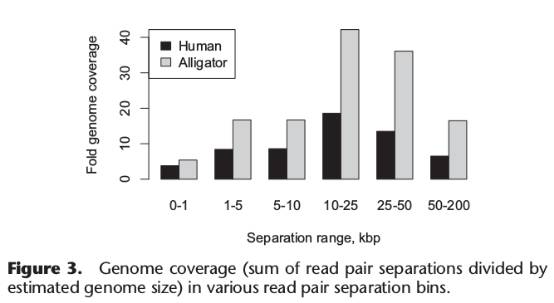
图3
Chicago不同插入片段长度覆盖基因组的深度
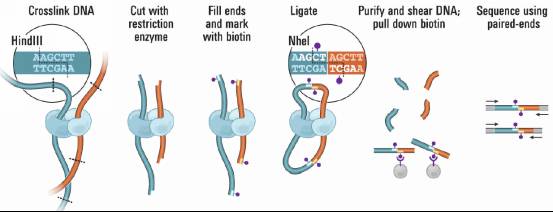
图4
Hi-C
技术原理图
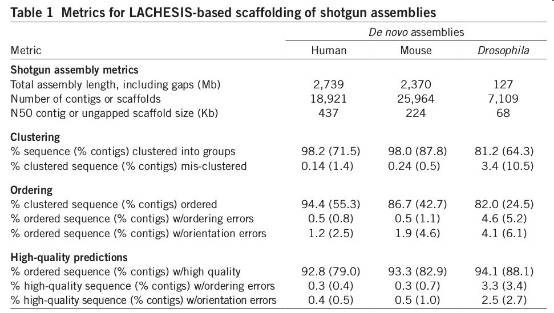
2013年发表在Nature biotechnology上人、小鼠、果蝇基因组首次使用Hi-C成功实现了二代组装基因组序列挂到了染色体水平。其中人与小鼠染色体挂载率超过了98%,准确性超过99%
相信好多人质疑Hi-C的超强能力,但是2017年3月23日Science发表利用Hi-C完成传播寨卡病毒和西尼罗病毒的两种蚊虫的3条染色体序列的组装近;2017年4月27日Nature发表利用Hi-C完成大麦基因组的组装,并阐明大麦染色体的空间结构,这两篇文献发表进一步证实Hi-C技术是靠谱的。
纵观三种技术,均是基于illumina测序的提升Scaffold水平的技术。10X和Chicago技术均已被国外公司技术垄断,唯独Hi-C技术是一种共享技术,人人可用(不用花大价钱去国外买使用权),且可以将基因组组装到染色体水平。
那么到底三种技术谁会赢呢?欢迎给出您的意见。
1.Weisenfeld N I, Kumar V, Shah P, et al. Direct determination of diploid genome sequences.[J].
Genome Research
, 2017, 27(5):757.
2.Putnam N H, O'Connell B L, Stites J C, et al. Chromosome-scale shotgun assembly using an in vitro method for long-range linkage.[J].
Genome Research
, 2016, 26(3):342.
3. Burton, J.N., et al., Chromosome-scale scaffolding of
de novo genome
assemblies based on chromatin interactions.
Nat Biotechnol
, 2013. 31(12): p. 1119-25.
4. Dudchenko O, Batra SS, Omer AD, et al. De novo assembly of the Aedes aegypti genome using Hi-C yields chromosome-length scaffolds.[J].
Science
, 2017.
5. Mascher M, Gundlach H, Himmelbach A, et al. A chromosome conformation capture ordered sequence of the barley genome[J].
Nature
, 2017, 544(7651): 427-433.
欢迎关注生信人






