题记:这篇读书笔记我们将展示用R实现两种非线性技术:K最近邻(KNN)与支持向量机(SVM)。本文参考Cory Lesmeister博士主编《Mastering Machine Learning with R》(第2版)一书。数据来自R语言MASS包自带Pima.tr和Pima.te数据集,代码来自Cory Lesmeister博士主编《Mastering Machine Learning with R》这本书配套的代码,笔者对部分代码进行修改。本文是个人的读书笔记,仅用于学习交流使用,我只是一个知识的搬运工。
1. 案例分析
在接下来的案例研究中,我们在同一个数据集上应用KNN和SVM,这可以比较解决同一问题的R代码和学习方法。我们从KNN开始,还会花一些时间对混淆矩阵进行深入研究,并对评价模型正确率的各个统计量进行比较。
我们要研究的数据来自美国国家糖尿病消化病肾病研究所,这个数据集包括532个观测,8个输入特征以及1个二值结果变量(Yes/No)。这项研究中的患者来自美国亚利桑那州中南部,是皮玛族印第安人的后裔。数据显示,在过去的30年中,科学家已经通过研究证明肥胖是引发糖尿病的重要因素。选择皮玛印第安人进行这项研究是因为,半数成年皮玛印第安人患有糖尿病。而这些患有糖尿病的人中,有95%超重。研究仅限于成年女性,病情则按照世界卫生组织的标准进行诊断为2型糖尿病。这种糖尿病的患者胰腺功能并未完全丧失,还可以产生胰岛素,因此又称“非胰岛素依赖型”糖尿病。
我们的任务是研究那些糖尿病患者,并对这个人群中可能导致糖尿病的风险因素进行预测。久坐不动的生活方式和高热量的饮食习惯使得糖尿病已经成为美国的流行病。根据美国糖尿病协会的数据,2010年,糖尿病成为美国排名第七的致死疾病,这个结果还不包括那些未被诊断出来的病例。糖尿病还会大大增加其他疾病的发病概率,比如高血压、血脂异常、中风、眼疾和肾脏疾病。糖尿病及其并发症的医疗成本非常巨大,据估计,美国2012年糖尿病治疗总成本大约为4900亿美元。要想知道这个问题的更多背景知识,请参考美国糖尿病协会网站http://diabetes.org/diabetes-basics/statistics/.
数据集包含了532位女性患者的信息,存储在两个数据框中。数据集变量如下:
(1)npreg:怀孕次数。
(2)glu:血糖浓度,由口服葡萄糖耐量测试给出。
(3)bp:舒张压(单位为mm Hg)。
(4)skin:三头肌皮褶厚度(单位为mm)。
(5)bmi:身体质量指数。
(6)ped:糖尿病家族影响因素。
(7)age:年龄。
(8)type:是否患有糖尿病(是/否)。
数据集包含在MASS这个R包中,一个数据框是Pima.tr,另一个数据框的是Pima.te。我们不将它们分别作为训练集和测试集,而是将其合在一起,然后建立自己的训练集和测试集,目的是学习如何使用R完成这样的任务。首先加载下面的程序包,练习时会用到它们:
library(class) #k-nearest neighbors
library(kknn) #weighted k-nearest neighbors
library(e1071) #SVM
library(caret) #select tuning parameters
## Loading required package: lattice
## Loading required package: ggplot2
## Attaching package: 'caret'
## The following object is masked from 'package:kknn':
## contr.dummy
library(MASS) # contains the data
library(reshape2) #assist in creating boxplots
library(ggplot2) #create boxplots
library(kernlab) #assist with SVM feature selection
## Attaching package: 'kernlab'
## The following object is masked from 'package:ggplot2':
## alpha
2. 数据准备
现在加载数据集并检查其结构,确保结构相同。从Pima.tr开始,如下所示:
data(Pima.tr)
str(Pima.tr)
## 'data.frame': 200 obs. of 8 variables:
## $ npreg: int 5 7 5 0 0 5 3 1 3 2 ...
## $ glu : int 86 195 77 165 107 97 83 193 142 128 ...
## $ bp : int 68 70 82 76 60 76 58 50 80 78 ...
## $ skin : int 28 33 41 43 25 27 31 16 15 37 ...
## $ bmi : num 30.2 25.1 35.8 47.9 26.4 35.6 34.3 25.9 32.4 43.3 ...
## $ ped : num 0.364 0.163 0.156 0.259 0.133 ...
## $ age : int 24 55 35 26 23 52 25 24 63 31 ...
## $ type : Factor w/ 2 levels "No","Yes": 1 2 1 1 1 2 1 1 1 2 ...
data(Pima.te)
str(Pima.te)
## 'data.frame': 332 obs. of 8 variables:
## $ npreg: int 6 1 1 3 2 5 0 1 3 9 ...
## $ glu : int 148 85 89 78 197 166 118 103 126 119 ...
## $ bp : int 72 66 66 50 70 72 84 30 88 80 ...
## $ skin : int 35 29 23 32 45 19 47 38 41 35 ...
## $ bmi : num 33.6 26.6 28.1 31 30.5 25.8 45.8 43.3 39.3 29 ...
## $ ped : num 0.627 0.351 0.167 0.248 0.158 0.587 0.551 0.183 0.704 0.263 ...
## $ age : int 50 31 21 26 53 51 31 33 27 29 ...
## $ type : Factor w/ 2 levels "No","Yes": 2 1 1 2 2 2 2 1 1 2 ...
检查数据集结构之后,我们确信可以把两个数据框合成一个。这非常容易,使用rbind()函数即可,它的功能是绑定行并追加数据。如果你的每个数据框都有相同的观测并想追加特征,则应该使用cbind()函数按列绑定数据。给你的新数据框命名也非常简单,可以使用语法:newdata=rbind(data frame1, data fram2)。代码如下:
pima
## 'data.frame': 532 obs. of 8 variables:
## $ npreg: int 5 7 5 0 0 5 3 1 3 2 ...
## $ glu : int 86 195 77 165 107 97 83 193 142 128 ...
## $ bp : int 68 70 82 76 60 76 58 50 80 78 ...
## $ skin : int 28 33 41 43 25 27 31 16 15 37 ...
## $ bmi : num 30.2 25.1 35.8 47.9 26.4 35.6 34.3 25.9 32.4 43.3 ...
## $ ped : num 0.364 0.163 0.156 0.259 0.133 ...
## $ age : int 24 55 35 26 23 52 25 24 63 31 ...
## $ type : Factor w/ 2 levels "No","Yes": 1 2 1 1 1 2 1 1 1 2 ...
通过箱线图进行探索性分析。为此,要使用结果变量“type”作为ID变量的值。和Logistic回归一样,melt()函数会融合数据并准备好用于生成箱线图的数据框。我们将新的数据框命名为pima.melt,如下所示:
pima.melt "type")
使用ggplot包对箱线图进行布局是非常有效的。在ggplot()函数中,我们要指定使用的数据、x变量和y变量、统计图类型,还要说明生成的是两列统计图。在下面的代码中,将响应变量作为aes()函数中的x,响应变量的值作为y,然后使用geom_boxplot()函数生成统计图。最后,使用facet_wrap()函数将统计图分两列显示:
ggplot(data = pima.melt, aes(x = type, y = value)) +
geom_boxplot() + facet_wrap(~ variable, ncol = 2)
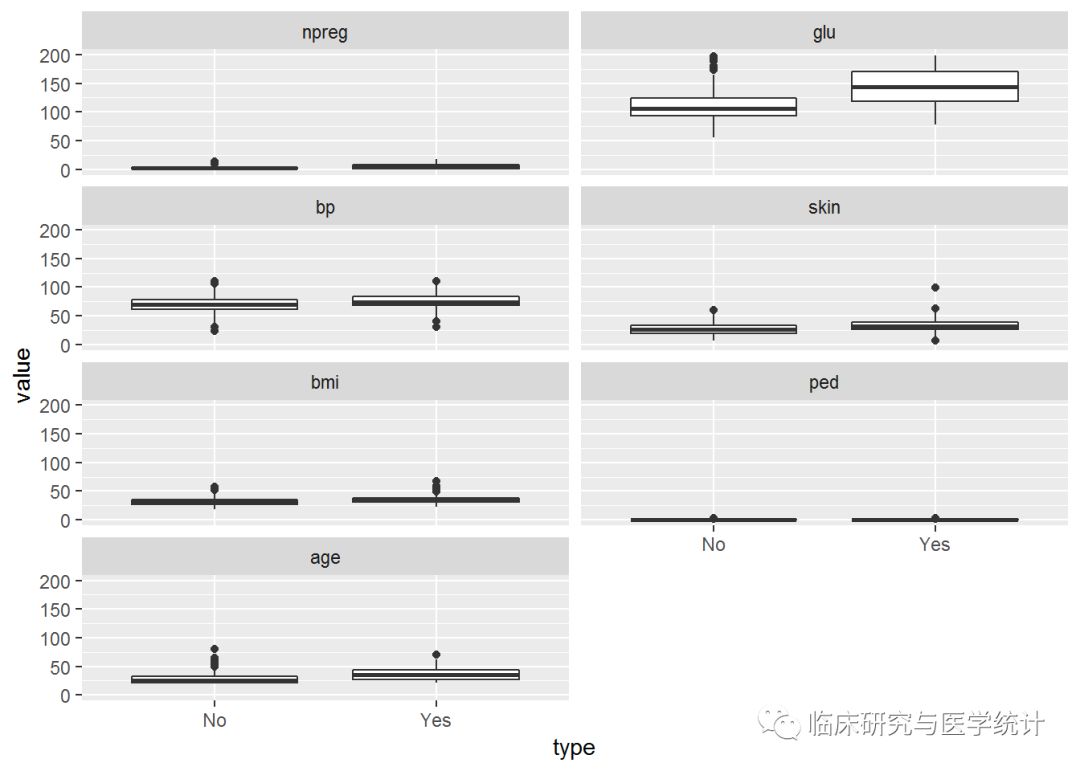
这张图很有趣,因为很难从中发现任何明显区别,除了血糖浓度(glu)。正如你预想的那样,如果患者的血糖浓度快速升高,那么一般可以确诊为糖尿病。这里最大的问题是,不同统计图的单位不同,但却共用一个Y轴。对数据进行标准化处理并重新做图,可以解决这个问题,并生成更有意义的统计图。
R有一个内建函数scale(),可以将数据转换为均值为0、标准差为1的标准形式。我们把经过标准化处理后的数据放到pima.scale新数据框,要对所有特征进行转换,只留下响应变量type。强调一下,进行KNN时,使所有特征具有同样的测量标准是很重要的。也就是说,要对数据进行标准化处理,使其均值为0,标准差为1。如果不进行标准化,那么对最近邻的距离计算就会出现错误。如果一个特征的测量标准是1
100,那么和另一个测量标准为1
10的特征相比,肯定会对结果有更大的影响。请记住,如果你对一个数据框应用了scale()函数,它就自动变成一个矩阵。使用as.data.frame()函数,将其重新变回数据框,如下所示:
pima.scale 8]))#scale.pima = as.data.frame(scale(pima[,1:7], byrow=FALSE)) #do not create own function
str(pima.scale)
## 'data.frame': 532 obs. of 7 variables:
## $ npreg: num 0.448 1.052 0.448 -1.062 -1.062 ...
## $ glu : num -1.13 2.386 -1.42 1.418 -0.453 ...
## $ bp : num -0.285 -0.122 0.852 0.365 -0.935 ...
## $ skin : num -0.112 0.363 1.123 1.313 -0.397 ...
## $ bmi : num -0.391 -1.132 0.423 2.181 -0.943 ...
## $ ped : num -0.403 -0.987 -1.007 -0.708 -1.074 ...
## $ age : num -0.708 2.173 0.315 -0.522 -0.801 ...
pima.scale$type "type")
ggplot(data=pima.scale.melt, aes(x = type, y = value)) +
geom_boxplot() + facet_wrap(~ variable, ncol = 2)
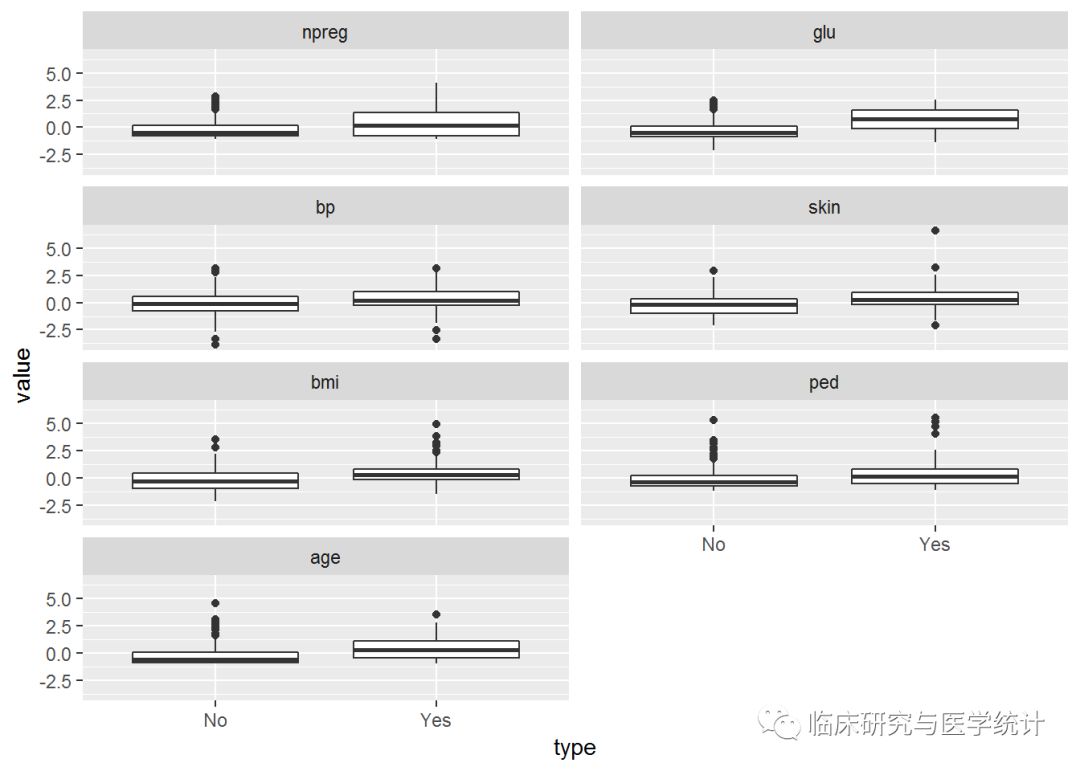
对特征进行标准化之后,箱线图好看多了。除了血糖浓度之外,可以看出其他特征也随着type发生变化,特别是age。将数据分为训练集和测试集之前,先使用R中的cor()函数查看相关性。这个函数不会生成皮尔逊相关性统计图,而生成一个矩阵:
cor(pima.scale[-8])
## npreg glu bp skin bmi ped
## npreg 1.000000000 0.1253296 0.204663421 0.09508511 0.008576282 0.007435104
## glu 0.125329647 1.0000000 0.219177950 0.22659042 0.247079294 0.165817411
## bp 0.204663421 0.2191779 1.000000000 0.22607244 0.307356904 0.008047249
## skin 0.095085114 0.2265904 0.226072440 1.00000000 0.647422386 0.118635569
## bmi 0.008576282 0.2470793 0.307356904 0.64742239 1.000000000 0.151107136
## ped 0.007435104 0.1658174 0.008047249 0.11863557 0.151107136 1.000000000
## age 0.640746866 0.2789071 0.346938723 0.16133614 0.073438257 0.071654133
## age
## npreg 0.64074687
## glu 0.27890711
## bp 0.34693872
## skin 0.16133614
## bmi 0.07343826
## ped 0.07165413
## age 1.00000000
有两对变量之间具有相关性:npreg/age和skin/bmi。如果能够正确训练模型,并能调整好超参数,那么多重共线性对于这些方法通常都不是问题。我认为我们已经做好了建立训练集和测试集的准备,但是我建议一定要先检查响应变量中Yes和No的比例。确保数据划分平衡是非常重要的,如果某个结果过于稀疏,就会导致问题,可能引起分类器在优势类和劣势类之间发生偏离。对于不平衡的判定没有一个固定的规则。一个比较好的经验法则是,结果中的比例至少应该达到2∶1(He与Wa,2013)。
table(pima.scale$type)
## No Yes
## 355 177
比例为2∶1,现在可以建立训练集和测试集了。使用我们常用的语法,划分比例为70/30,如下所示:
set.seed(502)
ind 2, nrow(pima.scale), replace = TRUE, prob = c(0.7, 0.3))
train 1, ]
test 2, ]
str(train)
## 'data.frame': 385 obs. of 8 variables:
## $ npreg: num 0.448 0.448 -0.156 -0.76 -0.156 ...
## $ glu : num -1.42 -0.775 -1.227 2.322 0.676 ...
## $ bp : num 0.852 0.365 -1.097 -1.747 0.69 ...
## $ skin : num 1.123 -0.207 0.173 -1.253 -1.348 ...
## $ bmi : num 0.4229 0.3938 0.2049 -1.0159 -0.0712 ...
## $ ped : num -1.007 -0.363 -0.485 0.441 -0.879 ...
## $ age : num 0.315 1.894 -0.615 -0.708 2.916 ...
## $ type : Factor w/ 2 levels "No","Yes": 1 2 1 1 1 2 2 1 1 1 ...
str(test)
## 'data.frame': 147 obs. of 8 variables:
## $ npreg: num 0.448 1.052 -1.062 -1.062 -0.458 ...
## $ glu : num -1.13 2.386 1.418 -0.453 0.225 ...
## $ bp : num -0.285 -0.122 0.365 -0.935 0.528 ...
## $ skin : num -0.112 0.363 1.313 -0.397 0.743 ...
## $ bmi : num -0.391 -1.132 2.181 -0.943 1.513 ...
## $ ped : num -0.403 -0.987 -0.708 -1.074 2.093 ...
## $ age : num -0.7076 2.173 -0.5217 -0.8005 -0.0571 ...
## $ type : Factor w/ 2 levels "No","Yes": 1 2 1 1 2 1 2 1 1 1 ...
一切就绪,下一步就是建立预测模型并进行评价。我们从KNN开始。
3. KNN建模
我们在前面提到过,使用KNN建模关键的一点就是选择最合适的参数(k)。在确定k值方面,caret包又可以大显身手了。先建立一个供实验用的输入网格,k值从2到20,每次增加1。使用expand.grid()和seq()函数可以轻松实现。在caret包中,作用于KNN函数的参数非常简单直接,就是.k:
grid1 2, 20, by = 1))
选择参数时,还是使用交叉验证。先建立一个名为control的对象,然后使用caret包中的trainControl()函数,如下所示:
control = trainControl(method = "cv")
现在,使用train()函数建立计算最优k值的对象,train()函数也在caret包中。别忘了在进行任何随机抽样之前,都要先设定随机数种子:
set.seed(123)
使用train()函数建立对象时,需要指定模型公式、训练数据集名称和一个合适的方法。模型公式和以前一样 – y ~ x,方法就是knn。这些参数设定之后,R代码就可以建立对象并计算最优k值了,如下所示:
knn.train "knn",
trControl = control,
tuneGrid = grid1)
knn.train
## k-Nearest Neighbors
##
## 385 samples
## 7 predictor
## 2 classes: 'No', 'Yes'
##
## No pre-processing
## Resampling: Cross-Validated (10 fold)
## Summary of sample sizes: 347, 346, 347, 347, 346, 347, ...
## Resampling results across tuning parameters:
##
## k Accuracy Kappa
## 2 0.7324831 0.3479337
## 3 0.7609615 0.4052636
## 4 0.7610324 0.4144867
## 5 0.7584649 0.3965911
## 6 0.7537348 0.3844074
## 7 0.7666262 0.4164286
## 8 0.7455094 0.3629167
## 9 0.7535324 0.3798513
## 10 0.7508333 0.3747918
## 11 0.7482051 0.3617260
## 12 0.7587955 0.3930875
## 13 0.7613596 0.3912383
## 14 0.7664912 0.4039438
## 15 0.7719534 0.4147952
## 16 0.7691228 0.4094041
## 17 0.7770816 0.4219200
## 18 0.7745175 0.4184854
## 19 0.7665587 0.4007147
## 20 0.7797807 0.4373146
##
## Accuracy was used to select the optimal model using the largest value.
## The final value used for the model was k = 20.
除了得到k = 17这个结果之外,我们在输出的表格中还可以看到正确率和Kappa统计量的信息,以及交叉验证过程中产生的标准差。正确率告诉我们模型正确分类的百分比。Kappa又通常用于测量两个分类器对观测值分类的一致性。Kappa可以使我们对分类问题的理解更加深入,它对正确率进行了修正,去除了仅靠偶然性(或随机性)获得正确分类的因素。计算这个统计量的公式是Kappa = (一致性百分比 - 期望一致性百分比)/(1 - 期望一致性百分比)。
一致性百分比是分类器的分类结果与实际分类相符合的程度(就是正确率),期望一致性百分比是分类器靠随机选择获得的与实际分类相符合的程度。Kappa统计量的值越大,分类器的分类效果越好,Kappa为1时达到一致性的最大值。下面将模型应用到测试数据集上,通过一个完整的例子说明如何计算正确率和Kappa。
使用class包中的knn()函数实现。要使用这个函数,至少需要指定4个参数:训练数据、测试数据、训练集中的正确标记、k值。生成一个名为knn.test的对象,看看效果如何:
knn.test 8], test[, -8], train[, 8], k = 17)
对象生成之后,检查混淆矩阵,算出正确率和Kappa:
table(knn.test, test$type)
## knn.test No Yes
## No 77 26
## Yes 16 28
正确率的计算非常简单,用分类正确的观测数除以观测总数即可:
(77+28)/147
## [1] 0.7142857
正确率为71%,这比我们在训练数据上得到的正确率(约80%)稍低一些。
下面看看如何使用代码计算Kappa统计量:
prob.agree 77+28
)/147
prob.chance 77+26)*(77+16)+(16+28)*(26+28))/(147*147)
prob.chance
## [1] 0.5532417
kappa 1 - prob.chance)
kappa
## [1] 0.3604723
Kappa统计量的值为0.36,和我们在训练数据集中得到的一样。以上Kappa值的计算方法很繁琐,推荐使用fmsb包中Kappa.test()函数可快速实现Kappa计算,代码如下:
library(fmsb)
mytable0.95)
## $Result
##
## Estimate Cohen's kappa statistics and test the null hypothesis
## that the extent of agreement is same as random (kappa=0)
##
## data: mytable
## Z = 3.9274, p-value = 4.293e-05
## 95 percent confidence interval:
## 0.1970096 0.5239351
## sample estimates:
## [1] 0.3604723
##
## $Judgement
## [1] "Fair agreement"
我们的Kappa只是“Fair agreement”,在测试集上的正确率仅比70%高一点,所以应该看看是否可以使用加权最近邻法得到更好的结果。加权最近邻法提高了离观测更近的邻居的影响力,降低了远离观测的邻居的影响力。观测离空间点越远,对它的影响力的惩罚就越大。要使用加权最近邻法,需要kknn包中的train.kknn()函数来选择最优的加权方式。
train.kknn()函数使用我们前面介绍过的LOOCV选择最优参数,比如最优的K最近邻数量、二选一的距离测量方式,以及核函数。
在前面的讨论中,不加权的K最近邻算法使用的是欧式距离。在kknn包中,除了欧式距离,还可以选择两点坐标差的绝对值之和。如果要使用这种距离计算方式,需要指定闵可夫斯基距离参数。
有多种方法可以对距离进行加权。我们要使用的kknn包中有10种不同的加权方式,不加权也是其中之一。它们是:retangular(不加权)、triangular、epanechnikov、biweight、triweight、consine、inversion、gaussian、rank和optimal。对这些加权技术的全面介绍请参考Hechenbichler K.与Schliep K.P.(2004)。
为简单起见,我们集中讨论其中两种:triangular和epanechnikov。赋予权重之前,算法对所有距离进行标准化处理,使它们的值都在0和1之间。triangular加权方法先算出1减去距离的差,再用差作为权重去乘这个距离。epanechnikov加权方法是用3/4乘以(1 - 距离的平方)。为了方便比较,我们把这两种加权方法和标准的不加权方法放在一起实现。
先指定随机数种子,然后使用kknn()函数建立训练集对象,这个函数要求指定的参数有:k值的最大值kmax、距离distance(1表示绝对值距离,2表示欧氏距离)、核函数。在我们的模型中,kmax设定为25,distance为2:
set.seed(123)
kknn.train 25, distance = 2,
kernel = c("rectangular", "triangular", "epanechnikov"))
plot(kknn.train)
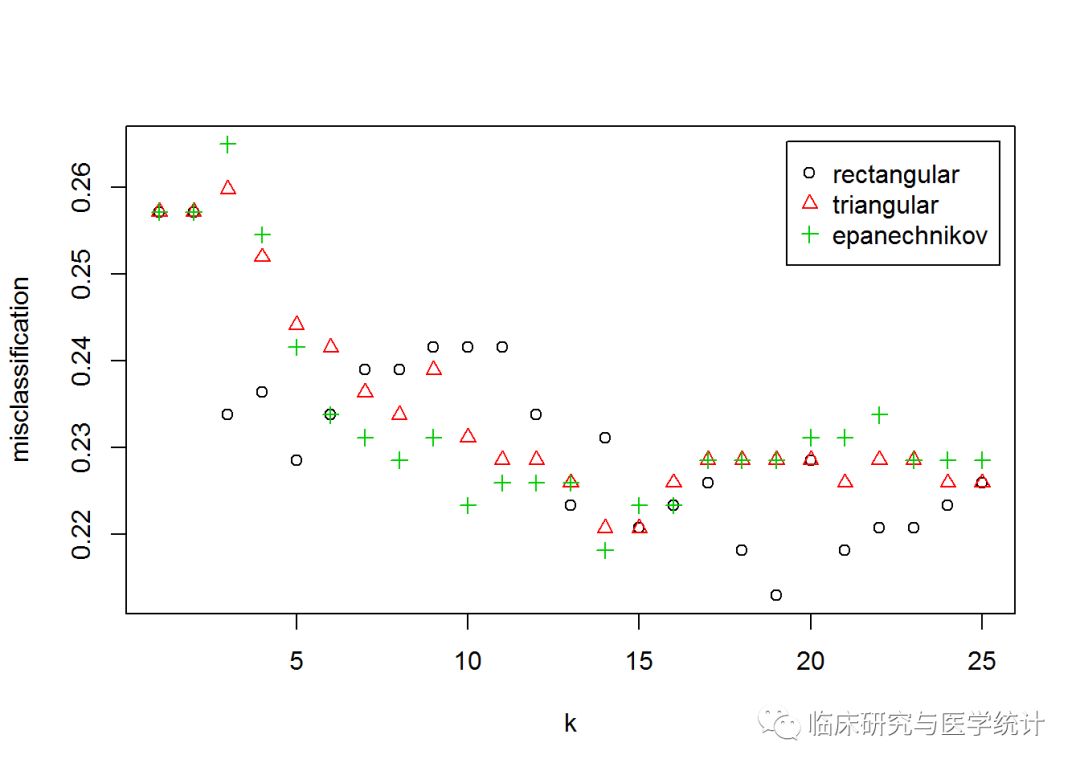
图中X轴表示的是k值,Y轴表示的是核函数误分类观测百分比。令人惊讶的是,不加权分类方式(rectangular)在k = 19的时候表现最好。可以调用对象看看分类误差和最优参数,如下所示:
kknn.train
## Call:
## train.kknn(formula = type ~ ., data = train, kmax = 25, distance = 2, kernel = c("rectangular", "triangular", "epanechnikov"))
##
## Type of response variable: nominal
## Minimal misclassification: 0.212987
## Best kernel: rectangular
## Best k: 19
从上面的数据可以看出,给距离加权不能提高模型在训练集上的正确率。而且从下面的代码可以看出,它同样不能提高测试集上的正确率:
kknn.pred
## kknn.pred No Yes
## No 76 27
## Yes 17 27
我们还可以试验其他加权方法,但其他方法的结果并不比这些更好。我们对KNN的讨论到此为止。
4. SVM建模
使用e1071包构建SVM模型,先从线性支持向量分类器开始,然后转入非线性模型。e1071包中有一个非常好的用于SVM的函数——tune.svm(),它可以帮助我们选择调优参数及核函数。tune.svm()使用交叉验证使调优参数达到最优。我们先建立一个名为linear.tune的对象,然后使用summary()函数看看其中的内容。如下所示:
set.seed(123)
linear.tune "linear",
cost = c(0.001, 0.01, 0.1, 1, 5, 10))
summary(linear.tune)
## Parameter tuning of 'svm':
##
## - sampling method: 10-fold cross validation
##
## - best parameters:
## cost
## 0.01
##
## - best performance: 0.2052632
##
## - Detailed performance results:
## cost error dispersion
## 1 1e-03 0.3192308 0.04875705
## 2 1e-02 0.2052632 0.05381760
## 3 1e-01 0.2128880 0.06712176
## 4 1e+00 0.2128880 0.06712176
## 5 5e+00 0.2128880 0.06712176
## 6 1e+01 0.2128880 0.06712176
对于这些数据,最优成本函数cost是1,这时的误分类误差率差不多为21%。我们在测试集上进行预测和检验,使用predict()函数,指定newdata=test:
best.linear
## tune.test No Yes
## No 82 24
## Yes 11 30
(82+30)/147
## [1] 0.7619048
线性支持向量分类器在训练集和测试集上表现得都比KNN稍好一些。e1071包中有一个用于SVM的非常好的函数——tune.svm(),它可以帮助我们选择调优参数或核函数。现在看看非线性模型能否表现得更好,依然使用交叉验证选择调优参数。我们要试验的第一个核函数是多项式核函数,需要调整优化两个参数:多项式的阶(degree)与核系数(coef0)。设定多项式的阶是3、4和5,核系数从0.1逐渐增加到4,如下所示:
set.seed(123)
poly.tune "polynomial",
degree = c(3, 4, 5),
coef0 = c(0.1, 0.5, 1, 2, 3, 4))
summary(poly.tune)
## Parameter tuning of 'svm':
##
## - sampling method: 10-fold cross validation
##
## - best parameters:
## degree coef0
## 3 0.1
##
## - best performance: 0.2310391
##
## - Detailed performance results:
## degree coef0 error dispersion
## 1 3 0.1 0.2310391 0.06769286
## 2 4 0.1 0.2467611 0.08006379
## 3 5 0.1 0.2416329 0.07459147
## 4 3 0.5 0.2466262 0.09248245
## 5 4 0.5 0.2674764 0.07238427
## 6 5 0.5 0.2439946 0.06582874
## 7 3 1.0 0.2519568 0.09058895
## 8 4 1.0 0.2725371 0.08483943
## 9 5 1.0 0.2674764 0.09422521
## 10 3 2.0 0.2545884 0.08789505
## 11 4 2.0 0.2804318 0.10825010
## 12 5 2.0 0.2750337 0.09683544
## 13 3 3.0 0.2493252 0.08625914
## 14 4 3.0 0.2674089 0.11792957
## 15 5 3.0 0.2879892 0.09289320
## 16 3 4.0 0.2414980 0.08131609
## 17 4 4.0 0.2569501 0.10577366
## 18 5 4.0 0.2778003 0.07678685
模型选择的多项式阶数为3,核系数为0.1。和线性SVM一样,可以用这些参数在测试集上进行预测,如下所示:
best.poly
## poly.test No Yes
## No 81 28
## Yes 12 26
(81 + 26) / 147
## [1] 0.7278912
这个模型的表现还不如线性模型。下面测试径向基核函数,此处只需找出一个参数gamma,在0.1~4中依次检验。如果gamma过小,模型就不能解释决策边界的复杂性;如果gamma过大,模型就会严重过拟合。
set.seed(123)
rbf.tune "radial",
gamma = c(0.1, 0.5, 1, 2, 3, 4))
summary(rbf.tune)
## Parameter tuning of 'svm':
##
## - sampling method: 10-fold cross validation
##
## - best parameters:
## gamma
## 0.5
##
## - best performance: 0.2284076
##
## - Detailed performance results:
## gamma error dispersion
## 1 0.1 0.2286775 0.08171824
## 2 0.5 0.2284076 0.08954819
## 3 1.0 0.2647099 0.06210439
## 4 2.0 0.3269906 0.05295757
## 5 3.0 0.3218623 0.04602815
## 6 4.0 0.3192308 0.04875705
最优的gamma值是0.5,这种模型的表现看上去也不比其他SVM模型好多少。我们还是要在测试集上进行验证,和前面一样:
best.rbf
## rbf.test No Yes
## No 73 33
## Yes 20 21
(73+21)/147
## [1] 0.6394558
表现很差!提高性能的最后一招只能是kernel=“sigmoid”了。需要找出两个参数 – gamma和核系数(coef0):
set.seed(123)
sigmoid.tune "sigmoid",
gamma = c(0.1, 0.5, 1, 2, 3, 4),
coef0 = c(0.1, 0.5, 1, 2, 3, 4))
summary(sigmoid.tune)
## Parameter tuning of 'svm':
##
## - sampling method: 10-fold cross validation
##
## - best parameters:
## gamma coef0
## 0.1 2
##
## - best performance: 0.2080972
##
## - Detailed performance results:
## gamma coef0 error dispersion
## 1 0.1 0.1 0.2126856 0.06635048
## 2 0.5 0.1 0.2751687 0.06698411
## 3 1.0 0.1 0.3061404 0.07336713
## 4 2.0 0.1 0.2803644 0.06450517
## 5 3.0 0.1 0.3242915 0.08207266
## 6 4.0 0.1 0.2722672 0.10958642
## 7 0.1 0.5 0.2518893 0.06655774
## 8 0.5 0.5 0.2962888 0.06077742
## 9 1.0 0.5 0.2986505 0.08203135
## 10 2.0 0.5 0.2860999 0.06320568
## 11 3.0 0.5 0.2986505 0.06887749
## 12 4.0 0.5 0.2701080 0.08935013
## 13 0.1 1.0 0.2728070 0.10364945
## 14 0.5 1.0 0.3219973 0.10655089
## 15 1.0 1.0 0.2802969 0.06933138
## 16 2.0 1.0 0.2857625 0.07804622
## 17 3.0 1.0 0.3221323 0.08095616
## 18 4.0 1.0 0.2986505 0.06707196
## 19 0.1 2.0 0.2080972 0.04725082
## 20 0.5 2.0 0.3659919 0.09795326
## 21 1.0 2.0 0.3088394 0.06199432
## 22 2.0 2.0 0.2806343 0.08834537
## 23 3.0 2.0 0.2827260 0.07186226
## 24 4.0 2.0 0.3037112 0.07983245
## 25 0.1 3.0 0.3192308 0.04875705
## 26 0.5 3.0 0.3660594 0.09587096
## 27 1.0 3.0 0.3632928 0.09922022
## 28 2.0 3.0 0.3091093 0.06709251
## 29 3.0 3.0 0.3117409 0.08931896
## 30 4.0 3.0 0.2858974 0.08992186
## 31 0.1 4.0 0.3192308 0.04875705
## 32 0.5 4.0 0.3533738 0.07987591
## 33 1.0 4.0 0.3711876 0.09756838
## 34 2.0 4.0 0.2780027 0.05037333
## 35 3.0 4.0 0.2830634 0.08721915
## 36 4.0 4.0 0.2982456 0.06231253
误差率和线性模型基本一样。现在只要看看能否在测试集上表现得更好:
best.sigmoid
## sigmoid.test No Yes
## No 82 19
## Yes 11 35
(82+35)/147
## [1] 0.7959184
我们终于找到了一个在测试集上和训练集上有相同表现的模型了。看起来,可以选择sigmoid核函数作为最优预测。下面要对这些模型(包括线性模型)的性能进行评价,不只看正确率,而要使用各种指标。
5. 模型选择
我们已经研究了两种不同类型的建模技术,从各方面来看,KNN都处于下风。KNN在测试集上最好的正确率只有71%左右,相反,通过SVM可以获得接近80%的正确率。在简单选择具有最优正确率的模型(本案例中是使用sigmoid核函数的SVM模型)之前,先看看如何通过对混淆矩阵的深入研究来比较各种模型。
为了完成这个任务,还是要求助于强大的caret包,使用其中的confusionMatrix()函数。请注意,在前面的内容中,我们使用过InformationValue包中的同名函数。但caret包中的这个函数会生成我们评价和选择最优模型所需的所有统计量。先从建立的最后一个模型开始,使用的语法和基础的table()函数一样,不同之处是要指定positive类,如下所示: