BioArt按:表观遗传学信息在早期胚胎发育中是否被遗传,以及如果被遗传的话,其遗传规律和机制为何,一直都是生物学里的核心问题。随着新的高通组学技术发展,近年来对这一问题从不同层面有了较深入的研究。高等生物的DNA复制、基因转录都是在细胞核的三维空间中完成,细胞核的三维空间和基因组结构对相关的生物功能影响很大。其中一个重要的生物学问题是配子、合子和早期胚胎发育过程中,基因组的三维结构是什么样的、以及与发育的关系如何。7月12-13日,清华大学生命科学学院颉伟研究组和中科院北京基因组研究所刘江研究组(上海科技大学黄行许教授为共同通讯作者)分别在Nature杂志和Cell杂志“背靠背”发表研究论文,系统报道了哺乳动物染色体三维结构在着床前胚胎发育过程中的动态重编程过程。两篇文章均很好地描述了配子、合子和早期胚胎细胞的三维基因组结构特点,为研究者认识早期胚胎中真实的立体的基因组结构做了良好的铺垫,也为研究者解释早期胚胎发育中精细调控提供了重要的调控模式。鉴于这两项工作的重要意义,BioArt特别邀请到了北京大学生命科学学院李程研究员、中科院北京基因组研究所张治华研究员和华中农业大学李国亮教授对上述工作进行了专业点评,以飨读者!


论文解读:
在哺乳动物中,受精后的合子其染色质空间结构会经历一轮剧烈的重编程过程【1】。表观遗传学信息在早期胚胎发育中是否被遗传,以及如果被遗传的话,其遗传规律和机制为何,一直都是生物学里的核心问题。过去几年,随着一系列方法学的突破,人们逐渐认识到了高等生物早期胚胎发育过程中染色质可接近性(Chromatin accessiblility )、DNA甲基化和组蛋白修饰等系列生物学事件的动态变化过程。在这方面,来自中科院基因组研究所的刘江课题组【2,3】、清华大学颉伟课题组【4,5,6】、同济大学高绍荣课题组【7】、哈佛大学张毅课题组【8】以及UCSD任兵课题组【9】等都作出了重要的贡献。
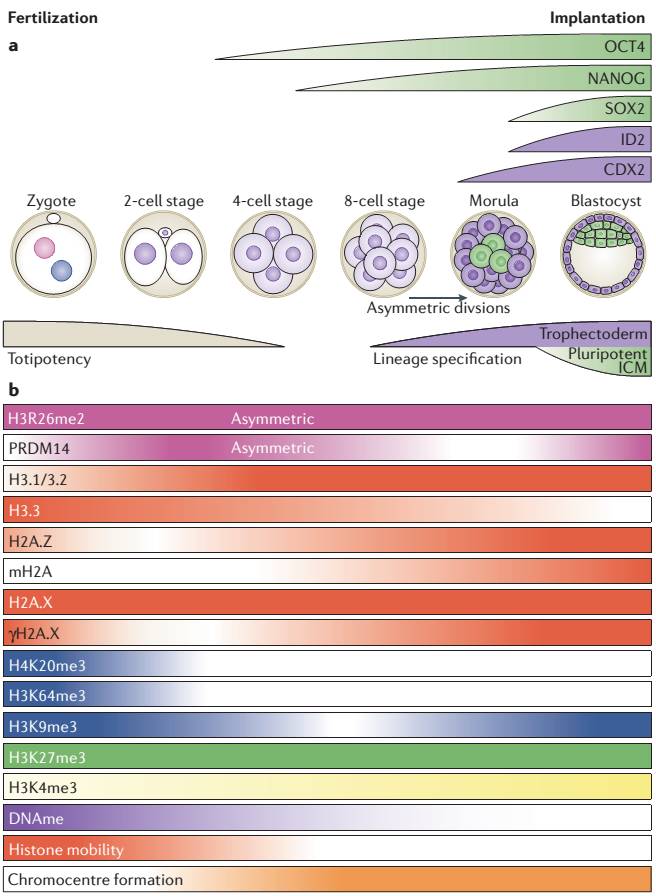
小鼠早期胚胎发育过程中染色质以及转录因子的变化过程示意图。引自:Burton, A., & Torres-Padilla, M. E. (2014). Chromatin dynamics in the regulation of cell fate allocation during early embryogenesis. Nature Reviews Molecular Cell Biology, 15(11), 723-735.
在真核生物中,线性DNA通过多层级地折叠以特定的三维结构存在于细胞核中。染色质的三维结构对于基因表达调控、DNA复制和重组等过程都具有至关重要的作用。例如基因组上的增强子(enhancer)可以通过形成染色质环结构与线性距离可能很远的启动子(promoter)相互作用以促进基因表达。近些年来,借助Hi-C(whole genome chromosome conformation capture)等染色质三维结构研究技术,科研人员获得了不同物种多种类型细胞的全基因组染色体三维结构信息【10】。
在研究过程中,人们发现了拓扑结构域(TADs, topologically associated domain)作为染色体三维结构的基本单位普遍存在于多个物种,而且这种结构在不同细胞类型甚至不同物种中非常保守。此外,在细胞分化过程中TADs也是相对稳定存在的,只是TADs之间相互作用的频率可能有所变化【11,12】。值得一提的是,来自UCSD的任兵教授在这个领域做出了系列重要贡献。
此外,以TADs为基本单位的染色质三维构象可被分为两类大的区室结构(Compartment A/B)。其中Compartment A与染色质开放区域、高基因密度区域以及转录活跃区域有很好的相关性。而Compartment B则经常出现在异染色质、基因荒漠(gene desert)和低转录区域【10】。
然而,由于细胞数量和实验手段的限制,染色体三维结构在哺乳动物早期胚胎发育过程中的动态变化却鲜为人知。来自清华大学颉伟研究组和中科院刘江研究组通过优化Hi-C技术【10,13】,开发出了一套适用于极少量细胞的Hi-C技术(sisHi-C, small scale in situ Hi-C),并成功将其应用于小鼠早期胚胎发育过程中染色体三维结构的研究中,揭示了哺乳动物受精前后染色体三维结构的亲本特异重编程过程。
在清华大学方面的工作中,研究者发现,精子保留了经典的染色体高级结构包括拓扑结构域(TADs)和区室结构。相反,卵子(MII oocyte)染色体呈现出一种均一性结构,并且缺乏拓扑结构域(TADs)和区室结构(compartment)。令人惊奇的是,在受精后,染色体的三维结构迅速呈现为一种极其松散的状态。
研究人员同时发现,不同于普通细胞,在早期胚胎发育过程中染色体三维结构的建立非常缓慢。这一过程表现为整个着床前发育过程拓扑结构域(TADs)的逐渐巩固以及染色体区室(Compartment A/B)的逐渐隔离。此外,研究人员通过分别对来源于父本和母本的两套基因组进行研究发现,在胚胎发育早期尽管原核已经融合,两套亲本基因组在空间上仍然是保持部分分离状态。另外,在受精卵中二者的染色体区室结构(Compartment)显著不同。这种亲本基因组在空间上的分离和染色体区室结构(Compartment)的差异直到8细胞阶段仍然存在。最后,研究者发现在抑制合子基因组激活(ZGA,zygotic genome activation)的情况下,染色体三维结构仍然能够部分建立,这表明这个时期的基因转录对建立三维结构并不是绝对必要的。
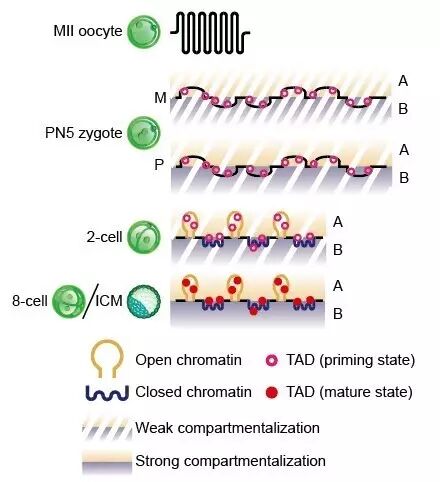
图1. 染色体三维结构在小鼠早期胚胎发育过程中的重编程模型。引自颉伟组Nature论文
而来自中科院方面的工作和清华大学方面的工作具有较高的相似性,并且实验结论也高度吻合,这样相互印证的结果更进一步表明了相关的科学性和可重复性。刘江研究组的实验结果同样表明了成熟卵子(MII oocyte)缺乏拓扑结构域(TADs),而且二细胞器TADs的形成依赖于DNA的复制,但是这个时期合子基因组的激活是非必需的。此外,研究人员还发现在精子基因组中存在高频的着超远距离(>4 Mb)的相互作用。值得注意的是,该项研究更详细地描述了精子中的三维基因组结构特点以及三维基因组结构对发育基因表达调控的影响,并发现CpG位点DNA甲基化状态与三维基因组结构建立的相关性。研究表明,相对于B染色体区室结构(B compartment)来讲,A染色体区室结构(A compartment)富集有更多未甲基化的CpGs,这也表明了A compartment具有更加松散开放的结构状态。
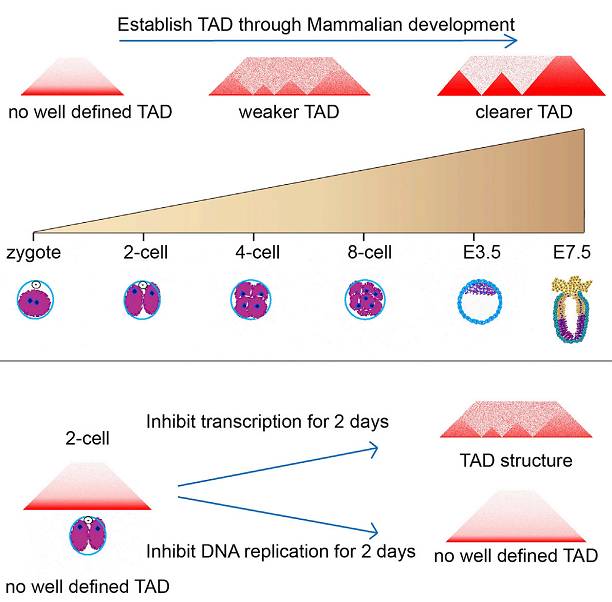
早期胚胎发育过程中TADs建立的动态变化模式图。引自刘江研究组的Cell论文
综上所述,上述工作发现染色体的三维结构在受精后首先呈现出一种极其松散的状态,并在随后的胚胎早期发育过程中逐步地以亲本特异的方式建立和成熟。两篇文章均很好地描述了配子、合子和早期胚胎细胞的三维基因组结构特点,为研究者认识早期胚胎中真实的立体的基因组结构做了良好的铺垫,也为研究者解释早期胚胎发育中精细调控提供了重要的调控模式。
据悉,清华方面,清华大学生命科学学院颉伟研究员为本文通讯作者。清华大学生命学院CLS项目博士生杜振海为本文第一作者,清华大学生命学院CLS项目毕业生郑辉博士、北京大学前沿交叉学科研究院CLS项目毕业生黄波博士和清华大学交叉信息学院硕士生马瑞在该工作中做出了重要贡献。清华大学生命学院PTN项目毕业生吴婧怡博士和清华大学自动化系博士生张祥林也参与了该课题。合作实验室包括清华大学交叉信息学院曾坚阳组、清华大学自动化系汪小我组、张奇伟(Michael Q. Zhang)组、高军涛、以及美国索尔克生物研究所(Salk Institute for Biological Studies)Jesse Dixon组。课题得到了清华大学实验动物中心和生物医学测试中心基因测序平台的大力协助和支持。该研究获得了国家重点专项研发计划、国家重点基础研究发展计划(973计划)、国家自然科学基金、中组部青年千人计划基金、生命科学联合中心、美国国立卫生研究院、清华大学信息科学与技术国家实验室学科交叉基金、北京市清华大学结构生物学高精尖创新中心的经费支持。
此外,刘江研究员和上海科技大学黄行许教授为Cell论文的共同通讯作者,清华方面的颉伟研究员和博士生杜振海在该文中亦有署名。
专家点评:
李程(北京大学生命科学学院、清华-北大生命科学联合中心、统计科学中心高级研究员)

Comments:三维基因组测序技术如Hi-C、ChIA-PET近些年在生物医学问题中应用越来越广泛,检测到癌症、干细胞、免疫、发育等生物学过程中细胞染色质空间组织结构的变化,以及这些变化和基因组变异、表观遗传修饰、基因表达的关联和相互影响。但是胚胎发育早期的三维基因组数据和研究还处于空白,主要限制因素是Hi-C等技术需要大量的细胞数目(10^6)。在这两个最新工作中,颉伟组和刘江组首先开发了基于少量细胞(500)的Hi-C技术,进而应用于检测小鼠受精卵前后时期的细胞三维基因组,从拓扑结构域(TAD)、A/B间隔、远端和近端染色质相互作用比例的变化层面,首次证明了染色质三维结构在2细胞时期开始逐渐建立,到ICM时期基本建成和胚胎干细胞相似的三维结构。有趣的是,作者发现2细胞时期的染色体复制对建立染色质三维结构是必要的,但这个时期的基因转录对建立三维结构并不是绝对必要。作者推测在早期胚胎的分裂过程中,每一次从分裂中期高度凝聚的染色体转换为G1期的染色质结构,都有更强的“驱动力”来形成TAD、A/B等结构。染色质三维结构如何在细胞分裂后维持,目前是一个没有完全理解的问题,候选模型包括染色质纤维自组织、与转录过程关联、表观遗传修饰和染色质结合因子伴随DNA复制传递。这两篇文章从早期胚胎发育的角度提出了新思路和证据。关于早期胚胎发育过程已经积累了表达谱、表观遗传、染色质可接近性等多组学数据,刘江组的文章将这些数据和Hi-C数据做了初步关联。相信作者公布的新数据将为表观遗传、生物信息研究领域提供宝贵的资源,以进一步整合挖掘这些数据,帮助理解胚胎发育和三维基因组的奥秘。
张治华(中科院北京基因组研究所研究员)

Comments:表观遗传学信息在早期胚胎发育中是否被遗传,以及如果被遗传的话,其遗传规律和机制为何,一直都是生物学里的核心问题。随着新的高通组学技术发展,近年对这一问题有了较深入的研究。中科院北京基因组所刘江研究员之前深入的研究了斑马鱼和哺乳动物早期胚胎发育中的DNA甲基化图谱的代际传递规律。清华大学颉伟教授则深入的研究了哺乳动物早期胚胎发育中染色质开放性,组蛋白修饰(与UCSD的任兵团队、同济大学的高邵荣/张勇团队同时报道)的代际传递规律(详见BioArt此前的报道:清华颉伟组和同济高绍荣组在《自然》发表“背靠背”论文,揭示早期胚胎发育的表观遗传图景)。作为领域内的又一个关键进展,这次刘江、颉伟同时报道的则是表观遗传学中的更高级结构,即基因组三维空间结构的代际传递规律。因为几乎所有核内生命活动都或多或少依赖于其发生的基因组三维结构,所以它的遗传规律是一个非常基础性的问题。刘江和颉伟同时报道了一个惊讶结果,即原本存在于精子中的一种精细结构(拓扑结构域)在其形成合子之后几乎马上就消失了,而合子要至少经历了一次到两次细胞分裂才会自发的重建了这一结构。这一结果如果和前段时间欧美以色列组发表的单细胞测序结果对照会更有意思。除了新的数据和现象之外,个人认为最重要的是,从两个课题组的结果中,我们可以提出了更多有意思的问题。比如基因组三维结构重建的信息是从哪里来的?是来自DNA序列本身吗?还是有未知的所谓书签因子?亦或是来自胞质?个人最感兴趣的是,这些新现象的发现为进一步研究核内基因组的空间折叠动力学过程,提供了新的线索。
李国亮(华中农业大学生命科学技术学院教授、国家“青年千人”)

Comments:高等生物的DNA复制、基因转录都是在细胞核的三维空间中完成,细胞核的三维空间和基因组结构对相关的生物功能影响很大。其中一个重要的生物学问题是配子、合子和早期胚胎发育过程中,基因组的三维结构是什么样的、以及与发育的关系如何。这周清华大学颉伟课题组、北京基因组所刘江课题组和上海科技大学黄行许课题组都以小鼠配子、早期胚胎为实验对象,分别在Nature和Cell发表了高水平的研究论文,对基因组三维结构进行了深入研究。在早期胚胎发育过程,细胞数目过少是相关研究的极大挑战。颉伟课题组致力于早期胚胎发育相关的表观遗传组技术开发和研究。在最新的Nature文章中, 颉伟课题组秉承了减少纯化步骤等少量细胞实验技术的原则,以成熟生殖细胞、胚胎早期发育细胞为实验材料,通过改良的in situ Hi-C(原位高通量染色质构象捕获)技术,解析了哺乳动物发育早期基因组三维结构建立的过程和特点,研究发现MII期的卵子的拓扑结构域(TAD)会丢失,而受精卵在分裂和发育过程中,至少到8细胞时期为止,由慢变快地恢复建立基因组三维结构,且父源和母源染色体相对独立。而北京基因组所刘江课题组和上海科技大学黄行许课题组依托高通量测序的优势平台,以传统的in situ Hi-C方法进行了类似的研究,得到了基本相同的结论,但同时还更详细地描述了精子中的三维基因组结构特点以及三维基因组结构对发育基因表达调控的影响,并发现CpG位点DNA甲基化状态与三维基因组结构建立的相关性,研究还证明了三维基因组结构的建立与DNA复制相关,但与合子基因组激活并不相关。
两篇文章均很好地描述了配子、合子和早期胚胎细胞的三维基因组结构特点,为研究者认识早期胚胎中真实的立体的基因组结构做了良好的铺垫,也为研究者解释早期胚胎发育中精细调控提供了重要的调控模式。这两篇论文提出了三维基因组建立的驱动因素、调控发育的具体功能等重大科学问题,留待研究者继续研究。
参考文献:
1、Burton, A., & Torres-Padilla, M. E. (2014). Chromatin dynamics in the regulation of cell fate allocation during early embryogenesis. Nature Reviews Molecular Cell Biology, 15(11), 723-735.
2、Jiang, L., Zhang, J., Wang, J. J., Wang, L., Zhang, L., Li, G., ... & Liu, J. (2013). Sperm, but not oocyte, DNA methylome is inherited by zebrafish early embryos. Cell, 153(4), 773-784.
3、Wang, L., Zhang, J., Duan, J., Gao, X., Zhu, W., Lu, X., ... & Liu, J. (2014). Programming and inheritance of parental DNA methylomes in mammals. Cell, 157(4), 979-991.
4、Wu, J., Huang,B., Chen, H., Yin, Q., Liu, Y., Xiang, Y., ... & Xie, W. (2016). The landscape of accessible chromatin in mammalianpreimplantation embryos. Nature, 534(7609), 652-657.
5、Zhang, B., Zheng, H., Huang, B., Li, W., Xiang, Y., Peng, X., ... & Xie, W. (2016). Allelic reprogramming of the histone modification H3K4me3 in early mammalian development. Nature, 537(7621), 553-557.
6、Zheng, H., Huang, B., Zhang, B., Xiang, Y., Du, Z., Xu, Q., ... &Xie, W. (2016). Resetting epigenetic memory by reprogramming of histone modifications in mammals. Molecular cell, 63(6), 1066-1079.
7、Liu, X., Wang, C., Liu, W., Li, J., Li, C., Kou, X., ... & Gao, S. (2016). Distinct features of H3K4me3 and H3K27me3 chromatin domains in pre-implantation embryos. Nature, 537(7621), 558-562.
8、Lu, F., Liu, Y., Inoue, A., Suzuki, T., Zhao, K., & Zhang, Y. (2016). Establishing chromatin regulatory landscape during mouse preimplantation development. Cell, 165(6), 1375-1388.
9、Dahl, J. A., Jung, I., Aanes, H., Greggains, G. D., Manaf, A., Lerdrup, M., ... & Preissl, S. (2016). Broad histone H3K4me3 domains in mouse oocytes modulate maternal-to-zygotic transition. Nature, 537(7621), 548-552.
10、Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., ... & Sandstrom, R. (2009). Comprehensive mapping of long-range interactions reveals folding principles of the human genome. science, 326(5950), 289-293.
11、Dixon, J. R., Selvaraj, S., Yue, F., Kim, A., Li, Y., Shen, Y., ... & Ren, B. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature, 485(7398), 376-380.
12、Sexton, T., Yaffe, E., Kenigsberg, E., Bantignies, F., Leblanc, B., Hoichman, M., ... & Cavalli, G. (2012). Three-dimensional folding and functional organization principles of the Drosophila genome. Cell, 148(3), 458-472.
13、Rao, S. S., Huntley, M. H., Durand, N. C., Stamenova, E. K., Bochkov, I. D., Robinson, J. T., ... & Aiden, E. L. (2014). A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell, 159(7), 1665-1680.

颉伟,2003年在北京大学生命科学学院获学士学位,2008年在美国加州大学洛杉矶分校获分子生物学博士学位、统计学硕士双学位。2009至2013年,在美国加州大学圣地亚哥分校从事博士后研究。2013年,受聘于清华大学生命科学学院,同时入选清华-北大生命科学联合中心。颉伟研究早期胚胎发育中染色质的重编程及表观遗传信息的传递、建立和调控,希望阐明在生命起始时期,精子和卵子是如何通过受精并进一步发育成为全能性胚胎、及在此过程中父母与子代的信息传递关系,这是个体发育和细胞分化生物学里最基本、最有挑战性的科学问题之一。2016年6月,颉伟实验室在Nature杂志以长文形式首次报道了哺乳动物早期胚胎染色质在分子水平的重编程模式。另外,颉伟和医学院那洁课题组合作,通过开发高灵敏检测方法在国际上首次报道了组蛋白修饰在哺乳动物早期胚胎发育中的重编程规律,第一次回答了“组蛋白修饰能否传递到下一代”这个长久以来悬而未决的重大科学问题。这一成果2016年9月发表于Nature和Molecular Cell杂志。2016年12月,颉伟实验室再次在Molecular Cell期刊发表论文,报道基因印记(一种特殊的亲代表观遗传记忆)是如何通过一种调节因子的蛋白亚型转化所调控的。这些研究成果首次在DNA水平揭示了受精后染色质剧烈的重编程过程,开创和建立了研究早期胚胎发育中染色质调控的一系列重要体系和方法,为更好地研究和理解生命如何起始铺平了道路。迄今,颉伟已发表高水平论文30篇,包括以第一或通讯作者身份在Nature、Science和Cell等杂志上发表的多篇论文。因其杰出的学术成就,颉伟入选中组部“青年千人”(2013),获得国家优秀青年基金(2014),并获评香港“求是杰出青年学者”(2014)。特别值得一提的是,今年5月,颉伟研究员还入选了HHMI的“国际学者项目”。

刘江,博士,现任中科院北京基因组研究所研究员,国家“杰青”,中青年科技创新领军人才。刘江研究员1998年本科毕业于烟台大学,2003年在中科院生物物理研究所获得博士学位,此后先后在美国密西根大学和耶鲁大学做博后,2006年-2009年在芝加哥大学担任研究助理,2009年7月回国到中科院北京基因组研究所担任“百人计划”研究员。近五年来,刘江研究员课题组在早期胚胎发育过程中DNA甲基化谱式的传递方面做出了一些列杰出的工作,特别是2013年在Cell杂志上以封面论文形式证明了在斑马鱼中除了DNA可以从父母传递到子代外,精子的DNA甲基化图谱也可以被遗传到子代中,并在胚胎发育中发挥重要作用;2014年该课题组与黄行许课题组合作继续在Cell杂志上揭示了哺乳动物如何继承亲代甲基化图谱的规律,突破了早期胚胎发育过程中DNA 甲基化重编程的传统认识。

BioArt,一心关注生命科学,只为分享更多有种、有趣、有料的信息。关注请长按上方二维码。投稿、合作、转载授权事宜请联系微信ID:fullbellies或邮箱:[email protected]。















