
今天小张就先介绍三篇最近很火的发文章的套路:
不做实验,只挖掘数据库就发表文章
。
第一篇是今年 5 月份刚发的文章,杂志是 Pathol Oncol Res,影响因子 1.7分,研究团队是伊朗的科学家。

文章主要通过 cBioportal 对 TCGA 数据中肝癌的数据进行挖掘,结果找到了一条 lncRNA SNHG6 作为肝癌的分子标志物。结果如下:
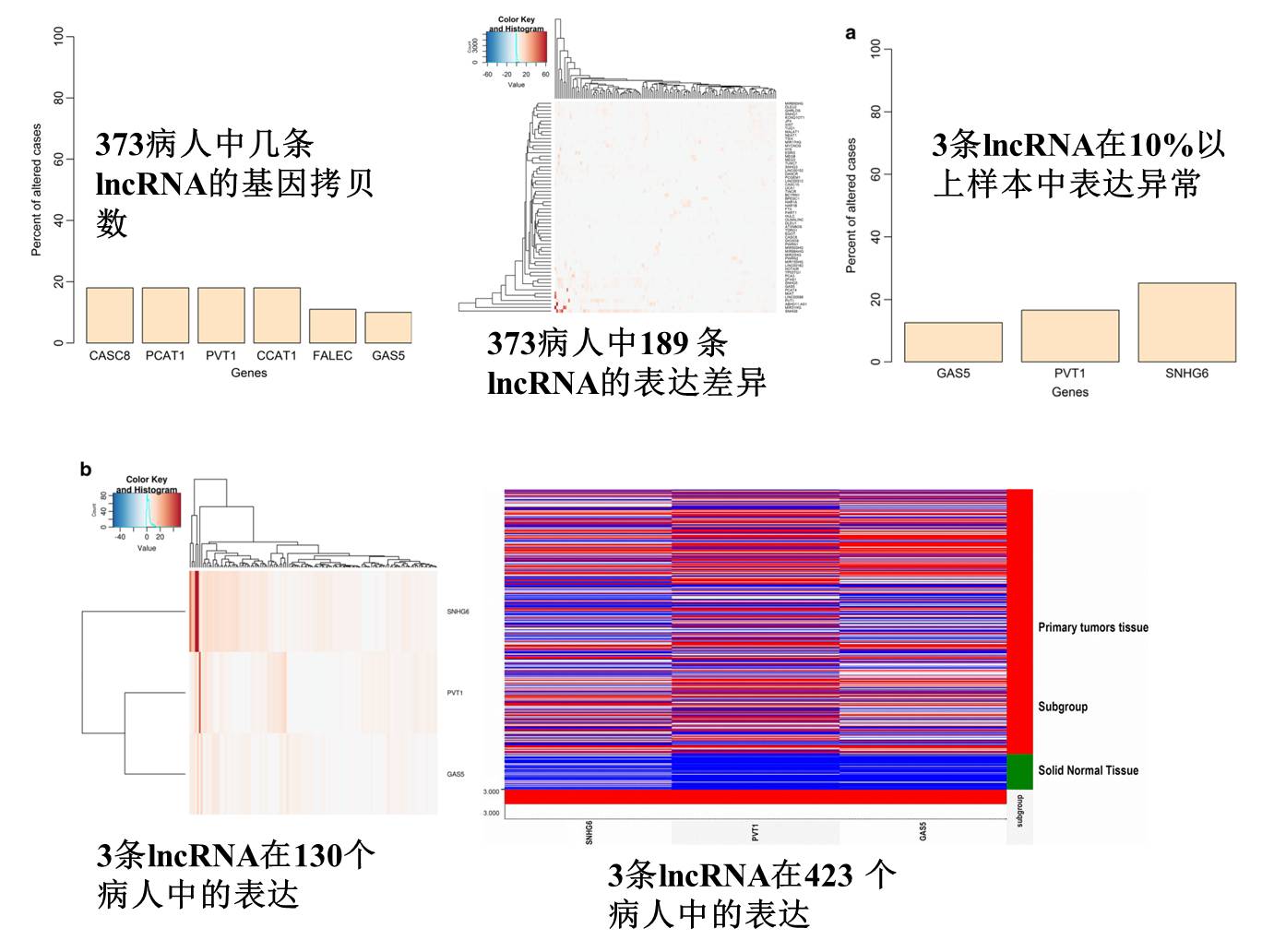
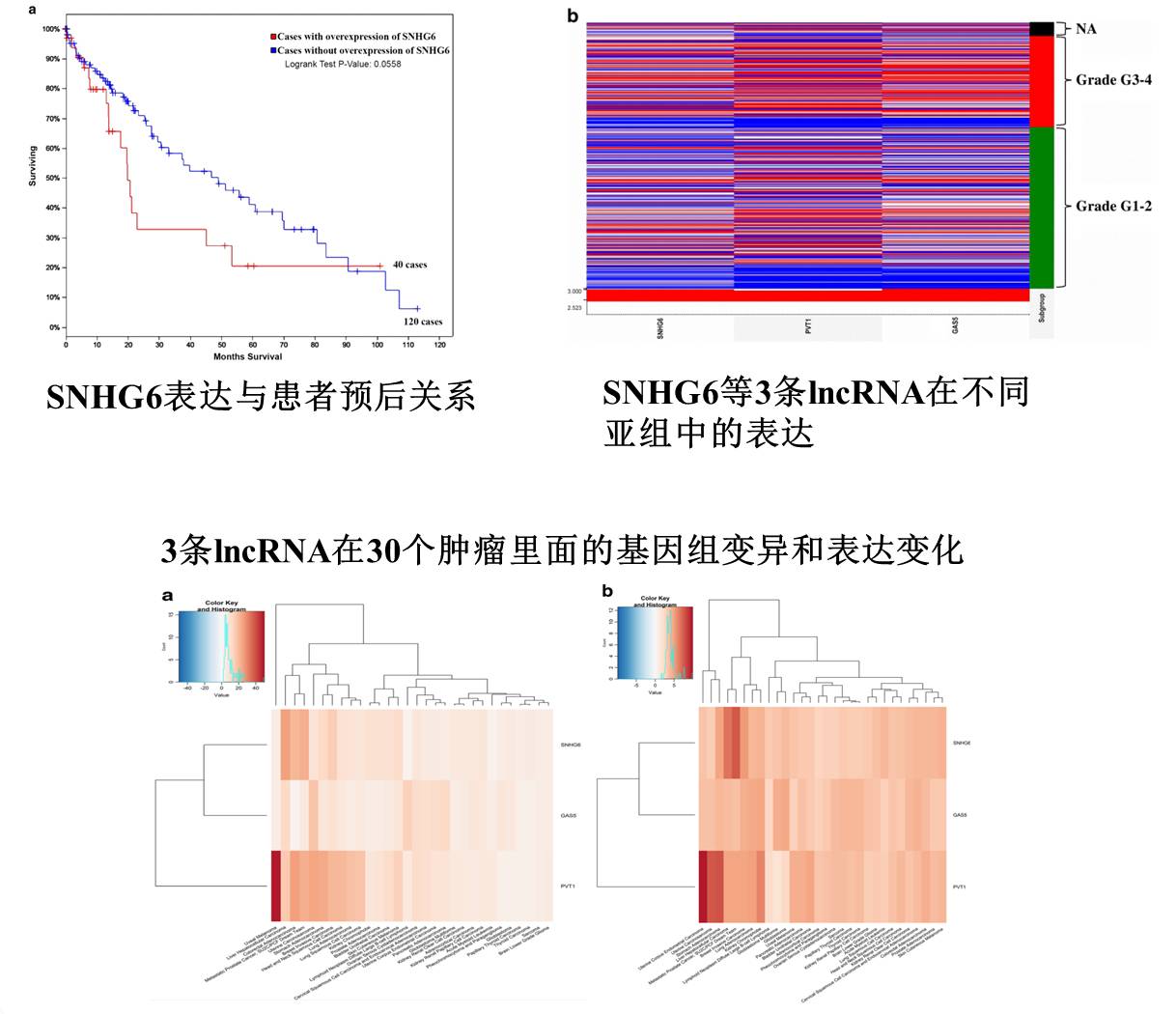
感觉做了好多东西,其实就说了两件事:
-
lncRNA SNHG6等3条lncRNA在肝癌患者中基因组水平的改变和表达情况;
-
lncRNA SNHG6与患者预后相关;
关键是只用了一个 TCG
A 使用工具:
cBioportal
,而这个工具使用起来也非常简单,就这么挖掘下数据库一篇 1.7 分的文章就发出来了。
这篇文章是国内团队 3 月底发的,杂志 International Journal of Molecular Sciences,影响因子 3.2,分数已经突破 3 分了,文章说的是
通过生物信息学分析鉴定结直肠癌关键候选基因和信号通路。

下面我们看研究内容:
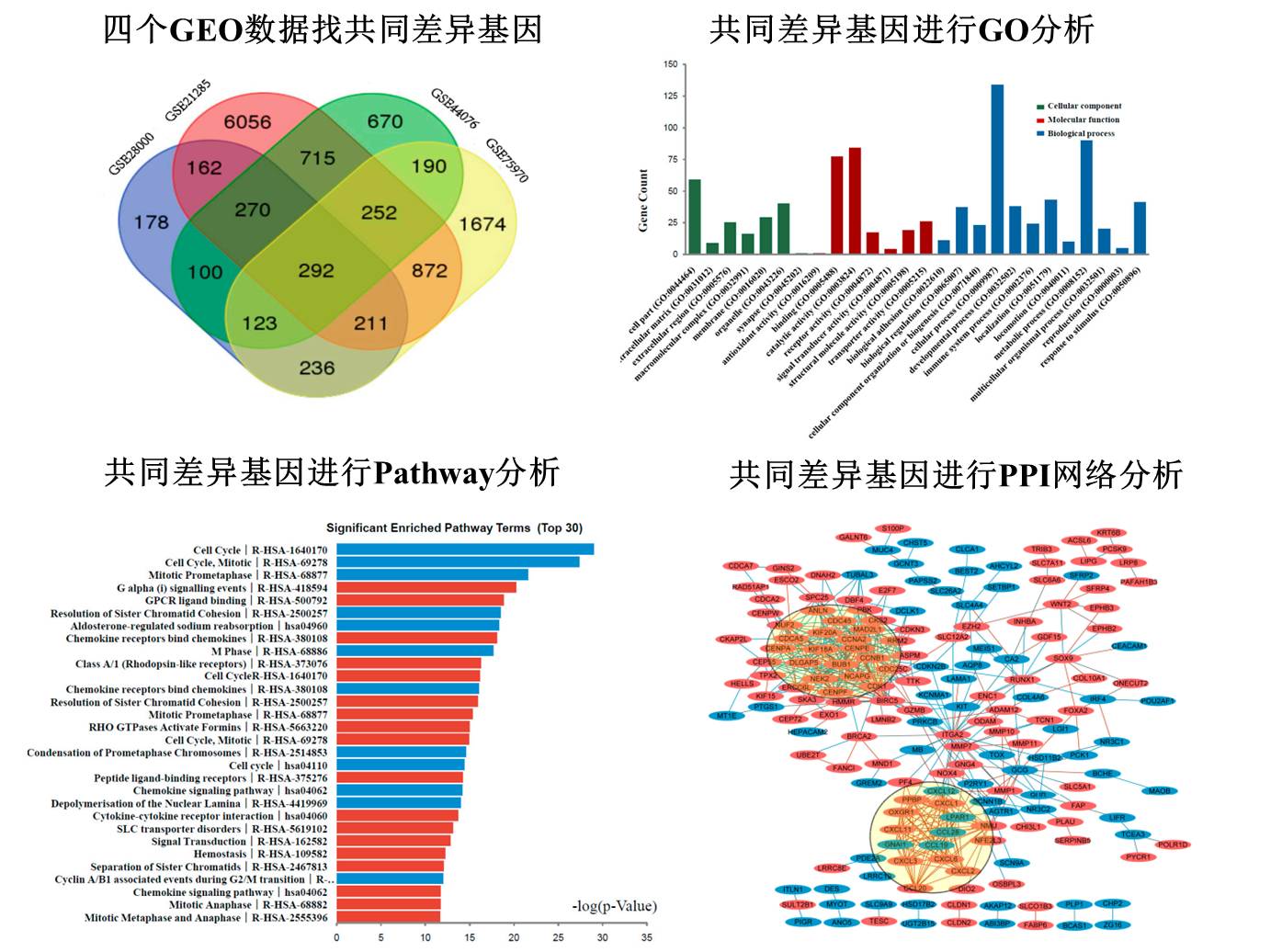
第一印象:图好漂亮,不过分析似乎有点太简单了吧。

今年 5 月 22 号发表,杂志是 Oncotarget,虽然 OT 被大家广为诟病,不过新影响因子出来还是保持住了 5 分大关,还略微升了那么一丢丢。文章说的是通过 RNA 测序和芯片数据挖掘研究异常表达的 lncRNA 在肺鳞癌中的临床意义,我们看看这篇是不是让我们对 OT 另眼相看:
首先通过 R 语言分析 TCGA 数据库中的差异表达 lncRNA,并用火山图展示:
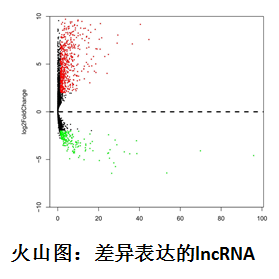
(
横过来的火山图还是火山图)
接下来,分别通过箱式图、ROC 曲线、KM 生存分析和与基因 FGFR1 的共表达分析展示 10 条最显著 lncRNA 的结果,四张图就是 4 个 fig:
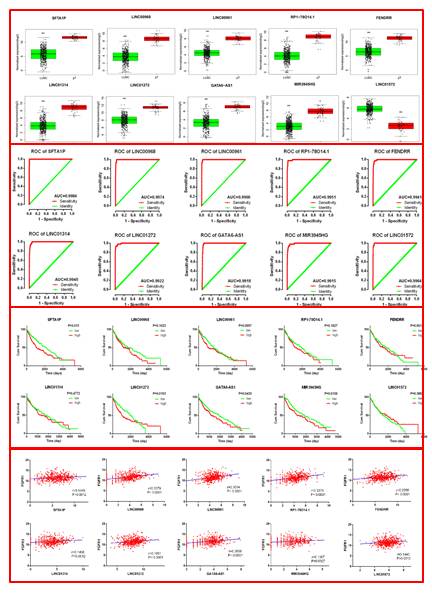
再往下是 lncRNA 表达在不同病理参数分组下的表达差异:





