CRISPR-Cas核酸酶是操纵核酸的有力工具; 然而,
靶向DNA的插入仍然是一个挑战,因为它需要宿主细胞修复机制。
2019年6月6日,美国博德研究所张锋在
Science
在线发表题为“
RNA-guided DNA insertion with CRISPR-associated transposases
”的研究论文,该研究
表征来自蓝细菌Scytonema hofmanni的CRISPR相关转座酶(CAST),其由Tn7样转座酶亚基和V-K CRISPR效应物(Cas12k)组成。 ShCAST通过在原型间隔区下游60-66bp处定向插入DNA片段,该过程是催化RNA指导的DNA转座。 ShCAST将DNA整合到大肠杆菌基因组中的独特位点,频率高达80%,无需正选择。
这项工作扩展了对CRISPR-Cas系统功能多样性的理解,并建立了精确DNA插入的范例。
总之,这项工作确定了CRISPR-Cas系统超出适应性免疫的功能,该功能不需要Cas核酸酶活性,并且
提供了靶向插入DNA的策略
,而
不涉及同源重组途径,在真核细胞中具有特别令人兴奋的基因组编辑潜力。
有趣的是,得益于这一消息,张锋等人的Editas Medicine公司股票股价迅速拉升。

CRISPR和CRISPR相关蛋白(Cas)系统通过指导RNA依赖性DNA或RNA核酸酶活性提供针对外源遗传元件的适应性免疫。
已经利用CRISPR效应物,例如Cas9和Cas12进行基因组编辑,并在基因组中产生靶向DNA双链断裂,然后通过内源DNA损伤修复途径修复。
Editas股票走势
虽然通过同源重组或非同源末端连接可以实现Cas9切割后新DNA的精确整合,但这些过程效率低并且根据细胞类型而变化很大。同源重组修复也与活性细胞分裂有关,使其不适合有丝分裂后细胞。最近,已经开发了一种在DNA上进行点突变的替代方法,其依赖于使用dCas9来募集胞苷或腺嘌呤脱氨酶以实现基因组DNA的碱基编辑。
然而,碱基编辑仅限于核苷酸取代,因此DNA有效且靶向地整合到基因组中仍然是主要挑战。
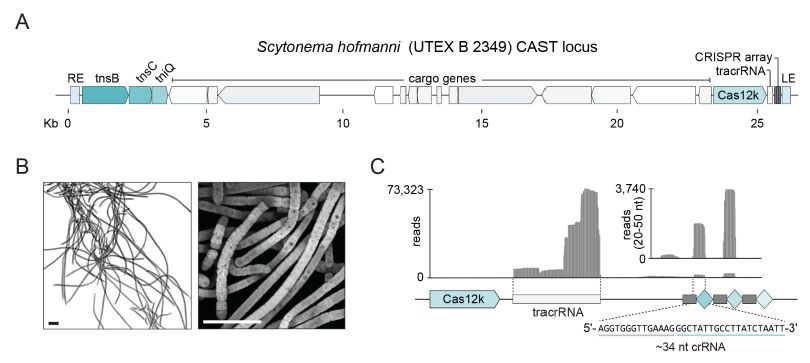
CRISPR相关转座酶(CAST)系统的靶向特点
先前已经鉴定了
许多缺乏活性核酸酶结构域的CRISPR-Cas系统,缺乏核酸酶结构域引发了关于这些CRISPR-Cas系统的生物学功能的问题,这些系统只能结合而不能切割DNA。
最近,报道了Tn7样转座子与亚型I-F,亚型I-B或亚型V-K(以前称为V-U5)CRISPR-Cas系统之间的关联。 CRISPR-Cas相关的Tn7样转座子含有tnsA,tnsB,tnsC和tniQ基因,类似于经典Tn7异源三聚体TnsABC复合物。
Tn7通过两种替代途径靶向DNA
,这两种途径分别由TnsD介导,TnsD是识别Tn7附着位点的序列特异性DNA结合蛋白;以及TnsE,其促进转座成结合质粒和复制DNA 。
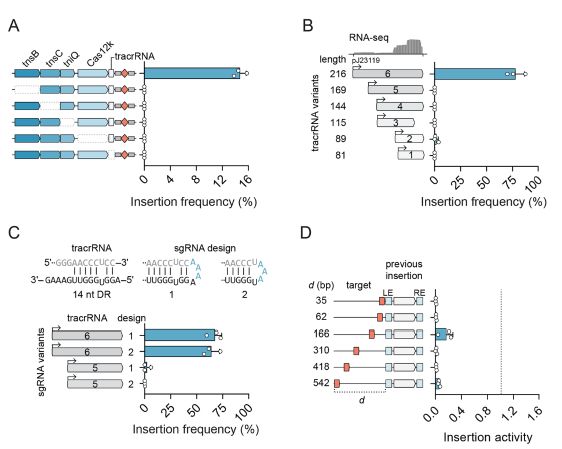
RNA引导插入的遗传要求
在转座子编码的CRISPR-Cas变体中,
亚型V-K是最简单的,因为它们含有单蛋白CRISPR-Cas效应子,Cas12k(以前称为C2c5),亚型V-K系统目前仅限于蓝细菌。
所有V-K系统都嵌入在预测的Tn7样转座因子中,没有额外的cas基因,这表明,
如果它们是活跃的CRISPR-Cas系统,它们可能依赖于反式提供的适应模块。
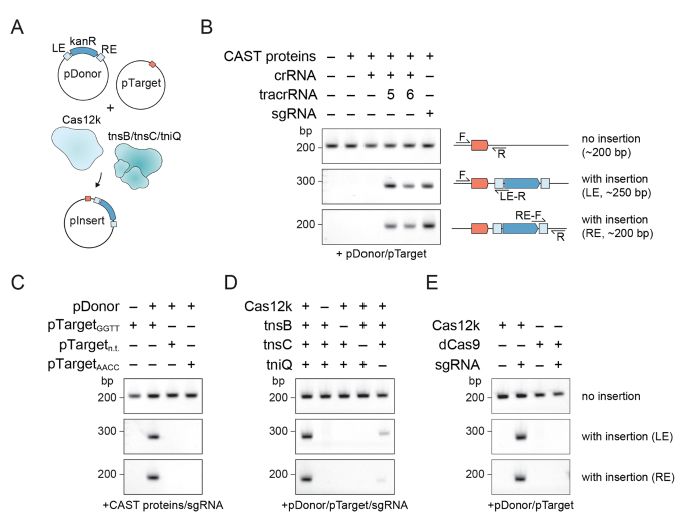
RNA引导的转座酶的体外重建
Tn7样转座子和CRISPR-Cas系统之间的关联表明,
转座子可能已经劫持了CRISPR效应子,在靶位点产生R-环,并通过质粒和噬菌体促进转座子的扩散。
在亚型V-K的情况下,CRISPR-Cas基因座的位置在预测的转座子中经常保守,表明CRISPR-Cas与转座连锁。然而,由于经典Tn7转座子通常携带具有对宿主细胞有益的防御功能的基因,因此CRISPR-Cas也可能是目标基因
。迄今为止,尚未报道转座子编码的CRISPR-Cas系统的功能数据。
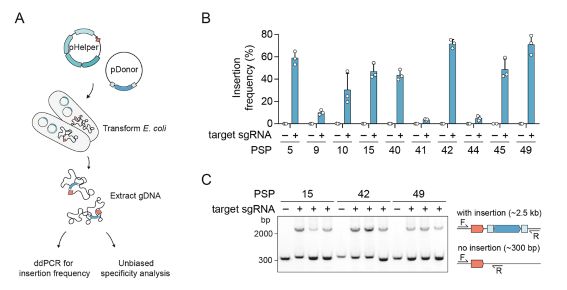
ShCAST介导大肠杆菌中的基因组插入
在这里,研究人员表征来自蓝细菌Scytonema hofmanni的CRISPR相关转座酶(CAST),其
由Tn7样转座酶亚基和V-K CRISPR效应物(Cas12k)组成
。 ShCAST通过在原型间隔区下游单向插入DNA片段60-66 bp来催化RNA指导的DNA转座。 ShCAST将DNA整合到大肠杆菌基因组中的独特位点,频率高达80%,无需正选择。
这项工作扩展了对CRISPR-Cas系统功能多样性的理解,并建立了精确DNA插入的范例。
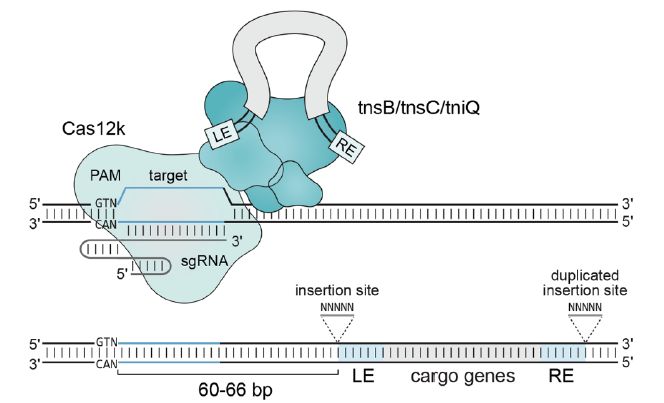
RNA指导的DNA转座模型
总之,这项工作确定了
CRISPR-Cas系统超出适应性免疫的功能,该功能不需要Cas核酸酶活性,并且提供了靶向插入DNA的策略,而不涉及同源重组途径,在真核细胞中具有特别令人兴奋的基因组编辑潜力。

—
END
—